Gráficos en R: Diagramas de barras (barplots)
Angelo Santana & Carmen Nieves Hernández,
Departamento de Matemáticas, ULPGC
Datos.
Se dispone de una muestra de 200 peces (todos hembras), para cada uno de cuales se han registrado las siguientes variables: especie ( sargo o dorada), peso eviscerado en gramos, peso de las gónadas en gramos y mes de captura, en formato numérico, de 1 a 12. Podemos leer el archivo de datos desde la dirección de internet en que se encuentra, simplemente mediante:
peces=read.csv2("https://estadistica-dma.ulpgc.es/cursoR4ULPGC/datos/datosGrafica.csv")
str(peces)
head(peces)## 'data.frame': 200 obs. of 4 variables:
## $ especie : Factor w/ 2 levels "Dorada","Sargo": 2 1 1 1 2 1 1 2 1 2 ...
## $ mes : int 7 7 7 9 10 11 9 4 12 6 ...
## $ pesoEvis: int 1061 1062 1207 1057 1303 1219 1494 1266 1178 1054 ...
## $ gonada : int 53 79 43 32 63 38 12 169 78 70 ...## especie mes pesoEvis gonada
## 1 Sargo 7 1061 53
## 2 Dorada 7 1062 79
## 3 Dorada 7 1207 43
## 4 Dorada 9 1057 32
## 5 Sargo 10 1303 63
## 6 Dorada 11 1219 38Nuestro objetivo en esta sección será construir el siguiente diagrama de barras con la variable especie. A esta gráfica la denominaremos “gráfica patrón” a lo largo del texto.
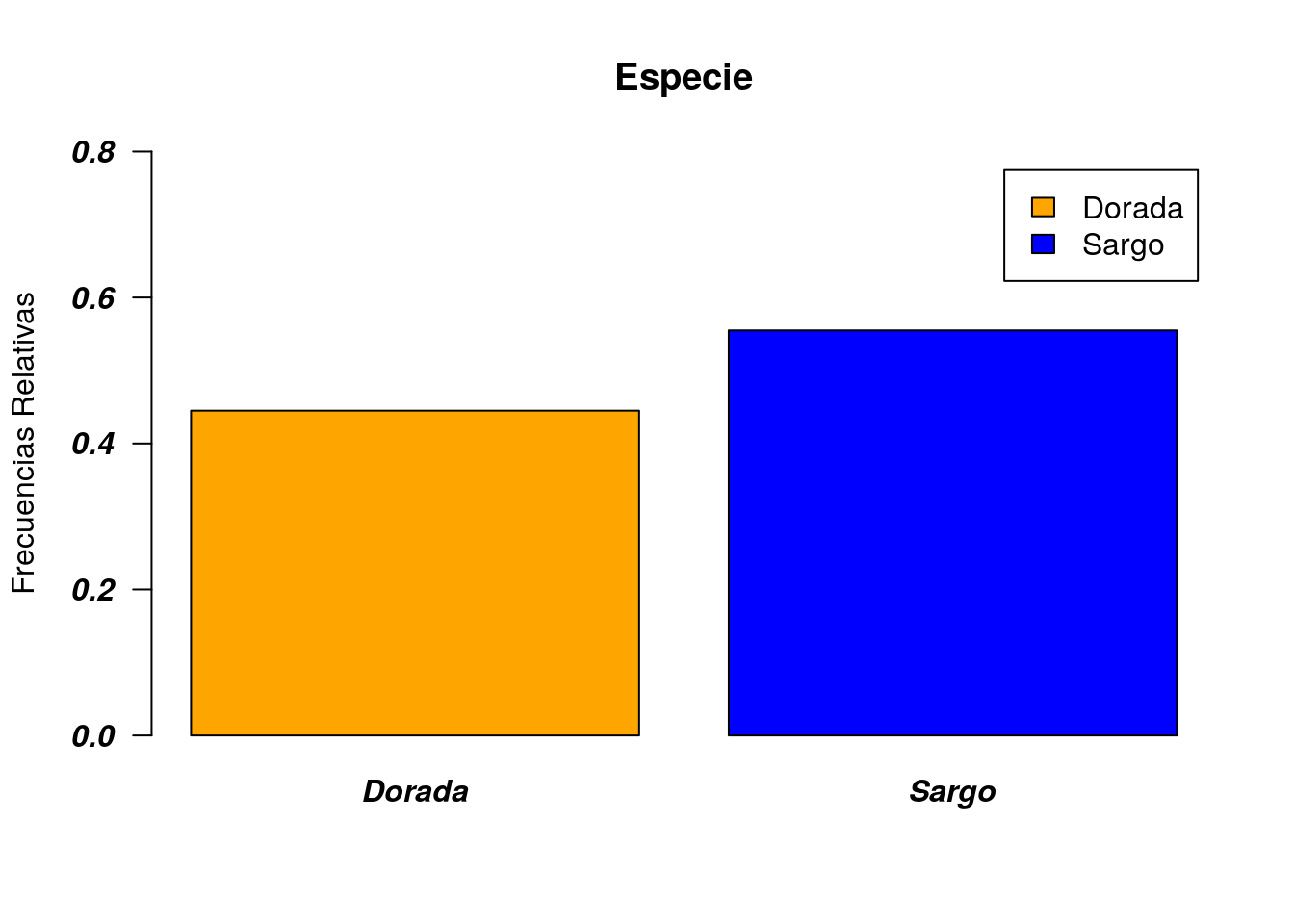
Primer Paso
En primer lugar ejecutamos help(barplot) para leer la información que proporciona la ayuda de R sobre la función barplot. Para empezar, debemos pasar como argumento a dicha función los datos de la variable que queremos representar. En concreto, debemos especificar las frecuencias relativas de las categorías presentes en la variable especie. El cálculo de las frecuencias relativas se realiza a través de las funciones table() y prop.table()
barplot(prop.table(table(peces$especie)))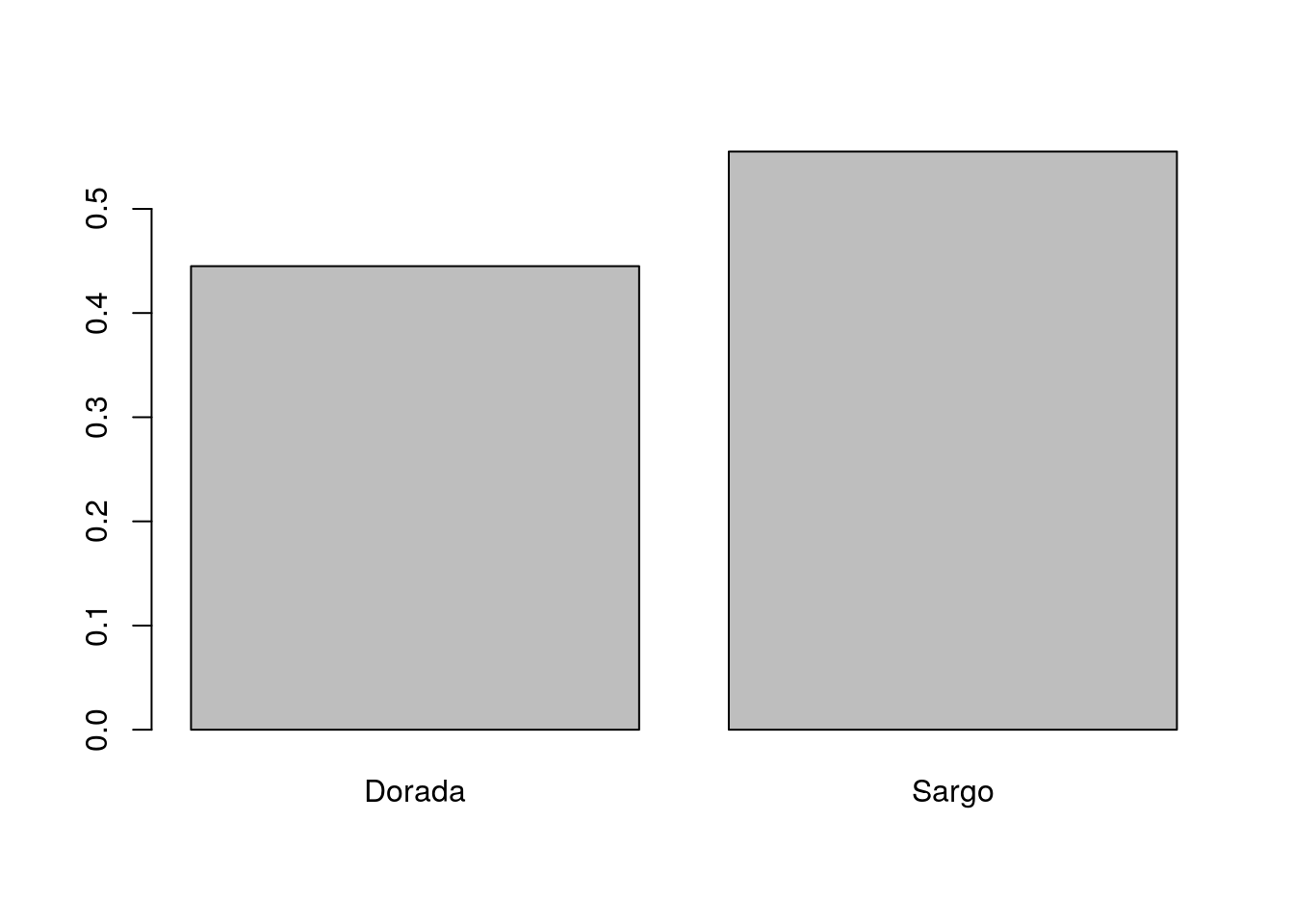
Color
En la gráfica anterior ya tenemos representado el mismo contenido que en la gráfica patrón. Vamos ahora a darle color. El argumento que controla los colores de las barras del barplot es col:
barplot(prop.table(table(peces$especie)),col=c("orange","blue"))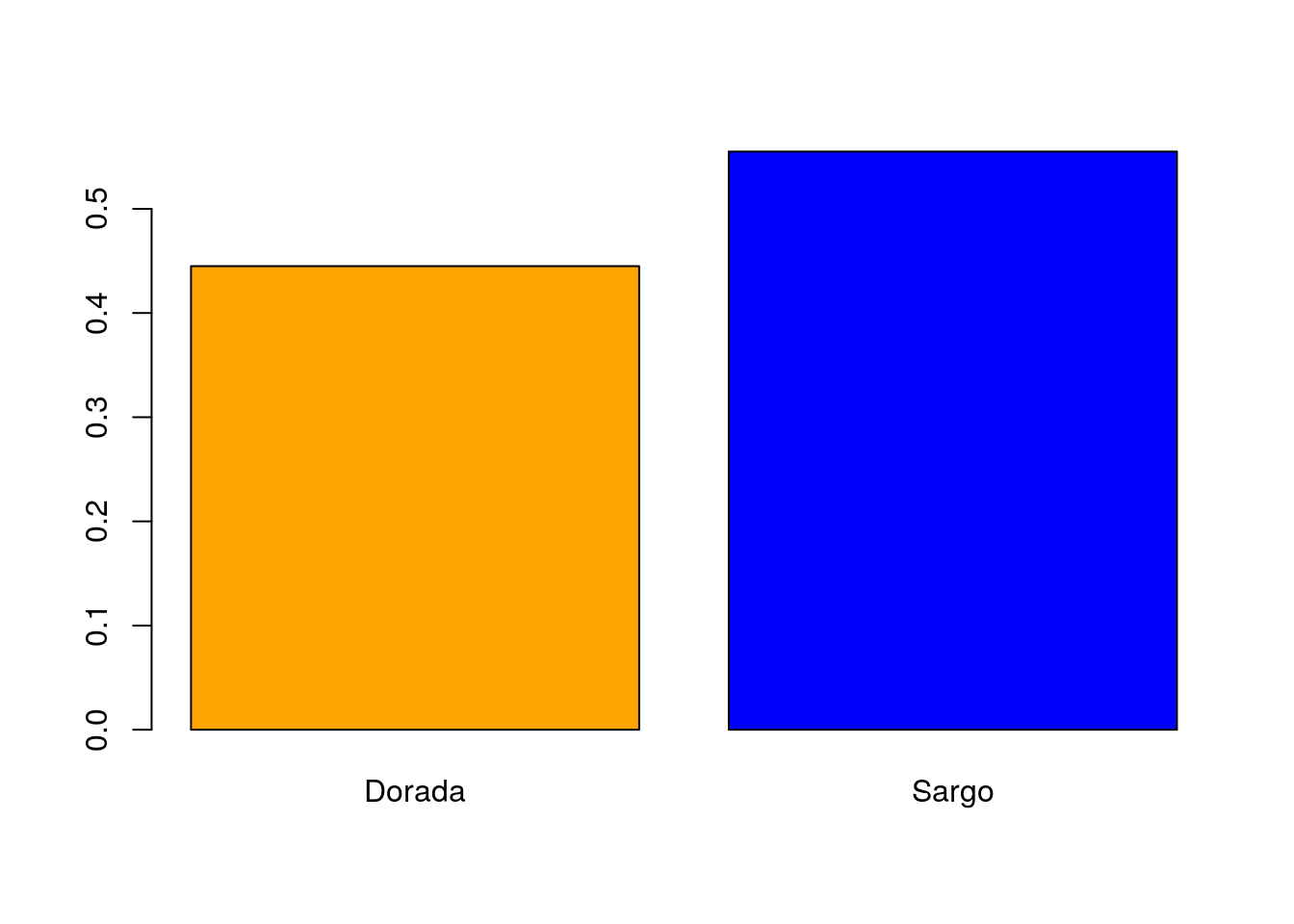
Leyenda
Ahora añadimos una leyenda utilizando el argumento legend.text:
barplot(prop.table(table(peces$especie)),col=c("orange","blue"),
legend.text=c("Dorada","Sargo"))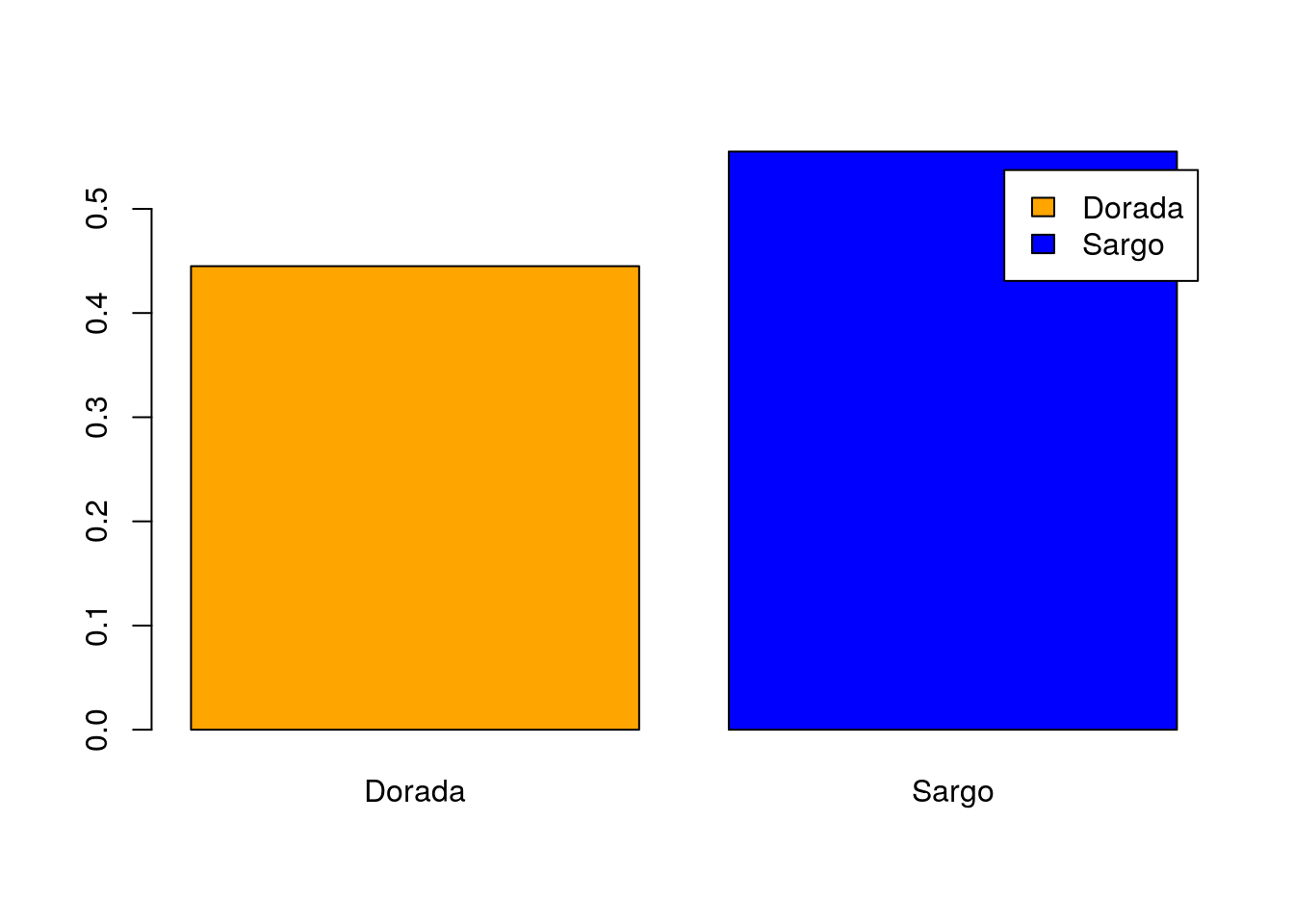
Ajustando los ejes para dejar sitio a la leyenda
La leyenda que que acabamos de colocar “pisa” una de las barras del gráfico. En la gráfica patrón podemos observar que el eje de ordenadas tiene una amplitud mayor, que va de 0 a 0.8 unidades. Para cambiar los límites del eje de ordenadas utilizamos el argumento ylim:
barplot(prop.table(table(peces$especie)),col=c("orange","blue"),
legend.text=c("Dorada","Sargo"),ylim=c(0,0.8))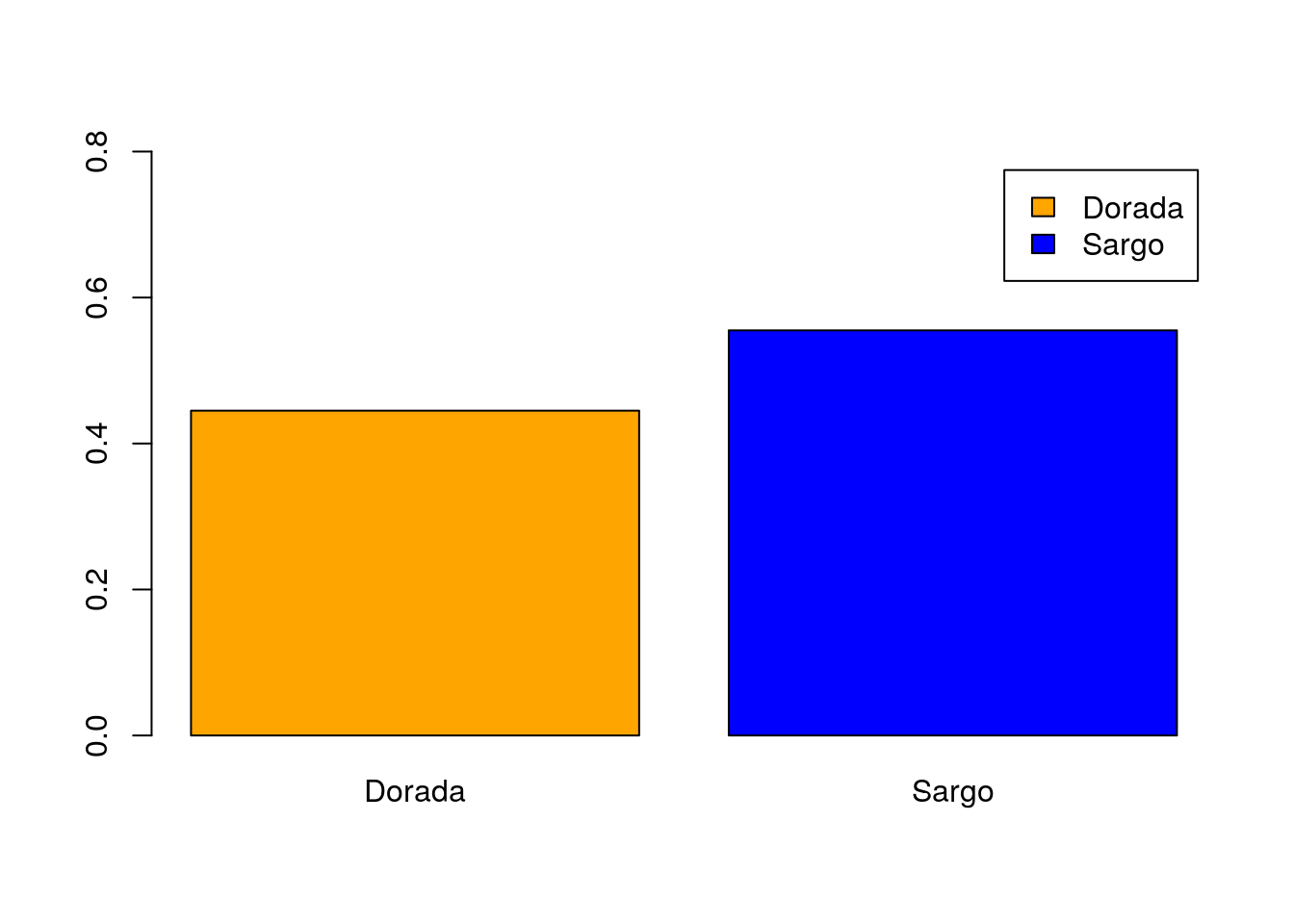
Otra opción es ampliar el eje X, de modo que quede sitio a la izquierda para colocar la leyenda. El problema, en principio, es que como este es un eje de categorías, no muestra valores numéricos, así que ¿como lo ampliamos?. La solución estriba en saber que barplotdevuelve de modo invisible las coordenadas de las posiciones de la base. La manera de capturar dichas coordenadas es asignar el gráfico a una variable y mostrar los valores de ésta (si además indicamos plot=FALSE nos ahorramos que R nos repita el gráfico, aunque tampoco tiene mayor importancia):
xCoords=barplot(prop.table(table(peces$especie)),col=c("orange","blue"),
legend.text=c("Dorada","Sargo"),ylim=c(0,0.8), plot=FALSE)
xCoords## [,1]
## [1,] 0.7
## [2,] 1.9Vemos, por tanto, que las barras están en las posiciones 0.7 y 1.9. Probamos a reconstruir el gráfico fijando la extensión del eje X como xlim=c(0,3.3) (si no quedamos satisfechos de entrada podemos probar unos cuantos valores):
barplot(prop.table(table(peces$especie)),col=c("orange","blue"),
legend.text=c("Dorada","Sargo"),xlim=c(0,3.3))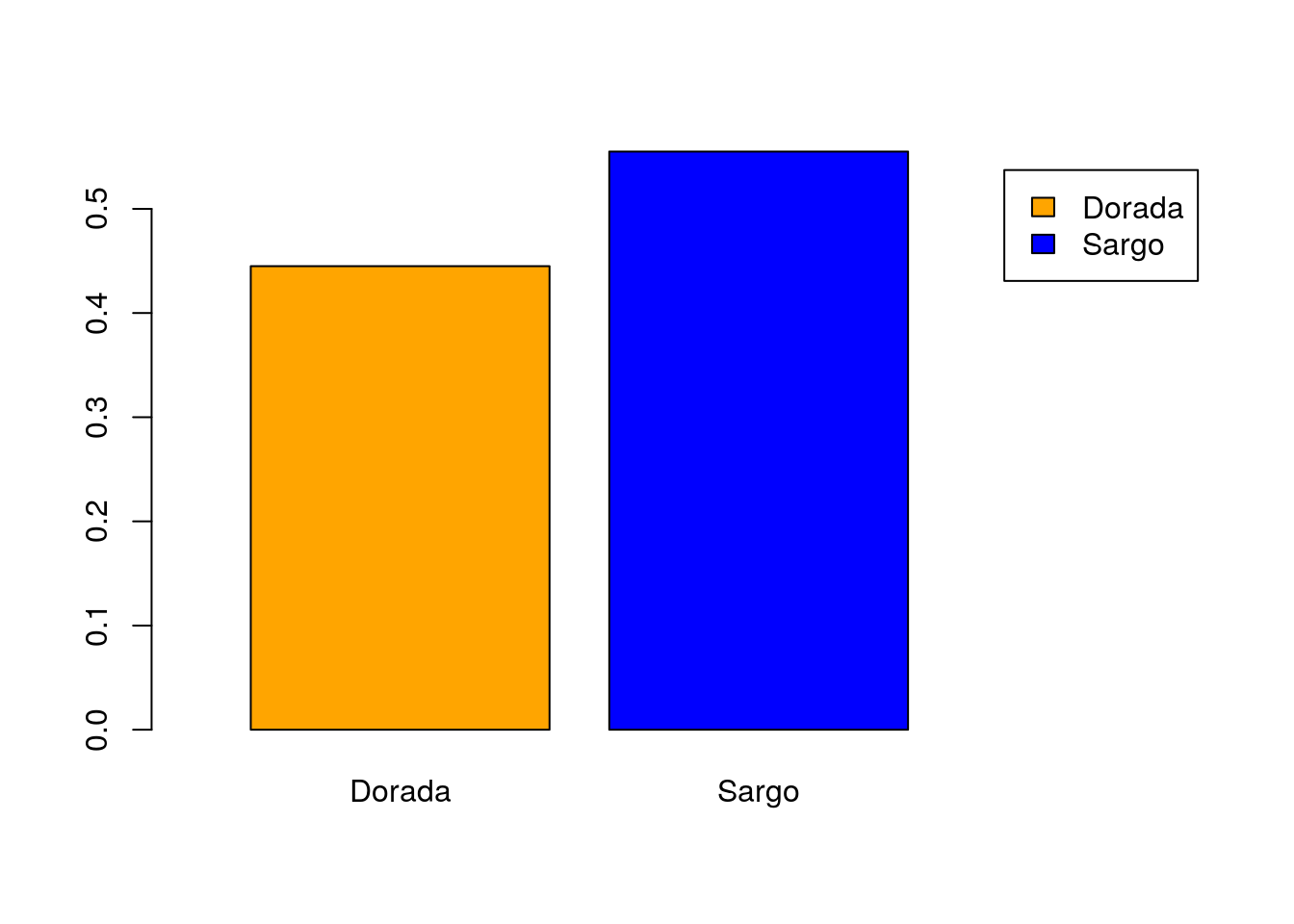
Título
El título se especifica mediante la opción main, escribiendo el texto del título entre comillas:
barplot(prop.table(table(peces$especie)),col=c("orange","blue"),
legend.text=c("Dorada","Sargo"),ylim=c(0,0.8),main="Especie")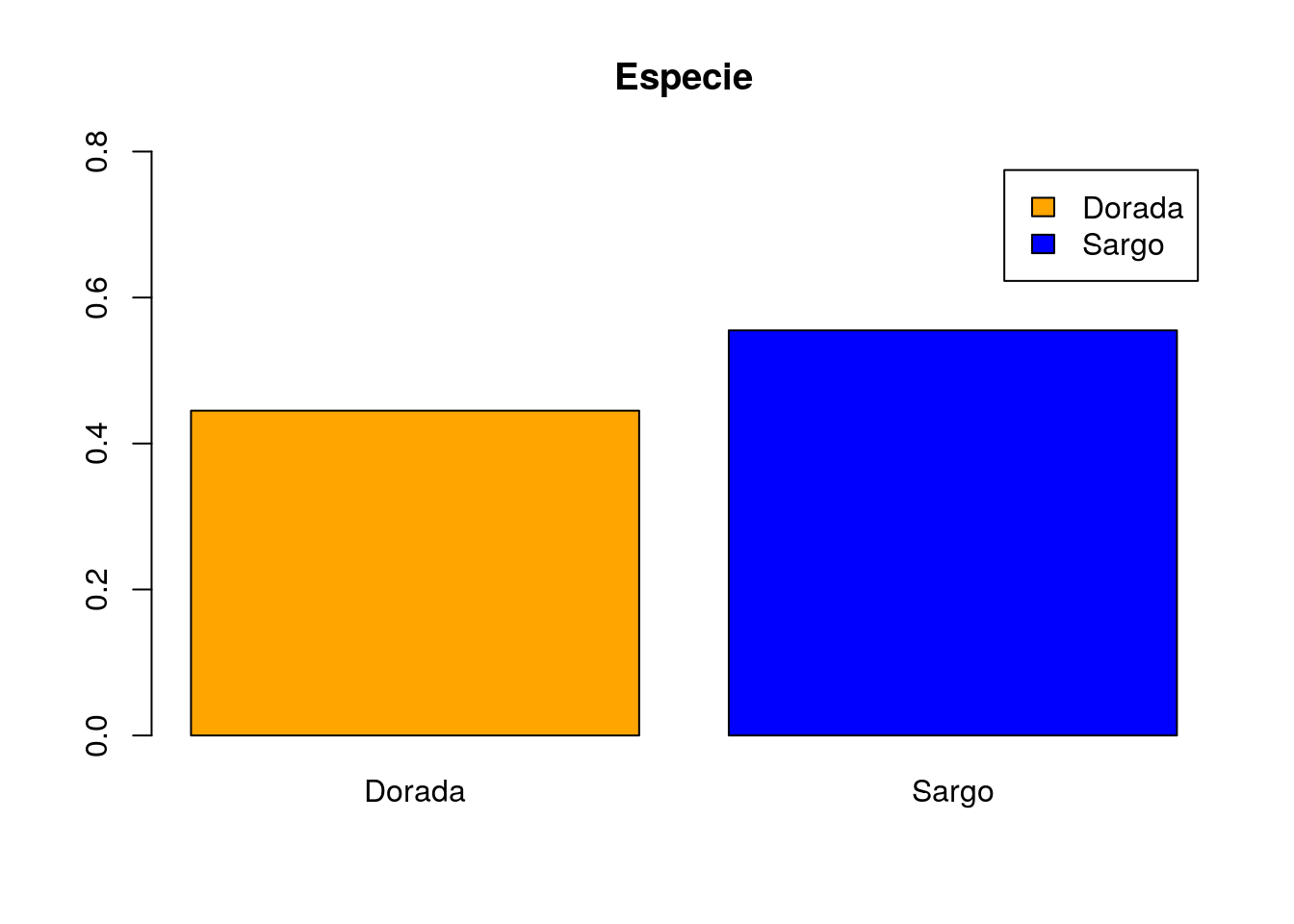
Texto en el eje de ordenadas
Mediante el argumento ylab especificamos el texto (también entre comillas) que deseamos como etiqueta del eje Y:
barplot(prop.table(table(peces$especie)),col=c("orange","blue"),
legend.text=c("Dorada","Sargo"), ylim=c(0,0.8), main="Especie", ylab ="Frecuencias Relativas")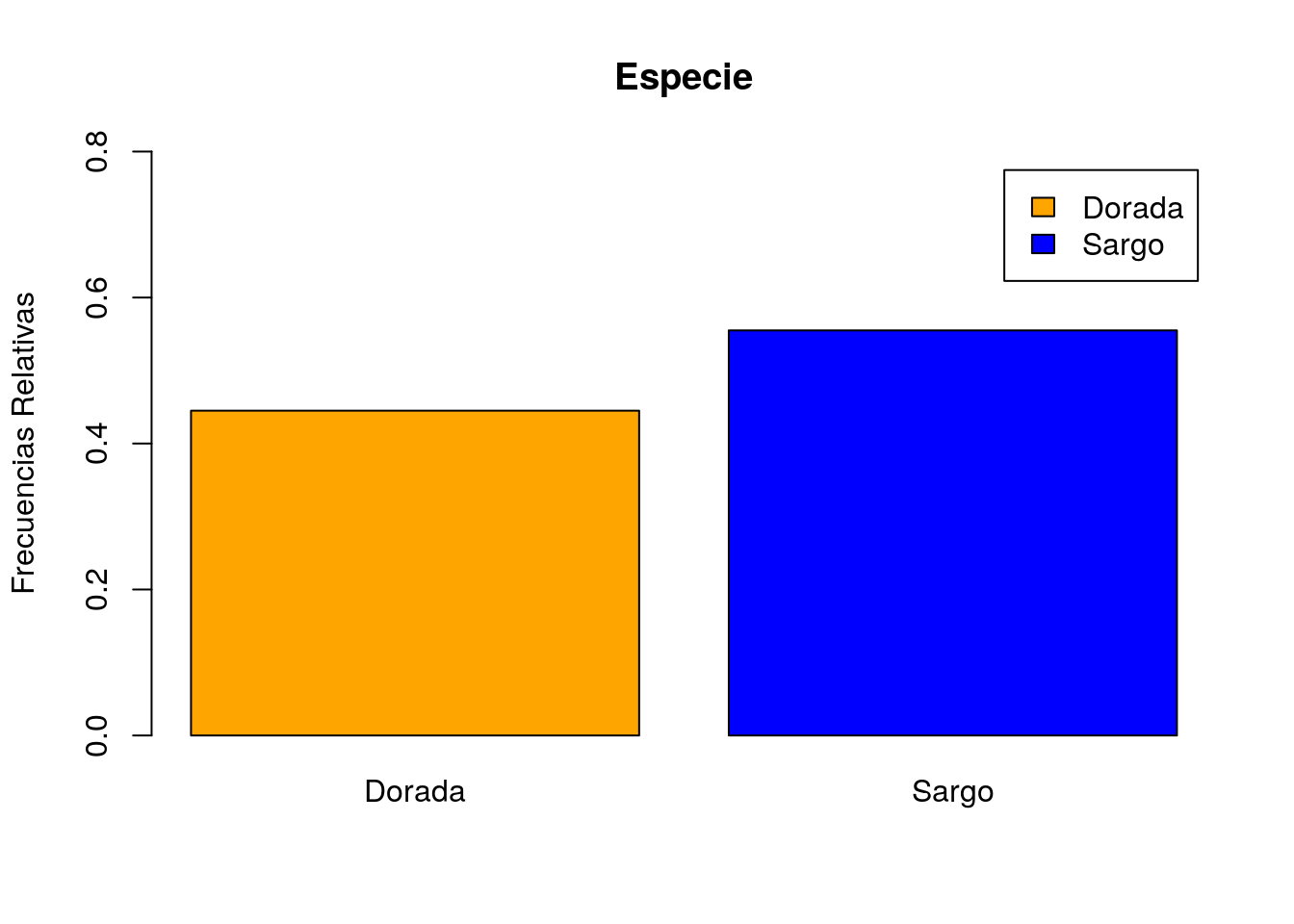
Orientación de las etiquetas en los ejes
El párametro las cambia la orientación de las etiquetas del eje de ordenadas (consultar help(par) para ver las distintas opciones)
barplot(prop.table(table(peces$especie)),col=c("orange","blue"),
legend.text=c("Dorada","Sargo"),ylim=c(0,0.8),main="Especie",
ylab ="Frecuencias Relativas",las=1)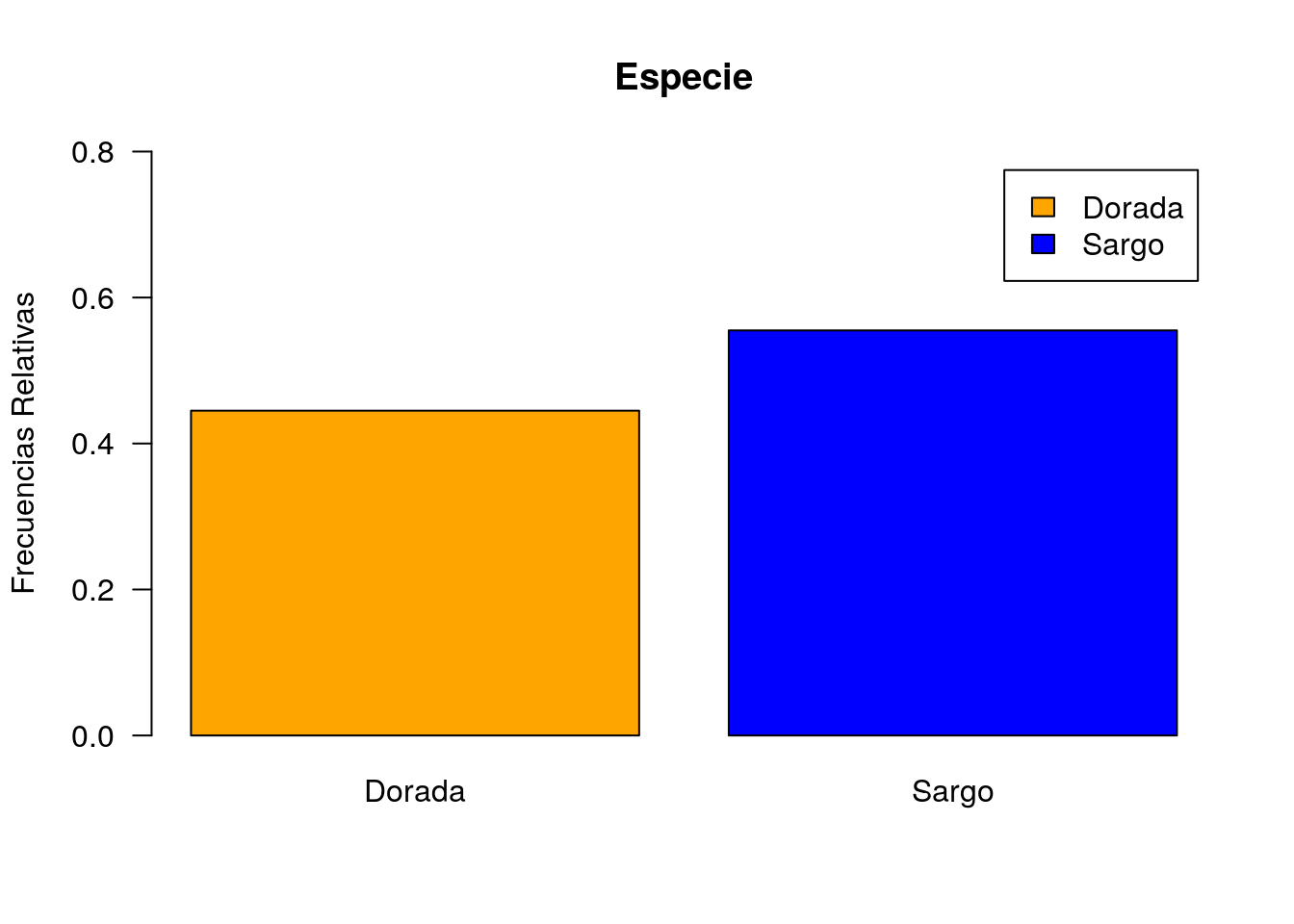
Cambio de la fuente para las letras de las etiquetas
Para cambiar el tipo de fuente que se usa en las letras de las etiquetas del eje de abscisas se utiliza el argumento font.axis (consultar help(par) para ver las distinas fuentes posibles)
barplot(prop.table(table(peces$especie)),col=c("orange","blue"),
legend.text=c("Dorada","Sargo"),main="Especie",ylim=c(0,0.8),
ylab ="Frecuencias Relativas",las=1,font.axis=4)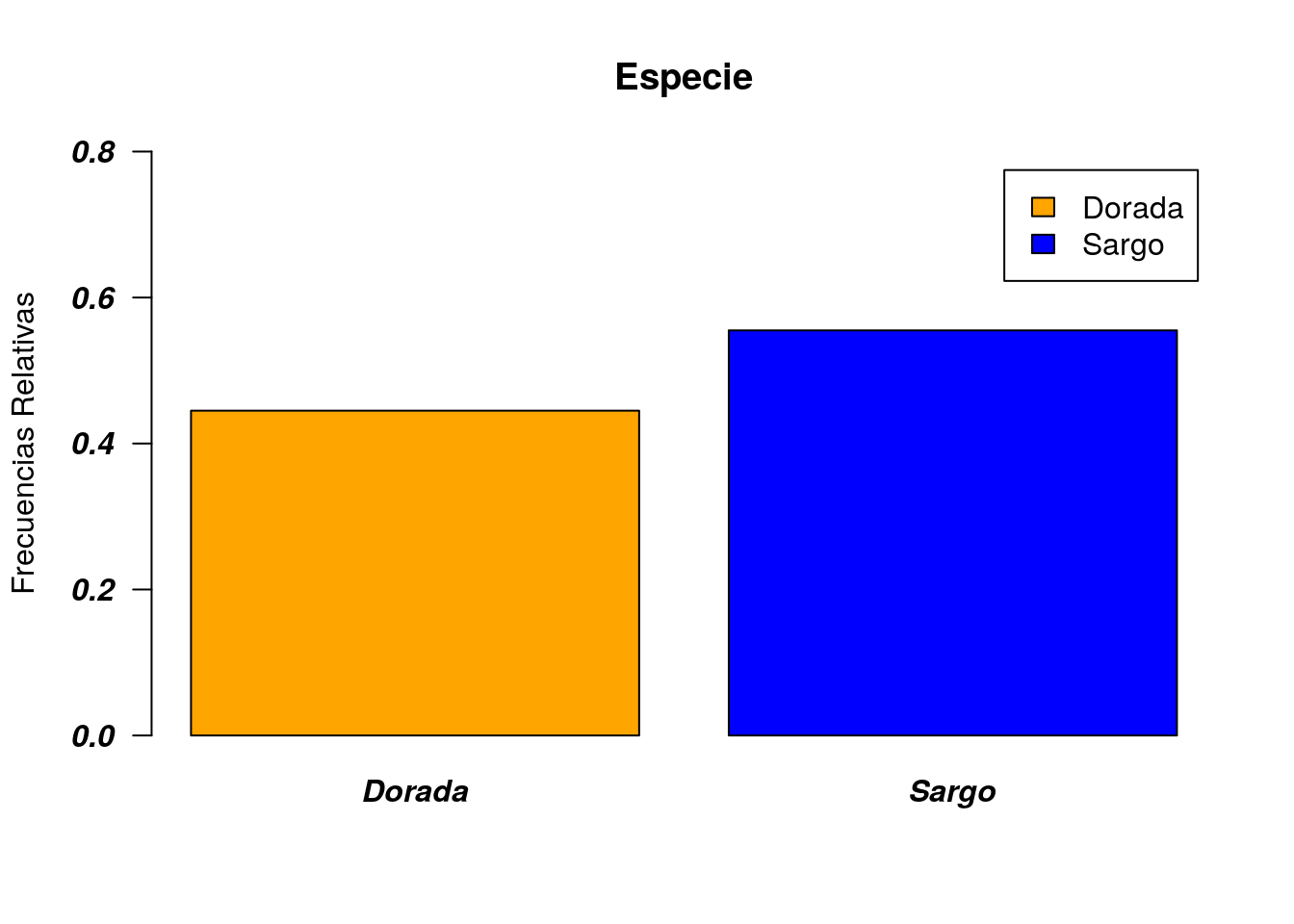
Exportación de una gráfica.
La gráfica anterior aparece en la ventana inferior derecha de Rstudio, en la pestaña Plots. Desde esta ventana podemos guardar la gráfica pinchando en la pestaña Export:

Se despliega entonces una nueva ventana en la que podemos seleccionar los distintos formatos en los que guardar la gráfica (JPEG, PNG, TIFF, BMP, SVG y EPS), además del directorio y nombre que queremos poner al archivo con la imagen:

Paso 2 para grabar gráfica
Construye la siguiente gráfica en la que se representan conjuntamente la frecuencia de cada especie por mes, mejorando todos los aspectos que sean necesarios para que se vea correctamente. Expórtala a un fichero con formato JPEG.
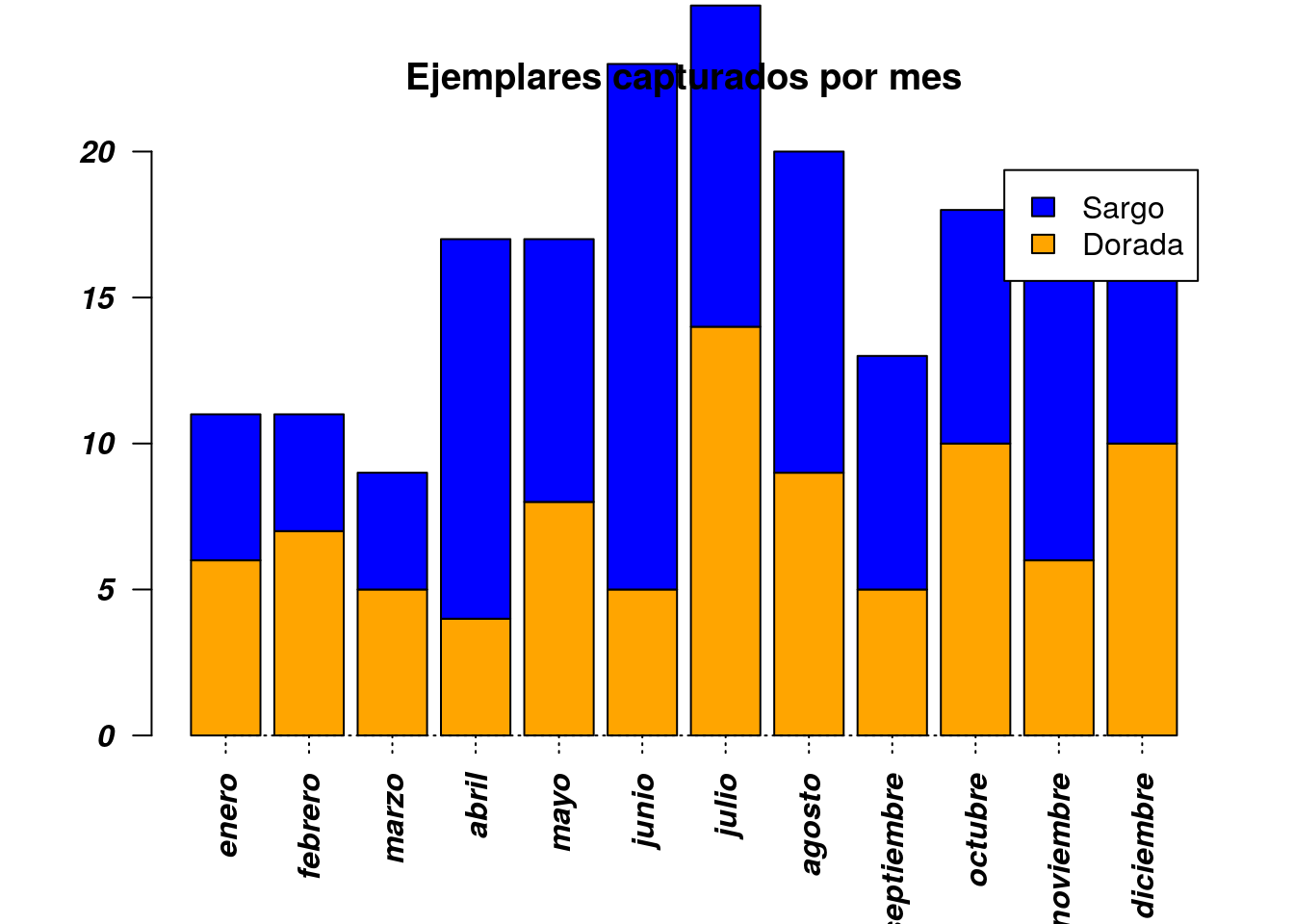
Diagramas de barras utilizando la libreria ggplot2
La librería ggplot2 dispone de procedimientos gráficos más avanzados para la construcción de figuras, así como de una sintaxis propia, que requiere un aprendizaje específico (aunque desde luego, vale la pena cuando se ven los resultados). La gráfica anterior, utilizando ggplot quedaría del siguiente modo:
library(ggplot2)
library(scales)##
## Attaching package: 'scales'## The following object is masked from 'package:plotrix':
##
## rescaleg = ggplot(peces, aes(mes, fill=especie) ) +
labs(title = "Ejemplares capturados por mes")+ylab("") +
theme(plot.title = element_text(size = rel(2), colour = "blue"))
g+geom_bar(position="dodge") + scale_fill_manual(values = alpha(c("orange", "blue"), 1)) +
theme(axis.title.x = element_text(face="bold", size=10)) 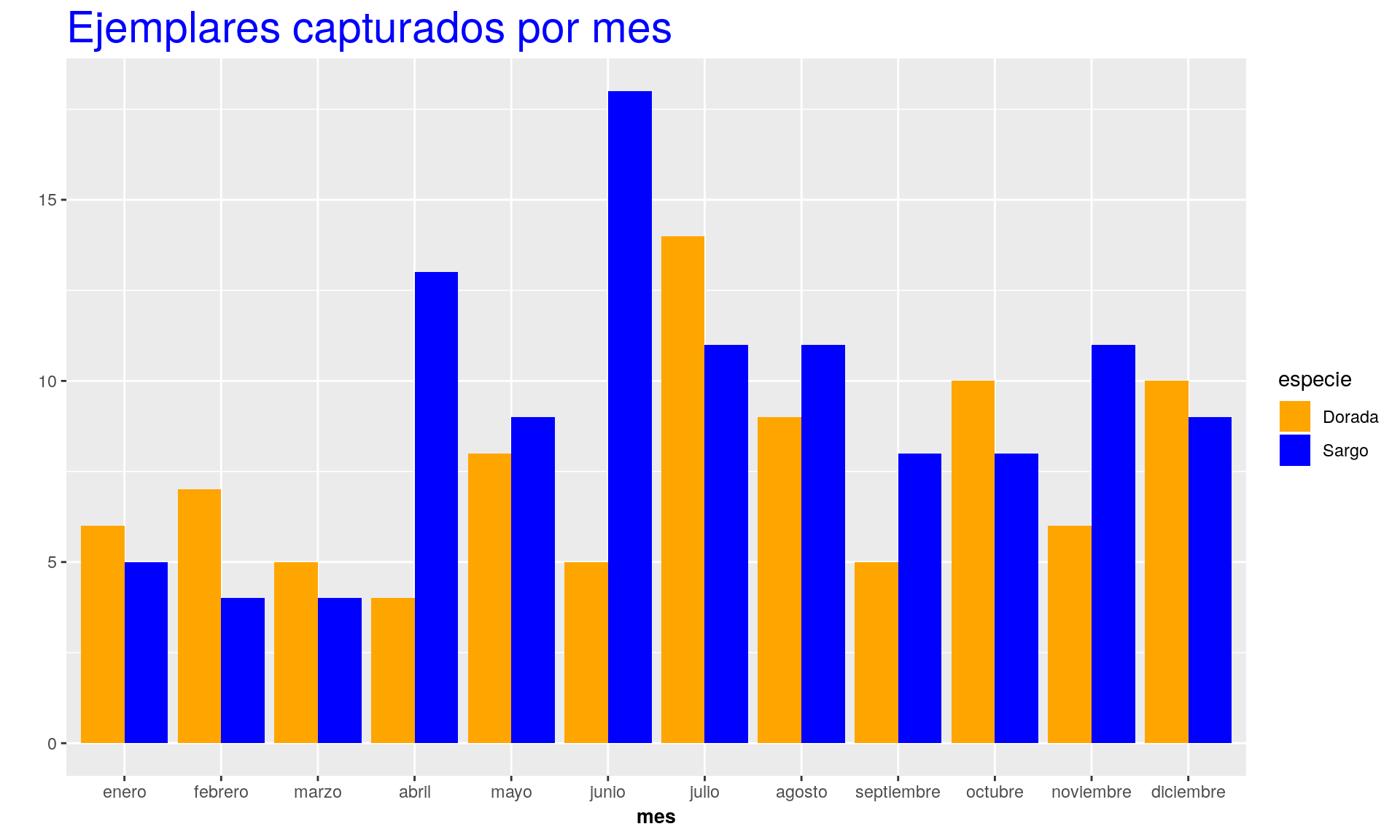
© 2016 Angelo Santana, Carmen N. Hernández, Departamento de Matemáticas ULPGC