Distribuciones de probabilidad en R
Angelo Santana & Carmen Nieves Hernández,
Departamento de Matemáticas, ULPGC
Distribuciones de probabilidad en R
El paquete stats de R (que se instala por defecto al instalar R, y se carga en memoria siempre que iniciamos sesión) implementa numerosas funciones para la realización de cálculos asociados a distintas distribuciones de probabilidad. Entre las utilizadas más comunmente podemos citar:
| Distribuciones Discretas | Distribuciones Continuas | |||
|---|---|---|---|---|
| Distribución | Nombre en R | Distribución | Nombre en R | |
| Binomial | binom | Uniforme | unif | |
| Poisson | pois | Normal | norm | |
| Geométrica | geom | t Student | t | |
| Hipergeométrica | hyper | F Fisher | F | |
| Binomial Negativa | nbinom | Chi-Cuadrado | chisq | |
| Exponencial | exp | |||
| Gamma | gamma | |||
| Weibull | weibull | |||
| W de Wilcoxon | wilcox | |||
Ejecutando:
help(Distributions)se nos muestra el listado de distribuciones de probabilidad disponibles en el paquete stats. Otras distribuciones están disponibles en otros paquetes.
Para cada distribución, R dispone de cuatro funciones. Se puede acceder a cada una de ellas simplemente precediendo el nombre de la distribución que figura en la tabla anterior por la letra que se indica a continuación:
- d: función de densidad o de probabilidad.
- p: función de distribución
- q: función para el cálculo de cuantiles.
- r: función para simular datos con dicha distribución.
Así, por ejemplo, para la distribución normal, la función de densidad se obtiene como dnorm(), la función de distribución como pnorm(), los cuantiles se calculan mediante qnorm() y se pueden generar valores aleatorios con distribución normal mediante rnorm(). Puede consultarse la ayuda, help(dnorm) para conocer la sintaxis específica de estas funciones.
Ejemplo 1: distribución binomial
Si \(X\) sigue una distribución binomial \(B(n,p)\), entonces:
\(P\left(X=k\right)\) =
dbinom(k,n,p)\(P\left(X\le k\right)\) =
pbinom(k,n,p)\(q_{a}=\min\left\{ x:\, P\left(X\le x\right)\ge a\right\}\) =
qbinom(a,n,p)rbinom(m,n,p)genera \(m\) valores aleatorios con esta distribución
Si \(X\approx B(10,0.6)\) tenemos:
- \(P\left(X=8\right)\)
dbinom(8,10,0.6)## [1] 0.1209324- \(P\left(X\le 8\right)\)
pbinom(8,10,0.6)## [1] 0.9536426o también:
sum(dbinom(0:8,10,0.6))## [1] 0.9536426- \(q_{0.95}=\min\left\{ x:\, P\left(X\le x\right)\ge 0.95\right\}\)
qbinom(0.95,10,0.6)## [1] 8Podemos obtener simultáneamente varios cuantiles:
qbinom(c(0.05,0.95),10,0.6)## [1] 3 8- Simulamos 15 valores de esta distribución:
rbinom(15,10,0.6)## [1] 5 6 5 5 5 5 6 6 8 6 7 8 4 6 9Podemos representar fácilmente la función de probabilidad de la distribución binomial:
plot(dbinom(0:10,10,0.6),type="h",xlab="k",ylab="P(X=k)",main="Función de Probabilidad B(10,0.6)")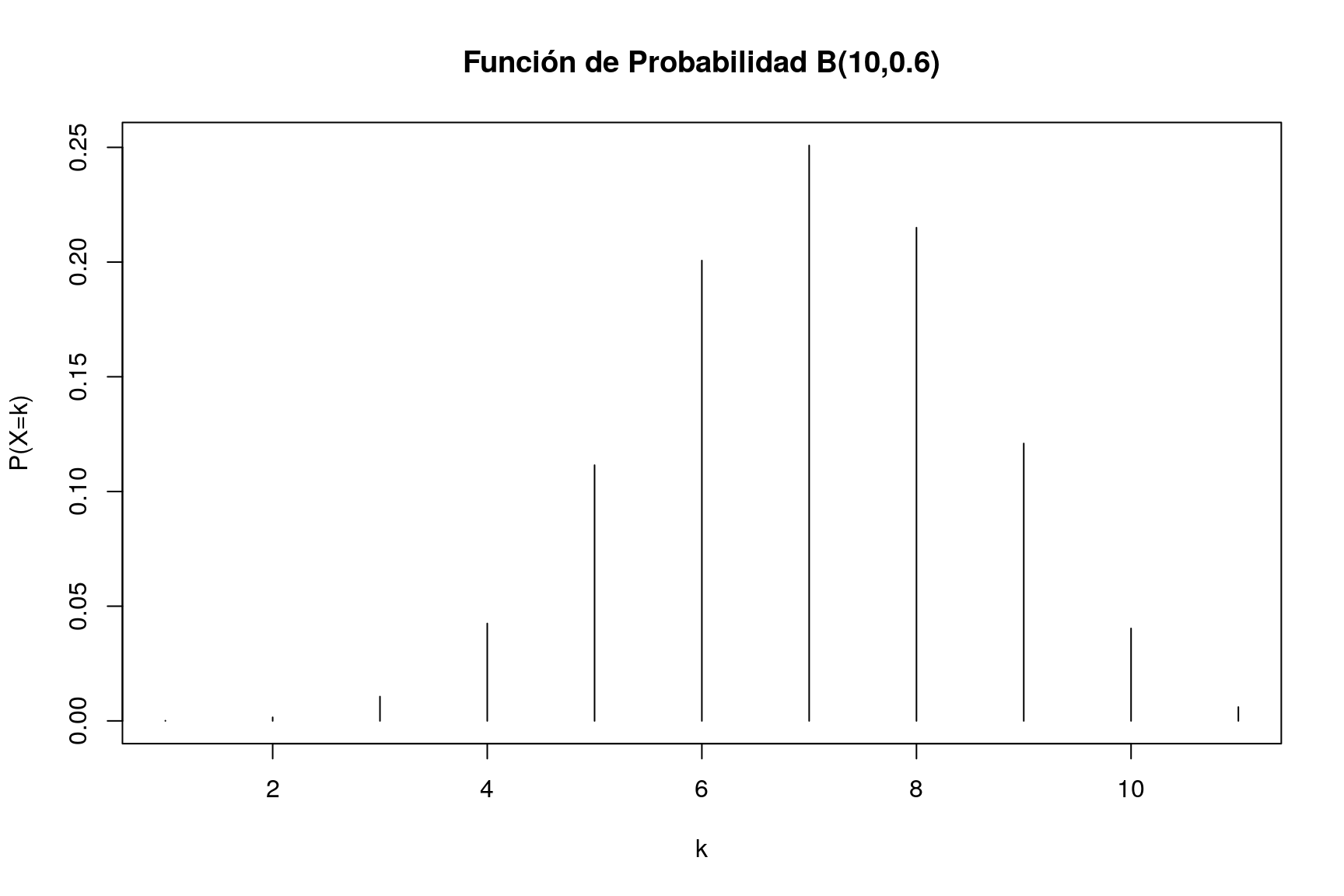
También podemos representar su función de distribución:
plot(stepfun(0:10,pbinom(0:11,10,0.6)),xlab="k",ylab="F(k)",main="Función de distribución B(10,0.6)")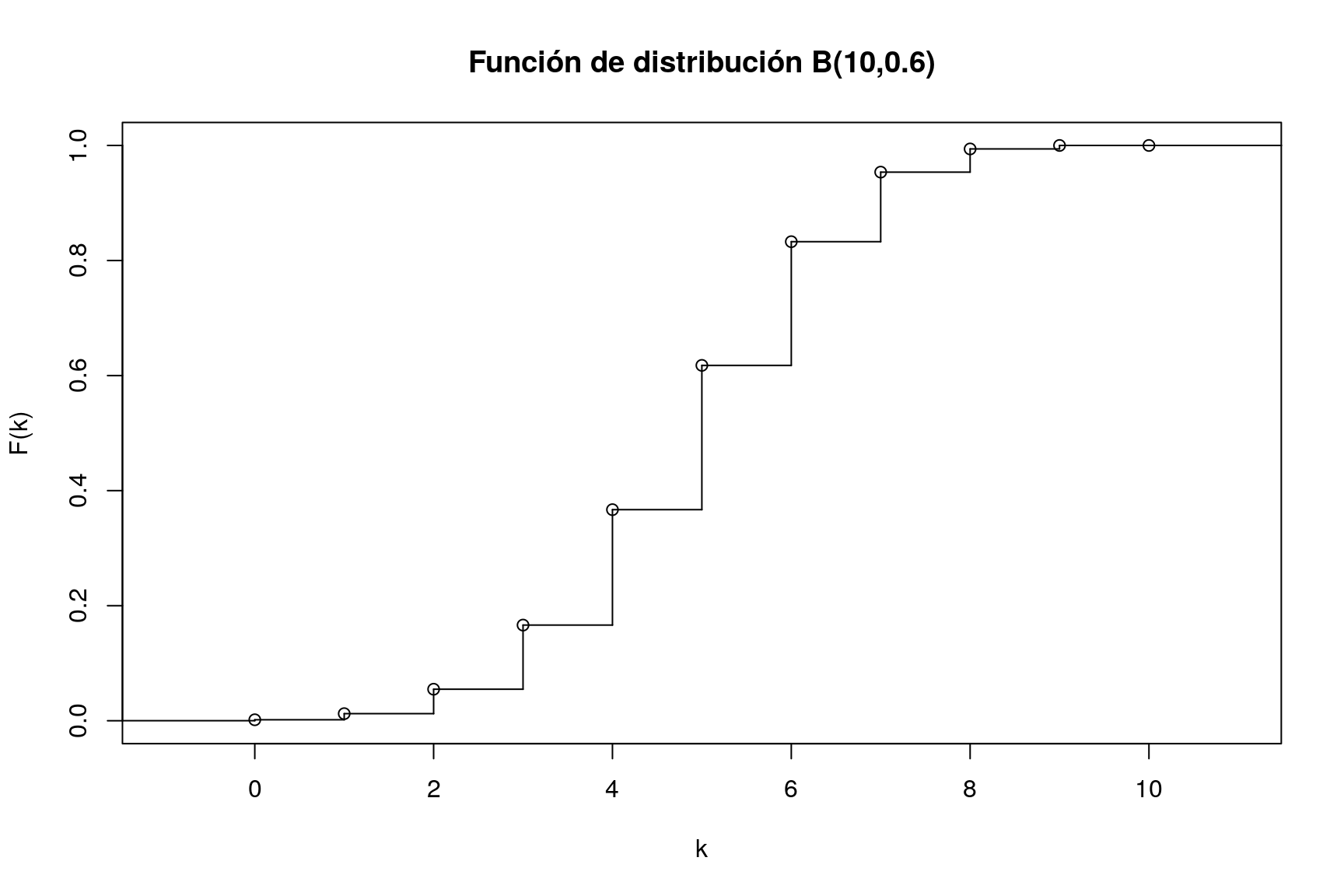
Si simulamos una muestra muy grande de esta distribución (por ejemplo, 10.000 valores), podemos comprobar como las frecuencias relativas son muy similares a las probabilidades teóricas:
X=rbinom(10000, 10, 0.6)
freqAbs=table(X)
freqAbs## X
## 0 1 2 3 4 5 6 7 8 9 10
## 2 20 106 434 1133 2056 2464 2121 1201 409 54freqRel=prop.table(freqAbs)
freqRel## X
## 0 1 2 3 4 5 6 7 8 9
## 0.0002 0.0020 0.0106 0.0434 0.1133 0.2056 0.2464 0.2121 0.1201 0.0409
## 10
## 0.0054Vamos a mostrar lado a lado las frecuencias relativas y las probabilidades teóricas. Para ello, generamos primero un dataframe con la tabla de probabilidades teóricas:
probsTeo=data.frame(X=0:10,Prob=dbinom(0:10,10,0.6))
probsTeo## X Prob
## 1 0 0.0001048576
## 2 1 0.0015728640
## 3 2 0.0106168320
## 4 3 0.0424673280
## 5 4 0.1114767360
## 6 5 0.2006581248
## 7 6 0.2508226560
## 8 7 0.2149908480
## 9 8 0.1209323520
## 10 9 0.0403107840
## 11 10 0.0060466176Para presentar en una única tabla las probabilidades teóricas y las frecuencias relativas de nuesta simulación, podemos utilizar la función merge. Esta función combina dataframes que tengan un campo en común (en este caso el valor de la variable \(X\)). El objeto probsTeo es obviamente un dataframe. Ahora bien, mediante class(freqRel) podemos comprobar que freqRel es un objeto de clase table. Para poder combinarlo con probsTeo hemos de convertirlo primero en dataframe:
freqRel=as.data.frame(freqRel)
str(freqRel)## 'data.frame': 11 obs. of 2 variables:
## $ X : Factor w/ 11 levels "0","1","2","3",..: 1 2 3 4 5 6 7 8 9 10 ...
## $ Freq: num 0.0002 0.002 0.0106 0.0434 0.1133 ...Vemos aquí que la variable X es de tipo factor; para poder combinar bien este dataframe con probsTeo hemos de convertir X a la clase integer (ya que en ese dataframe, X es entera, como puede comprobarse mediante str(probsTeo)):
freqRel$X=as.integer(as.character(freqRel$X))Por último procedemos a combinar ambos objetos mediante la función merge y mostramos el resultado:
compara=merge(freqRel,probsTeo,all=TRUE)
compara## X Freq Prob
## 1 0 0.0002 0.0001048576
## 2 1 0.0020 0.0015728640
## 3 2 0.0106 0.0106168320
## 4 3 0.0434 0.0424673280
## 5 4 0.1133 0.1114767360
## 6 5 0.2056 0.2006581248
## 7 6 0.2464 0.2508226560
## 8 7 0.2121 0.2149908480
## 9 8 0.1201 0.1209323520
## 10 9 0.0409 0.0403107840
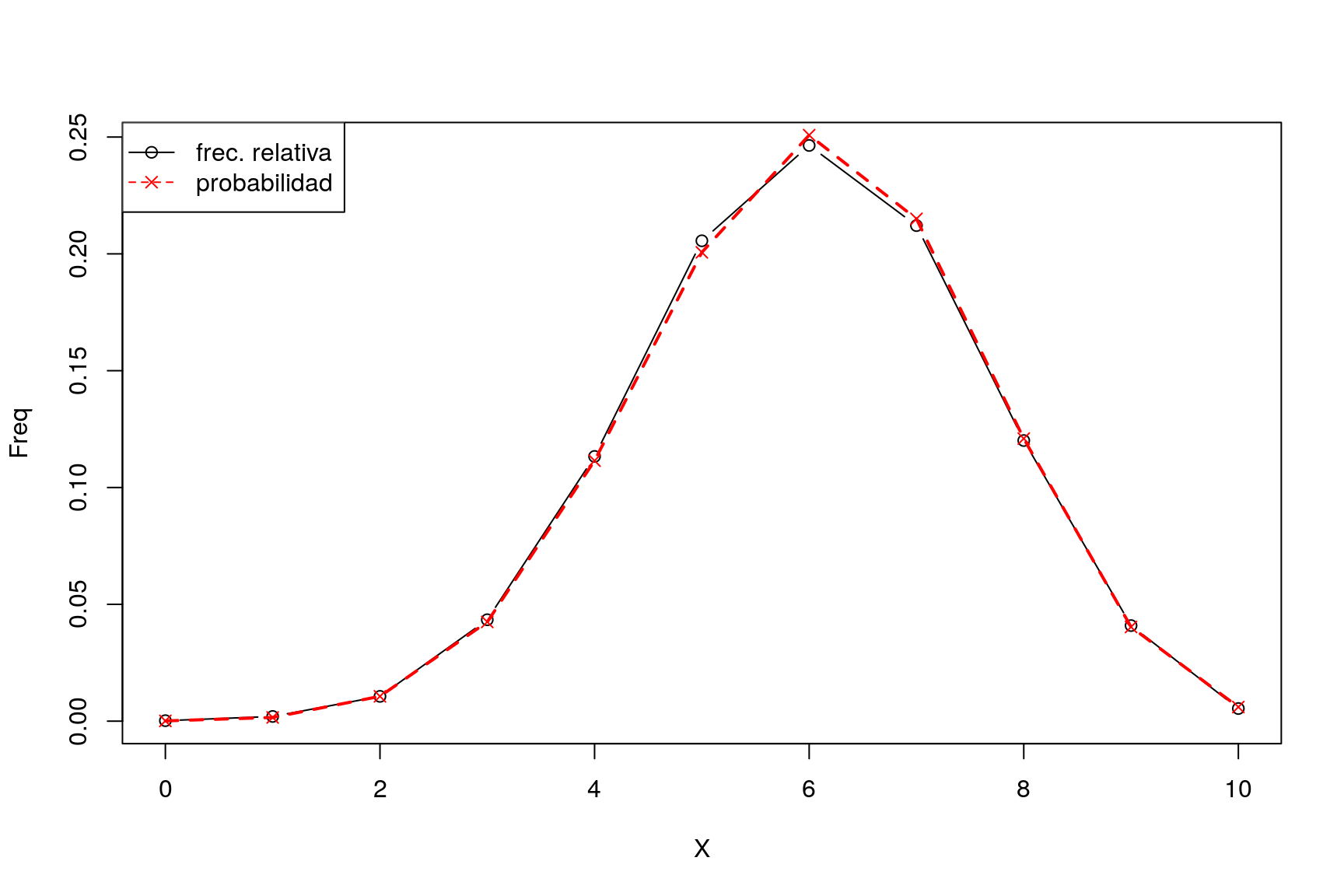
## 11 10 0.0054 0.0060466176Podemos hacer un gráfico que nos muestre la similitud entre probabilidad y frecuencia relativa:
with(compara,{
plot(X,Freq, type="b")
points(X,Prob,col="red",pch=4)
lines(X,Prob,col="red",lty=2,lwd=2)
legend("topleft",c("frec. relativa","probabilidad"),col=c("black","red"),lty=1:2,pch=c(1,4))
})
Ejemplo2: Distribución normal.
Si \(X\) sigue una distribución normal \(N\left(\mu,\sigma\right)\), entonces:
\(f\left(x\right)\) =
dnorm(x,mu,sigma)\(P\left(X\le k\right)\) =
pnorm(x,mu,sigma)\(q_{a}=\min\left\{ x:\, P\left(X\le x\right)\ge a\right\}\) =
qnorm(a,mu,sigma)rnorm(n,mu,sigma)genera \(n\) valores aleatorios \(N\left(\mu,\sigma\right)\)
Supongamos que \(X\approx N\left(170,12\right)\). Entonces:
- Calculamos \(f\left(178\right)\)
dnorm(171,170,12)## [1] 0.03312996- Podemos calcular fácilmente los valores de la función de densidad sobre una secuencia de valores de \(x\):
x=seq(165,175,by=0.5)
dnorm(x,170,12)## [1] 0.03048103 0.03098792 0.03144860 0.03186077 0.03222234 0.03253150
## [7] 0.03278664 0.03298647 0.03312996 0.03321634 0.03324519 0.03321634
## [13] 0.03312996 0.03298647 0.03278664 0.03253150 0.03222234 0.03186077
## [19] 0.03144860 0.03098792 0.03048103- La representación gráfica de la función de densidad se obtiene fácilmente como:
curve(dnorm(x,170,12),xlim=c(130,210),col="blue",lwd=2,
xlab="x",ylab="f(x)",main="Función de Densidad N(170,12)")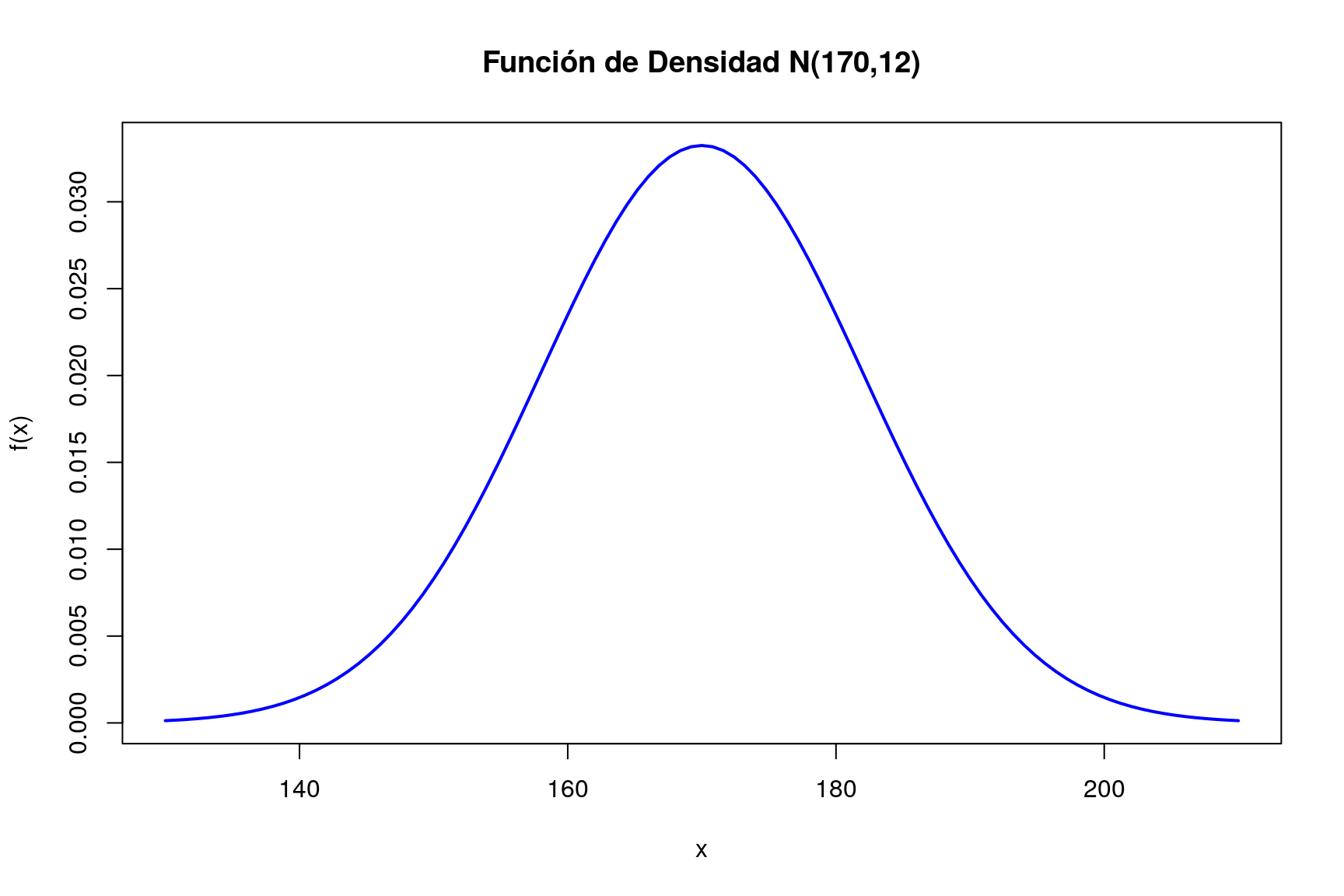
- También podemos representar la función de distribución:
curve(pnorm(x,170,12),xlim=c(130,210),col="blue",lwd=2,
xlab="x",ylab="F(x)",main="Función de Distribución N(170,12)")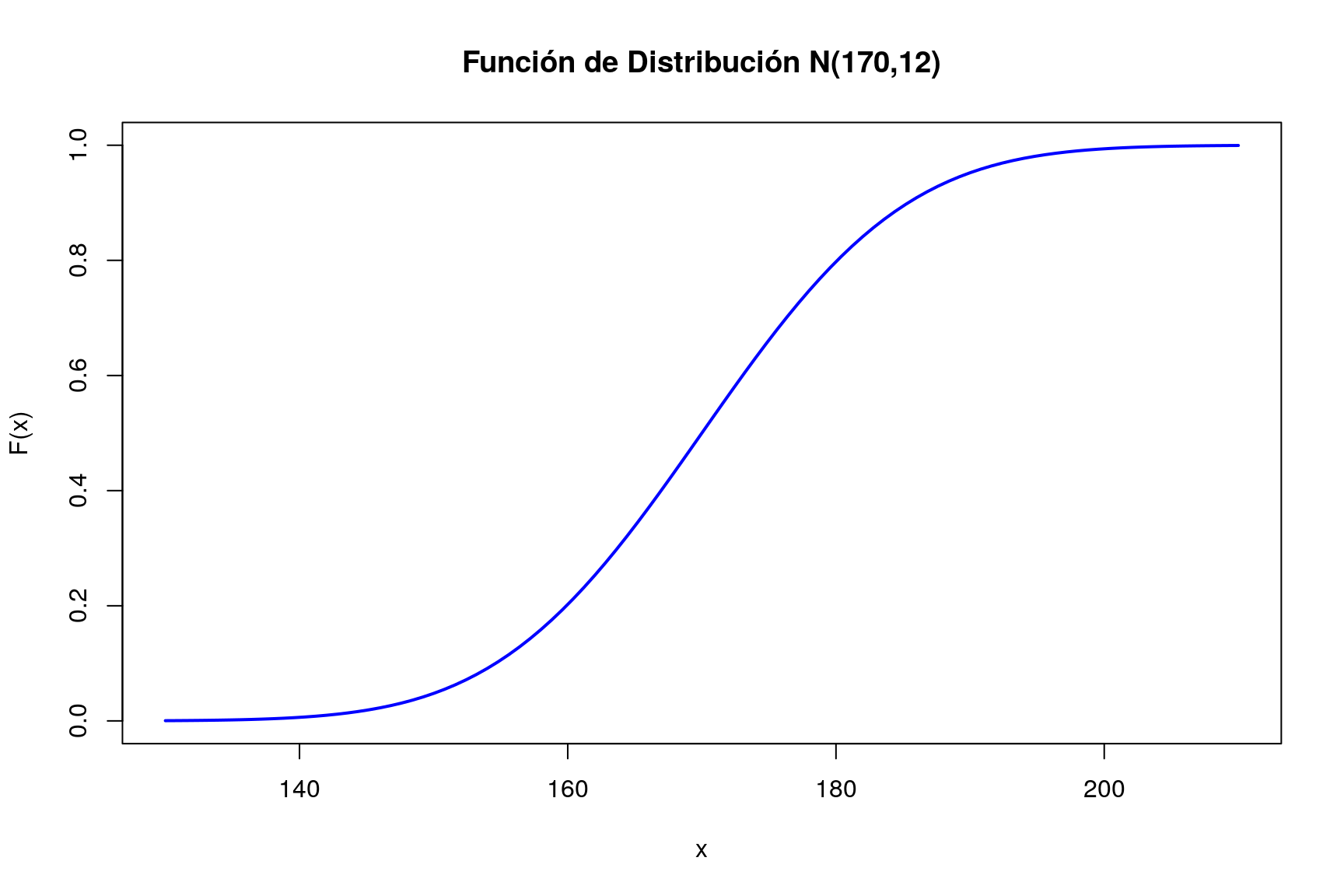
- Calculamos la probabilidad \(P\left(X\le 180\right)\)
pnorm(180,170,12)## [1] 0.7976716- \(P\left(X\gt 168\right)\)
1-pnorm(168,170,12)## [1] 0.5661838o también
pnorm(168,170,12, lower.tail=FALSE)## [1] 0.5661838- \(P\left(150\le X\le 168\right)\)
pnorm(168,170,12)-pnorm(150,170,12)## [1] 0.3860258o también:
miDensidad=function(x) dnorm(x,170,12)
integrate(miDensidad,150,168)## 0.3860258 with absolute error < 4.3e-15No es demasiado difícil representar el área correspondiente a la probabilidad que se acaba de calcular:
regionX=seq(150,168,0.01) # Intervalo a sombrear
xP <- c(150,regionX,168) # Base de los polígonos que crean el efecto "sombra"
yP <- c(0,dnorm(regionX,170,12),0) # Altura de los polígonos sombreados
curve(dnorm(x,170,12),xlim=c(130,210),yaxs="i",ylim=c(0,0.035),ylab="f(x)",
main='Densidad N(170,12)')
polygon(xP,yP,col="orange1")
box()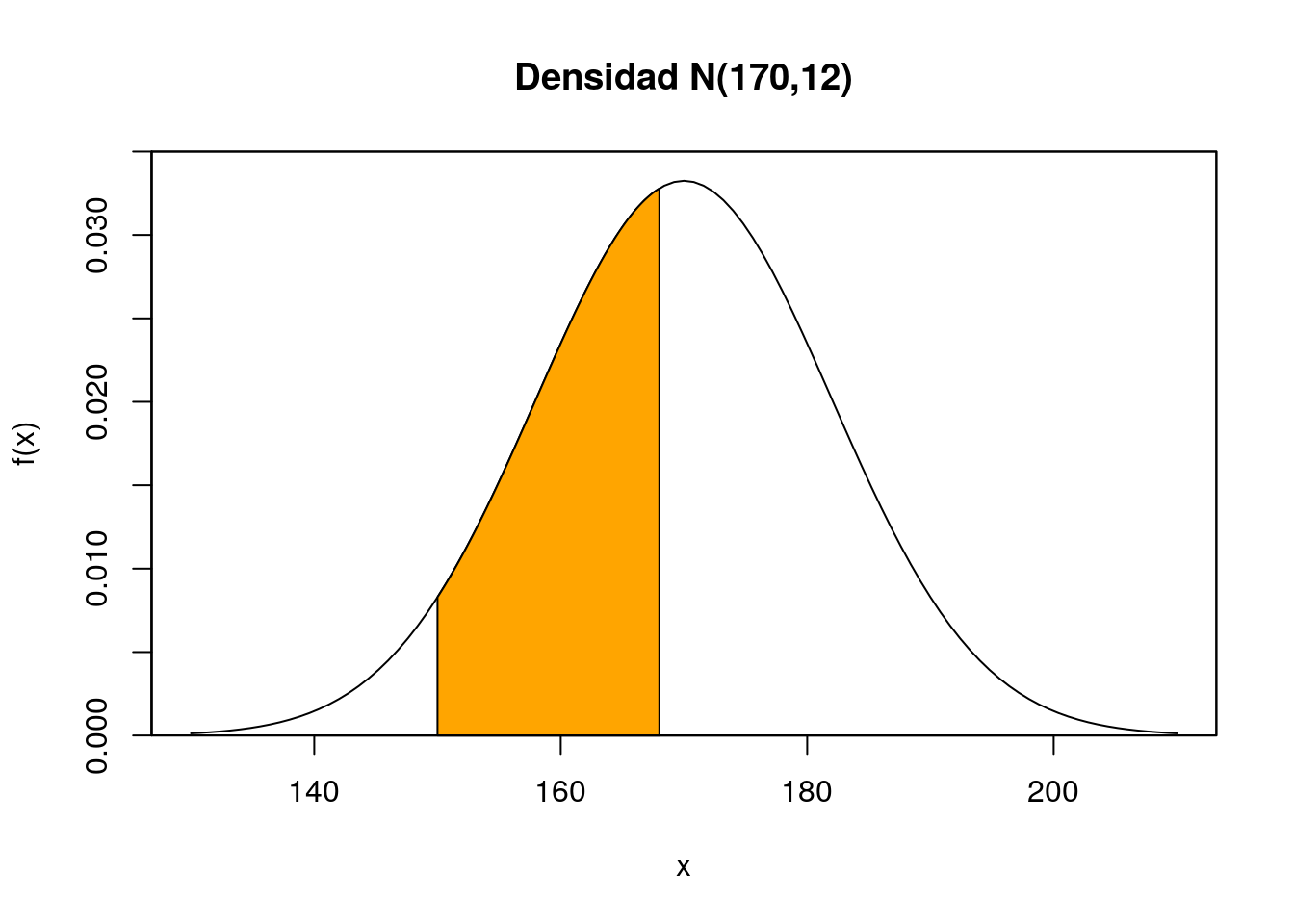
- \(z_{0.95}=\min\left\{ x:\, P\left(X\le x\right)\ge 0.95\right\}\)
qnorm(0.95,170,12)## [1] 189.7382- Si no se especifican media y varianza, R entiende que trabajamos con la distribución normal estándar. La siguiente instrucción nos proporciona los cuantiles 0.025 y 0.975 de la \(N(0,1)\):
qnorm(c(0.025,0.975))## [1] -1.959964 1.959964- Simulamos 10 valores de esta distribución:
rnorm(10,170,12)## [1] 176.4797 172.3405 163.0540 171.7483 170.2532 168.9778 173.6709
## [8] 186.1627 156.3749 166.4526- Simulamos una muestra grande de la distribución normal y comprobamos que el histograma es muy parecido a la función de densidad:
X=rnorm(10000, 170, 12)
hist(X,freq=FALSE,col="lightsalmon",main="Histograma",sub="Datos simulados de una N(170,12)")
curve(dnorm(x,170,12),xlim=c(110,220),col="blue",lwd=2,add=TRUE)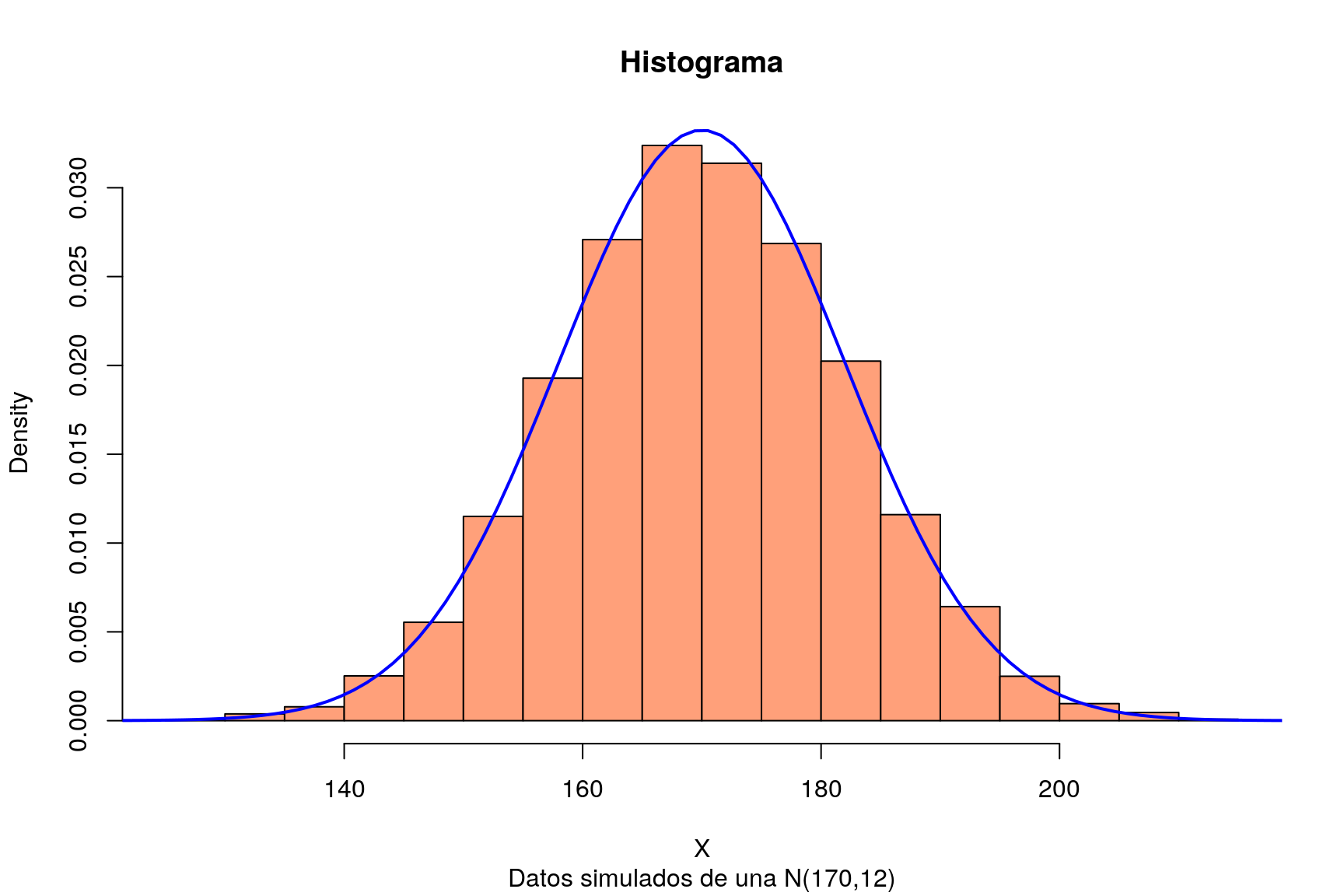
Podemos comprobar también que la distribución acumulativa empírica de esta simulación es muy similar a la función de distribución teórica de la normal:
plot(ecdf(X))
curve(pnorm(x,170,12),xlim=c(110,220),col="red",lwd=2,lty=2,add=TRUE)
legend("topleft",lty=c(1,2),lwd=c(1,2),col=c("black","red"),legend=c("Distribución empírica","Distribución teórica"))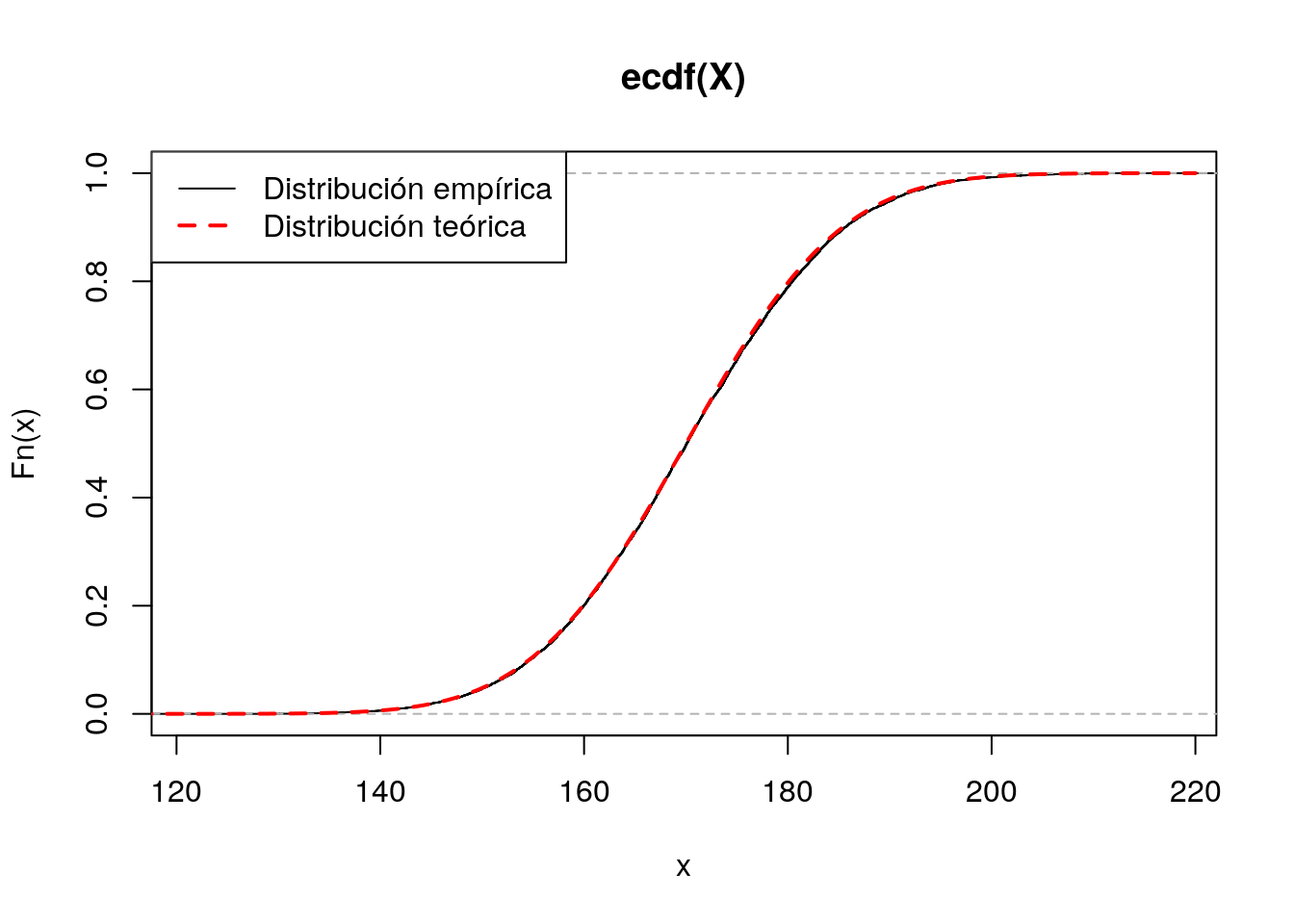
Distribución de la media en el muestreo. Teorema central del límite.
R es una herramienta excelente para ayudar a entender el significado de muchos conceptos estadísticos. Un concepto a menudo mal entendido es el de la distribución de un estadístico en el muestreo. Para aclarar este concepto podemos comenzar por realizar un sencillo experimento: tomar una muestra aleatoria simple en una población normal de, por ejemplo, media \(\mu=170\) y desviación típica \(\sigma=12\). Fijamos inicialmente un tamaño de muestra \(n=25\), tomamos la muestra y calculamos su media:
n=25
muestra1=rnorm(n,170,12)
media1=mean(muestra1)
media1## [1] 172.1519En esta muestra hemos obtenido un valor medio muestral de 172.2. Si repetimos el proceso obtenemos otra media distinta:
muestra2=rnorm(n,170,12)
media2=mean(muestra2)
media2## [1] 170.9573Y si volvemos a repetir:
muestra3=rnorm(n,170,12)
media3=mean(muestra3)
media3## [1] 173.8301En este punto el lector habrá observado que cada vez que se toma una muestra se obtiene una media diferente. Como a priori es imposible predecir en cada muestreo cuál será el valor medio resultante, la media muestral es una variable aleatoria. El lector habrá observado también que las medias muestrales se parecen a la media de la población, \(\mu=170\). Cabe preguntarse entonces: ¿tenderá la media muestral a comportarse de esta manera en todas las muestras? ¿Cuánto llega a apartarse de la media de la población?
Para responder a estas preguntas podemos repetir el proceso anterior no tres, sino muchísimas más veces. En R esto es muy sencillo de hacer. Una forma muy eficiente de replicar muchas veces el proceso anterior consiste en encapsular el proceso de muestreo en una función que haremos depender del tamaño de la muestra:
mediaMuestral=function(n){
muestra=rnorm(n,170,12)
media=mean(muestra)
return(media)
}Cada vez que ejecutemos esta función estaremos eligiendo una muestra de tamaño \(n\) de esa población \(N(170,12)\) y calculando su media:
mediaMuestral(25)## [1] 167.8375mediaMuestral(25)## [1] 170.2032mediaMuestral(25)## [1] 169.7148Para repetir \(m\) veces el proceso de extraer una muestra de tamaño \(n\) y calcular su media podemos utilizar la función replicate():
m=10000
muchasMedias=replicate(m,mediaMuestral(25))Las primeras 20 medias obtenidas en esta simulación son:
muchasMedias[1:20]## [1] 170.0775 168.9949 168.2631 172.9702 168.5740 171.2849 167.9372
## [8] 171.3661 168.6483 168.5249 174.9818 166.0221 165.4734 172.2704
## [15] 168.2311 171.9379 173.6650 169.6938 170.6365 170.8489La media y desviación típica de todas estas medias muestrales son:
mean(muchasMedias)## [1] 169.98sd(muchasMedias)## [1] 2.4088Por último, representamos gráficamente la distribución de frecuencias de estas medias muestrales mediante un histograma, al que le superponemos una densidad normal:
hist(muchasMedias,xlab="Media muestral", ylab="Frecuencia", col="lightcyan",
xlim=c(160,180),freq=FALSE,ylim=c(0,0.75),
main="Histograma de las medias muestrales observadas\n en 10000 muestras de tamaño 25")
curve(dnorm(x,170,sd(muchasMedias)),xlim=c(160,180),col="blue",lwd=2,add=TRUE) 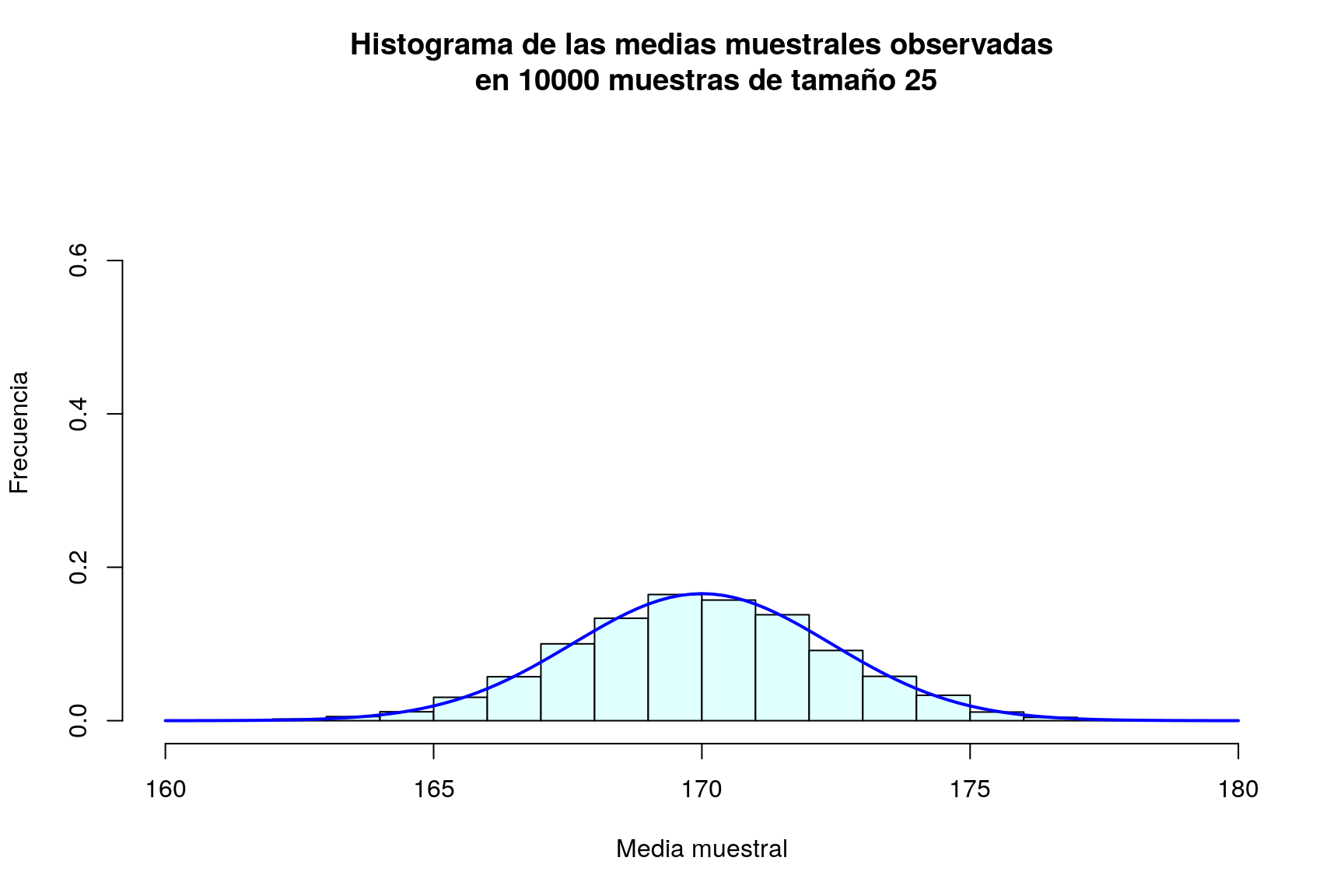
A estas alturas, para el lector debe de haber quedado claro que la media muestral es una variable aleatoria, y que su distribución de probabilidad es muy parecida a la normal; además la media de todas las medias muestrales es casi idéntica a la media de la población.
¿Cuál es el efecto de aumentar el tamaño de la muestra? Podemos evaluarlo repitiendo el proceso anterior para \(n=50\), \(n=100\) y \(n=500\) (en todos los casos presentaremos los gráficos en la misma escala para facilitar la comparación):
muchasMedias50=replicate(m,mediaMuestral(50))
muchasMedias100=replicate(m,mediaMuestral(100))
muchasMedias500=replicate(m,mediaMuestral(500))
mean(muchasMedias50); sd(muchasMedias50)## [1] 170## [1] 1.6959mean(muchasMedias100); sd(muchasMedias100)## [1] 170## [1] 1.1843mean(muchasMedias500); sd(muchasMedias500)## [1] 170## [1] 0.53569hist(muchasMedias50,xlab="Media muestral", ylab="Frecuencia", col="lightcyan",
xlim=c(160,180),freq=FALSE,ylim=c(0,0.75),
main="Histograma de las medias muestrales observadas\n en 10000 muestras de tamaño 50")
curve(dnorm(x,170,sd(muchasMedias50)),xlim=c(160,180),col="blue",lwd=2,add=TRUE)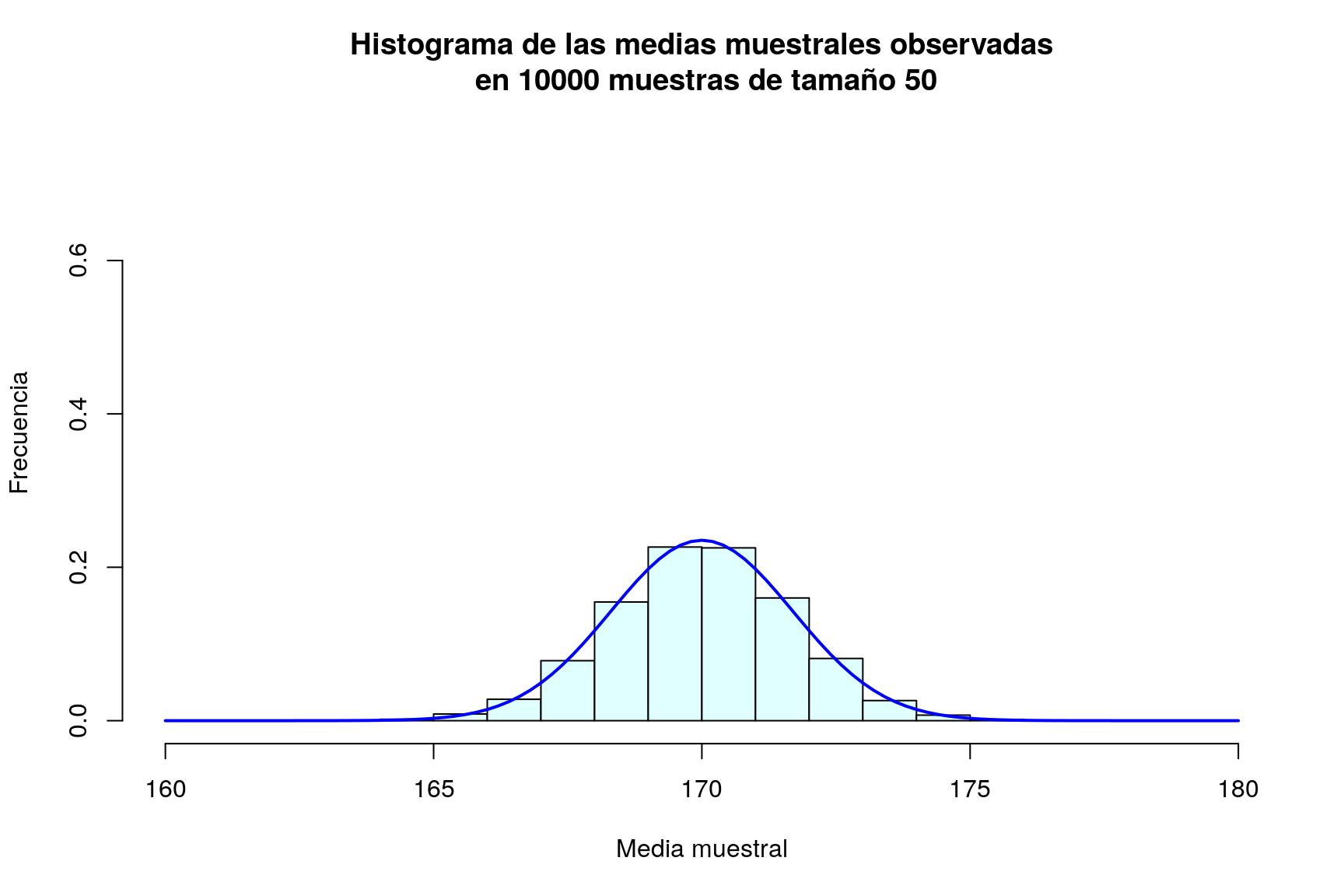
hist(muchasMedias100,xlab="Media muestral", ylab="Frecuencia", col="lightcyan",
xlim=c(160,180),freq=FALSE,ylim=c(0,0.75),
main="Histograma de las medias muestrales observadas\n en 10000 muestras de tamaño 100")
curve(dnorm(x,170,sd(muchasMedias100)),xlim=c(160,180),col="blue",lwd=2,add=TRUE)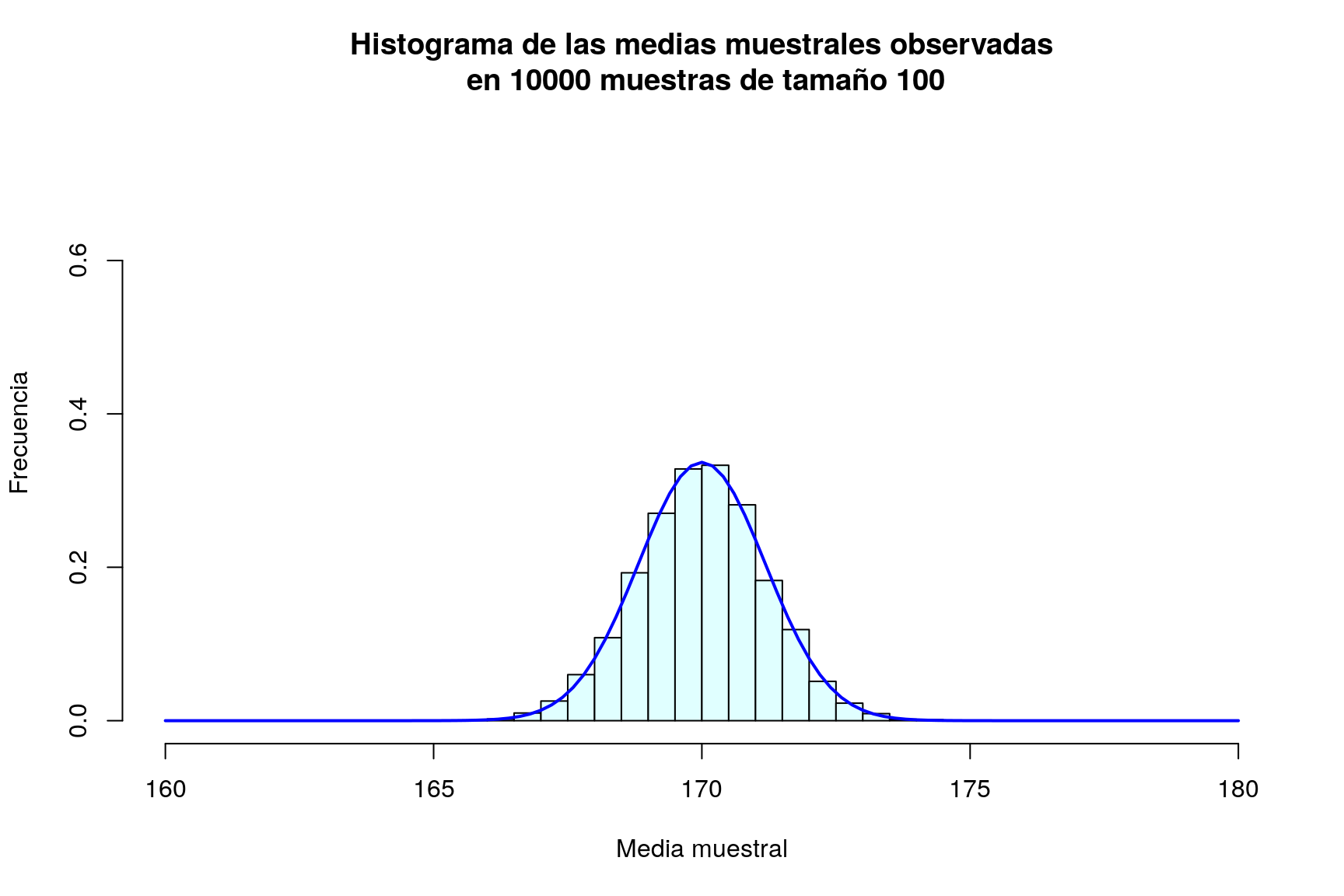
hist(muchasMedias500,xlab="Media muestral", ylab="Frecuencia", col="lightcyan",
xlim=c(160,180),freq=FALSE,ylim=c(0,0.75),
main="Histograma de las medias muestrales observadas\n en 10000 muestras de tamaño 500")
curve(dnorm(x,170,sd(muchasMedias500)),xlim=c(160,180),col="blue",lwd=2,add=TRUE,n=200)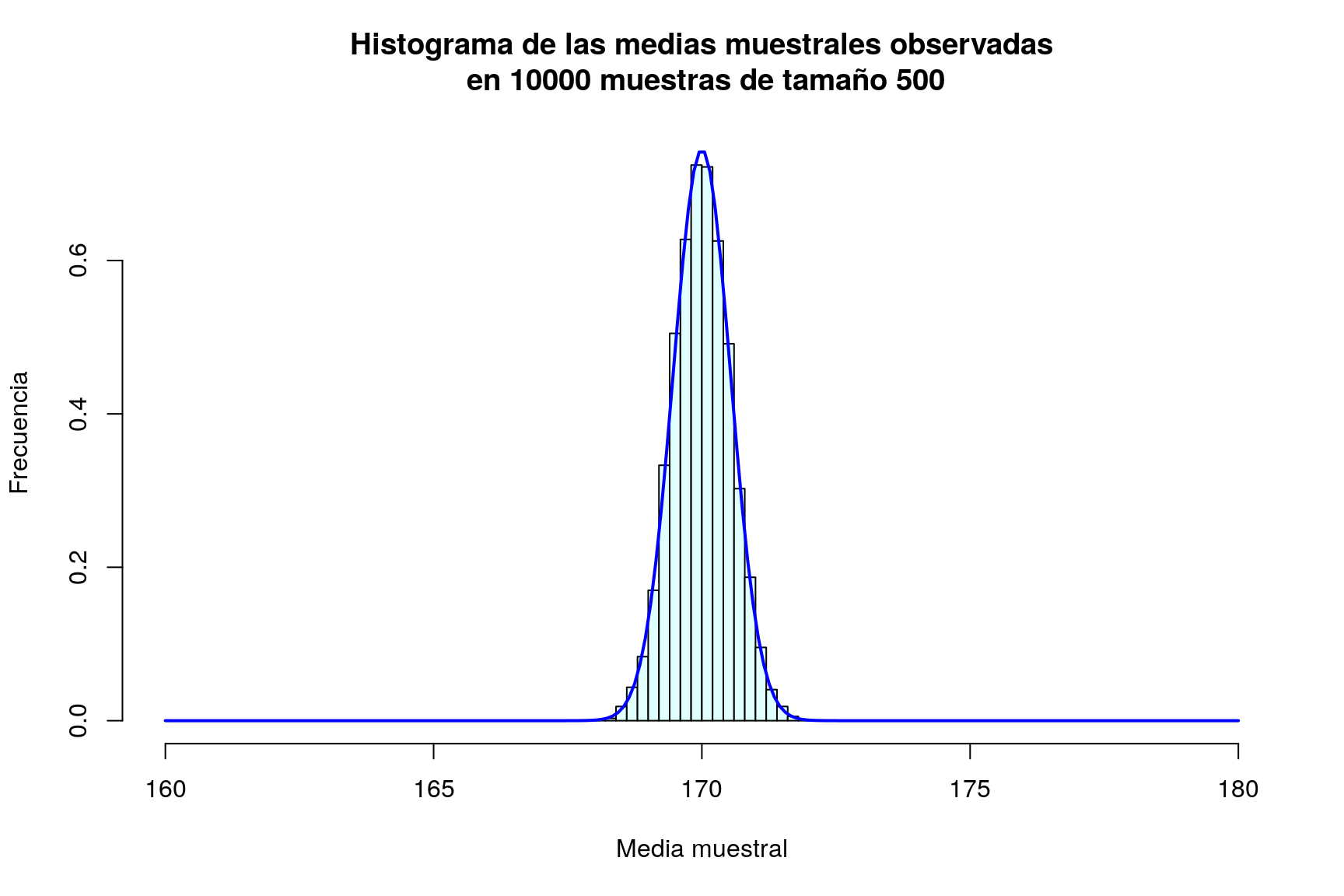
Resulta evidente en las gráficas, que el incremento del tamaño de la muestra tiene como consecuencia que las posibles medias muestrales se concentran más en torno a su media; en otras palabras, cuanto más grande sea la muestra, más probable es que la media de la muestra esté muy cerca de la media de la población. Puede comprobarse además que la desviación típica de las medias muestrales es en todos los casos un valor muy parecido a \(\frac{\sigma}{\sqrt{n}}=\frac{12}{\sqrt{n}}\):
| Tamaño de muestra (\(n\)) | Desviación típica de las medias muestrales | \(\frac{\sigma}{\sqrt{n}}\) |
|---|---|---|
| 25 | 2.40882 | 2.4 |
| 50 | 1.69587 | 1.69706 |
| 100 | 1.18425 | 1.2 |
| 1000 | 0.53569 | 0.53666 |
Tras esta serie de simulaciones, el lector debe haber quedado bastante convencido de que la media muestral de variables \(N(\mu,\sigma)\) es una variable aleatoria también normal de media \(\mu\) y desviación típica \(\frac{\sigma}{\sqrt{n}}\).
Bien, hemos visto la media de variables normales es también normal. ¿Ocurrirá lo mismo si las variables que se promedian no son normales? Podemos aquí plantear el siguiente ejercicio, para que el lector “descubra” el teorema central del límite.
- Repetir el proceso anterior cuando la variable aleatoria original es de Poisson de parámetro \(\lambda=1.3\)
- Idem cuando es exponencial de parámetro \(\mu=1.5\)
- Idem cuando es uniforme en el intervalo \(\left[5,10,\right]\)
- Idem cuando es de Weibull de parámetros 1.2 y 0.5.
Distribuciones bootstrap
La metodología bootstrap constituye una alternativa para el cálculo de errores estándar, la construcción de intervalos de confianza y de contrastes de hipótesis en aquellos casos en que hay dudas sobre la validez de los supuestos sobre la distribución de los datos (habitualmente la normalidad). Generalmente los métodos bootstrap operan del siguiente modo:
- Se remuestrea un cierto número de veces sobre el conjunto de datos disponible.
- Se calcula un estadístico particular para cada muestra.
- Se calcula la desviación típica de la distribución de ese estadístico.
Podemos observar que el proceso descrito es justo el que hemos llevado a cabo en la sección anterior, con una diferencia importante: allí teníamos la población a nuestra disposición, y podíamos sacar todas las muestras que necesitásemos de un tamaño prefijado. En en caso del bootstrap, en lugar de la población, tenemos una muestra, y de dicha muestra tenemos que sacar muchas muestras, en principio, distintas. ¿Cómo se consigue ésto?. La clave estriba realizar el remuestreo con reemplazamiento. Y R realiza muestreo con reemplazamiento de manera muy sencilla mediante la función sample() (consultar help(sample))
Volvamos al primer ejemplo que vimos en la sección anterior. Partíamos de la muestra1 de tamaño 25. Como “sabíamos” que los datos eran normales, generamos muchas muestras independientes de tamaño 25 y calculamos su desviación típica, obteniendo en aquel caso una estimación de 2.40882, muy próxima al verdadero valor de la desviación típica de la media muestral \(\frac{\sigma}{\sqrt{25}}\) = 2.4. Con una sóla muestra, si la población de procedencia es normal, se puede estimar esta desviación típica mediante el error estándar de la media (la desviación típica de la muestra dividida por \(\sqrt{n}\), en este caso, \(\frac{s}{\sqrt{n}}\)= 12.24132/5 = 2.44826.
El método bootstrap que nos permitiría obtener esta estimación sin utilizar esta fórmula (que, repetimos, sólo es válida si la población es normal). Para ello:
- Sacamos 2000 muestras de tamaño 25 de nuestra muestra original, calculando la media en cada muestra. El procedimiento es completamente análogo al que hemos utilizado antes salvo que las nuevas muestras no se generan a partir de la distribución normal (mediante
rnorm()), sino por remuestreo con reemplazamiento sobre los valores que ya tenemos:
mediaMuestraBoots=function(muestra){
nuevaMuestra=sample(muestra,25,replace=TRUE)
media=mean(nuevaMuestra)
return(media)
}
muchasMediasBoots=replicate(2000,mediaMuestraBoots(muestra1))- Calculamos la desviación típica de los valores obtenidos:
sd(muchasMediasBoots)## [1] 2.3716Representamos gráficamente el histograma de las medias bootstrap, la función de densidad aproximada por este método y la función de densidad exacta que ya vimos antes:
hist(muchasMediasBoots,col="lavender",freq=FALSE,ylim=c(0,0.2))
lines(density(muchasMediasBoots),col="red",lwd=2,lty=2)
curve(dnorm(x,mean(muestra1),sd(muchasMedias)),xlim=c(160,180),col="blue",lwd=2,add=TRUE) 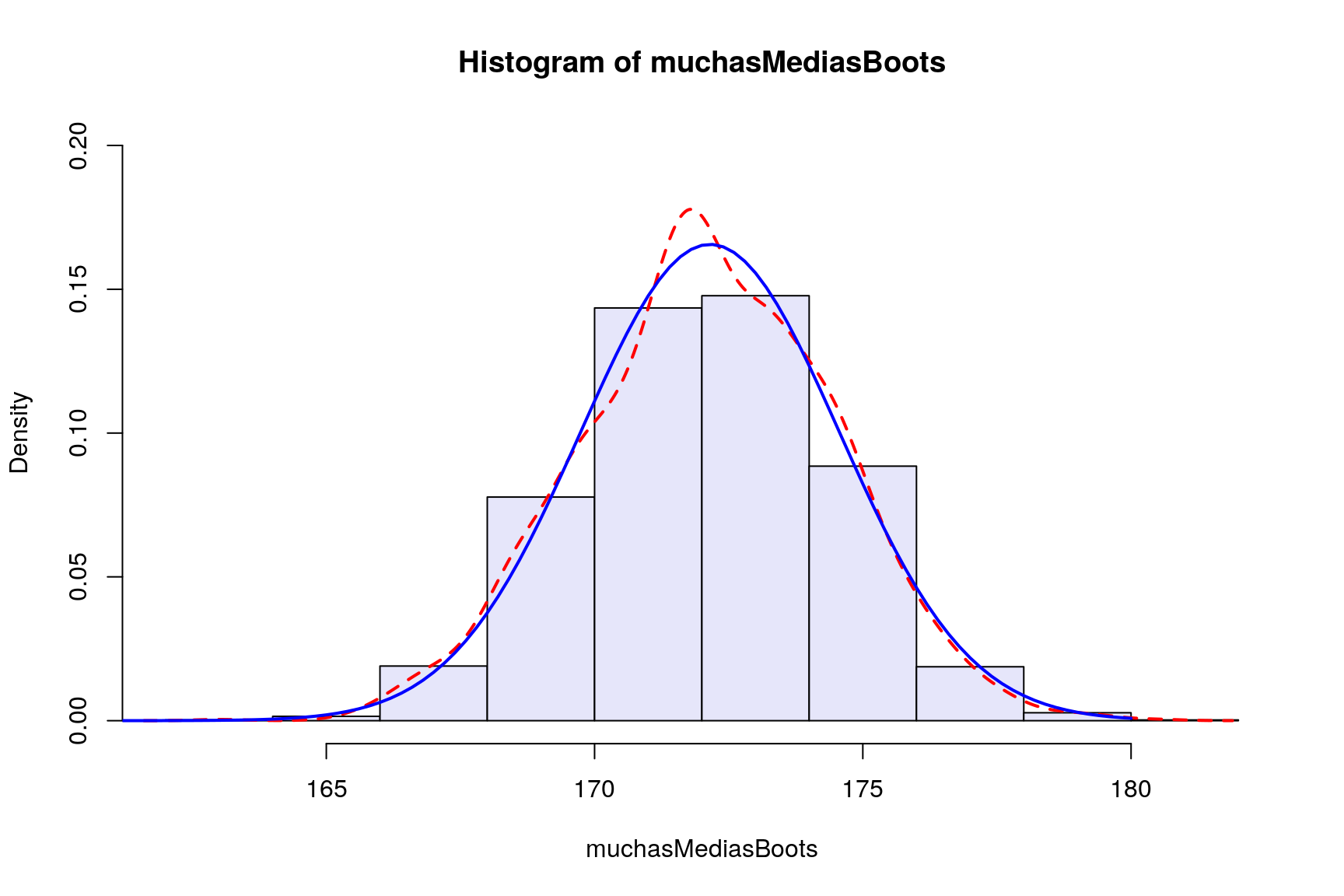
Como puede apreciarse la desviación típica que hemos obtenido mediante el método bootstrap (que no requiere la hipótesis de normalidad) es muy similar a la obtenida utilizando \(\frac{s}{\sqrt{n}}\) (que sí requiere dicha hipótesis). Por tanto, cuando no estemos seguros de que se cumpla la hipótesis de normalidad, resulta aconsejable utilizar el estimador bootstrap del error estándar.
Estimación por máxima verosimilitud
El método de máxima verosimilitud permite estimar los parámetros de la distribución de probabilidad de una variable aleatoria \(X\) a partir de la información aportada por una muestra aleatoria simple de la misma. Si \(\Theta\) es el vector de parámetros que caracteriza a dicha distribución y \(f_{\Theta}(x)\) es su función de densidad, el estimador de máxima verosimilitud dada la muestra \(\{x_1,x_2,\dots,x_n\}\) es el valor que maximiza la log verosimilitud:
\[\ell\left(\Theta\right)=\sum_{i=1}^{n}\log\left(f_{\Theta}\left(x_{i}\right)\right)\]
R cuenta con varios algoritmos eficientes para la optimización de funciones. Asimismo tiene implementadas las funciones de densidad de muchas variables aleatorias. Para llevar a cabo la estimación MV bastaría con implementar la log-verosimilitud a partir de la función de densidad y los datos disponibles, y optimizar la función así obtenida.
A modo de ejemplo, si \(X\) sigue una distribución de Weibull de parámetros \(\kappa\) y \(\eta\), la log-verosimilitud puede calcularse sencillamente como:
logver=function(params,x) {
k=params[1]
eta=params[2]
lv=sum(log(dweibull(x,k,eta)))
return(lv)
}supongamos que se dispone de la muestra:
muestra=c(1.35, 1.18, 1.98, 1.35, 0.85, 1.06, 0.77, 2.02, 1.13, 2.14)Ahora, para maximizar la log-verosimilitud con estos datos utilizamos la función optim(); a esta función es preciso pasarle una aproximación inicial de los valores de los parámetros; en este caso le pasamos (arbitrariamente) la aproximación \(\left(\kappa,\eta\right)=\left(1,1\right)\) :
optim(par=c(1,1),logver,x=muestra,control=list(fnscale=-1))Esta función nos devuelve los siguientes valores, que constituyen los estimadores MV de \(\kappa\) y \(\eta\):
## [1] 3.2161 1.5486br>
El paquete MASS incluye la función fitdistr() que realiza la estimación MV de los parámetros de diversas distribuciones sin que sea necesario suministrar valores iniciales. Calcula además el error estándar de dichos estimadores. Consultar help(fitdistr) para ver la lista de distribuciones incluidas. Para estimar los parámetros de la distribución de Weibull con los datos anteriores, procederíamos del siguiente modo:
library(MASS)
ajuste=fitdistr(muestra,"weibull")
ajuste## shape scale
## 3.21639 1.54850
## (0.79006) (0.16136)La función confint() proporciona además intervalos de confianza para los estimadores:
confint(ajuste)## 2.5 % 97.5 %
## shape 1.6679 4.7649
## scale 1.2322 1.8648Más información
G. Jay Kerns (Youngstown State University, Ohio) ha creado dos paquetes de R,
prob,IPSURque corresponden a dos libros de probabilidad y estadística de este autor (los libros pueden descargarse libremente aquí y aquí), y los paquetes se pueden descargar directamente de CRAN. Ambos libros se pueden utilizar para un curso de probabilidad y estadística de primero de carrera. Los paquetes citados, además de incluir versiones pdf de ambos libros, incluyen datos, funciones y ejemplos de utilización de R para el aprendizaje del cálculo de probabilidades y la estadística.http://www.r-tutor.com/elementary-statistics/probability-distributions
© 2016 Angelo Santana, Carmen N. Hernández, Departamento de Matemáticas ULPGC