Objetos en R: Data Frames y Listas
Angelo Santana & Carmen Nieves Hernández,
Departamento de Matemáticas, ULPGC
Junio 2014
Data.frames
El término “dataframe” es difícil de traducir al castellano. Podría traducirse como Hoja de datos o Marco de datos. Los dataframes son una clase de objetos especial en R. Normalmente, cuando se realiza un estudio estadístico sobre los sujetos u objetos de una muestra, la información se organiza precisamente en un dataframe: una hoja de datos, en los que cada fila corresponde a un sujeto y cada columna a una variable. La estructura de un data.frame es muy similar a la de una matriz. La diferencia es que una matriz sólo admite valores numéricos, mientras que en un dataframe podemos incluir también datos alfanuméricos.
Creación de un dataframe
El siguiente ejemplo nos muestra como crear un data.frame a partir de los datos recogidos sobre una muestra de 10 personas, para cada una de las cuales se ha registrado su edad, sexo y tiempo en minutos que estuvo hablando por teléfono el día antes de la encuesta:
edad <- c(22, 34, 29, 25, 30, 33, 31, 27, 25, 25)
tiempo <- c(14.21, 10.36, 11.89, 13.81, 12.03, 10.99, 12.48, 13.37, 12.29, 11.92)
sexo <- c("M","H","H","M","M","H","M","M","H","H")
misDatos <- data.frame(edad,tiempo,sexo)
misDatos## edad tiempo sexo
## 1 22 14.21 M
## 2 34 10.36 H
## 3 29 11.89 H
## 4 25 13.81 M
## 5 30 12.03 M
## 6 33 10.99 H
## 7 31 12.48 M
## 8 27 13.37 M
## 9 25 12.29 H
## 10 25 11.92 Hstr(misDatos) # Estructura de 'misDatos'## 'data.frame': 10 obs. of 3 variables:
## $ edad : num 22 34 29 25 30 33 31 27 25 25
## $ tiempo: num 14.2 10.4 11.9 13.8 12 ...
## $ sexo : Factor w/ 2 levels "H","M": 2 1 1 2 2 1 2 2 1 1names(misDatos) # Nombre de las variables contenidas en 'misDatos'## [1] "edad" "tiempo" "sexo"En este ejemplo hemos creado un data.frame llamado misDatos que contiene a las tres variables edad, tiempo y sexo. La función str() nos muestra la estructura de este objeto, confirmándonos que es un data.frame de tres variables con 10 observaciones cada una. Nos informa además de que las dos primeras variables son numéricas y la tercera, el sexo, es un factor con dos valores, “H” y “M”. La función names() por su parte, nos devuelve los nombres de las variables contenidas en misDatos.
Cuando desde R se leen datos situados en un fichero externo (un fichero de texto, una hoja excel, un archivo de datos de SPSS,…), estos datos se importan en un dataframe.
Acceso a variables dentro de un dataframe
El acceso a los datos que se encuentran en un data.frame es muy similar al acceso a los datos de una matriz que ya vimos en la sección anterior. Sin embargo, para los data.frames R dispone de algunas funciones que facilitan la tarea de seleccionar o filtrar datos. Así por ejemplo, si queremos ver sólo los datos de los sujetos (filas) 3 a 6, escribiríamos:
misDatos[3:6,]## edad tiempo sexo
## 3 29 11.89 H
## 4 25 13.81 M
## 5 30 12.03 M
## 6 33 10.99 HSi queremos seleccionar los datos de edad (primera columna), podemos tratar a misDatos igual que si fuese una matriz:
misDatos[,1]## [1] 22 34 29 25 30 33 31 27 25 25Aunque también podemos referirnos a la columna por su nombre:
misDatos$edad## [1] 22 34 29 25 30 33 31 27 25 25Nótese que en este caso hemos de utilizar el nombre del data.frame (misDatos) seguido del símbolo $ y del nombre de la variable que nos interesa (edad). De manera equivalente, la selección de esa variable puede realizarse mediante:
misDatos[,"edad"]## [1] 22 34 29 25 30 33 31 27 25 25o poniendo el nombre de la variable entre dobles corchetes y entre comillas:
misDatos[["edad"]]## [1] 22 34 29 25 30 33 31 27 25 25Así pues, los siguientes comandos son equivalentes y dan el mismo resultado:
mean(misDatos[,1])
mean(misDatos$edad)
mean(misDatos[,"edad"])
mean(misDatos[["edad"]])## [1] 28.1Los comandos attach y detach
El acceso a las variables dentro de un dataframe puede hacerse engorroso cuando hemos de escribir constantemente el nombre del dataframe (en particular si éste es muy largo). Imaginemos, por ejemplo, que para el conjunto misDatos deseamos construir tablas de frecuencias de cada una de las variables que contiene, una tabla de frecuencias cruzadas para el nivel de estudios por sexo, y que además queremos calcular la edad medio de los individuos de cada sexo. La sintaxis a utilizar sería la siguiente:
table(misDatos$estudios)
table(misDatos$sexo)
table(misDatos$edad)
table(misDatos$sexo,misDatos$edad)
mean(misDatos$edad[misDatos$sexo=="M"])
mean(misDatos$edad[misDatos$sexo=="H"])Obviamente, escribir tantas veces misDatos resulta tedioso, al margen de que se multiplica el riesgo de cometer errores en la redacción de los comandos. Para evitar este problema podemos utilizar el comando attach(), cuyo objetivo consiste básicamente en “enganchar” el contenido del dataframe al entorno donde R busca los nombres de variable; de esta forma se puede acceder directamente a las variables del dataframe por su nombre, sin necesidad de que éste tenga que ser precedido con el nombre del dataframe y el símbolo $; una vez que hayamos acabado nuestra tarea “desenganchamos” el dataframe con detach(). La tarea anterior, utilizando estos comandos, se puede llevar a cabo mediante:
attach(misDatos)
table(estudios)
table(sexo)
table(edad)
table(sexo,edad)
mean(edad[sexo=="M"])
mean(edad[sexo=="H"])
detach(misDatos)lo cual es notablemente más simple. No obstante hay que ser extremadamente precavido al utilizar estos comandos ya que cuando se utilizan varios dataframes simultáneamente es muy fácil hacer attachs y detachs en lugares incorrectos, lo que puede conducir a mezclar datos de distinta procedencia y cometer errores inadvertidos. Obsérvese el siguiente ejemplo:
# Creamos el dataframe 'divisas' con la tasa de cambio de algunas monedas
divisas = data.frame(moneda=c("Libra", "Euro", "Rublo"), cambio=c(1.2, 1, 0.02))
divisas## moneda cambio
## 1 Libra 1.20
## 2 Euro 1.00
## 3 Rublo 0.02# Creamos un dataframe con el nombre de algunos paises y su moneda nacional
paises = data.frame(pais=c("EEUU", "Venezuela", "Japón"), moneda=c("Dólar", "Bolívar", "Yen"))
paises## pais moneda
## 1 EEUU Dólar
## 2 Venezuela Bolívar
## 3 Japón Yenattach(divisas) # Enganchamos 'divisas' al entorno de búsqueda de nombres de variables.
moneda # Se muestra la variable 'moneda' del dataframe 'divisas'## [1] Libra Euro Rublo
## Levels: Euro Libra Rubloattach(paises) # ¡CUIDADO! no se ha hecho el 'detach' del datafame 'divisas'## The following object is masked from divisas:
##
## monedamoneda # 'moneda' se lee del último dataframe "enganchado", que es 'paises'## [1] Dólar Bolívar Yen
## Levels: Bolívar Dólar Yencambio # ¡Pero esta variable se sigue leyendo del dataframe 'divisas'!## [1] 1.20 1.00 0.02# Si, por ejemplo, pegamos los valores de ambas variables pensando que corresponden al mismo dataframe estaríamos metiendo la pata.
paste(moneda,cambio, sep=": ") ## [1] "Dólar: 1.2" "Bolívar: 1" "Yen: 0.02"detach(paises) # Desenganchamos el dataframe 'paises'
moneda # Recuperamos la variable 'moneda' que estaba en 'divisas'## [1] Libra Euro Rublo
## Levels: Euro Libra Rublodetach(divisas) # Desenganchamos 'divisas'
moneda # Ya no hay ninguna variable 'moneda' a la que R pueda acceder directamente por su nombre## Error in eval(expr, envir, enclos): objeto 'moneda' no encontradoPor otra parte, si ya existe una variable en el entorno de trabajo con el mismo nombre que alguna variable del dataframe que se engancha mediante el attach(), la variable del dataframe no resulta accesible ya que queda enmascarada por la variable existente previamente.
El siguiente ejemplo ilustra esta situación: creamos una variable llamada longitud, con 6 valores, y a continuación un dataframe llamado medidas que contiene tres variables con tres valores cada una; en el dataframe una de las variables también se llama longitud:
longitud=c(12,10,11,13,14,17)
medidas=data.frame(longitud=c(6,4,7), peso=c(240,326,315), diametro=c(8,9,9))Calculamos el valor medio de estas cuatro variables:
mean(longitud)## [1] 12.8333mean(medidas$longitud)## [1] 5.66667mean(medidas$peso)## [1] 293.667mean(medidas$diametro)## [1] 8.66667Efectuamos el attach del dataframe medidas para acceder a sus variables y calculamos de nuevo la media de cada una:
attach(medidas)## The following objects are masked _by_ .GlobalEnv:
##
## longitud, pesomean(peso)## [1] NAmean(diametro)## [1] 8.66667mean(longitud)## [1] 12.8333Obsérvese de que R nos advierte de que el objeto longitud ha quedado enmascarado. Vemos que, si bien R ha calculado los valores medios del diámetro y peso correspondientes a las variables del dataframe medidas, la longitud sigue siendo la de la variable longitud previa a hacer el attach.
El comando with
El comando with permite ejecutar una o varias instrucciones sobre las variables de un dataframe accediendo a ellas solamente por su nombre, sin necesidad de utilizar attach. Resulta particularmente útil para realizar cálculos con las variables dentro de un dataframe.
Si, por ejemplo, queremos calcular la densidad de los objetos cuyas medidas figuran en el dataframe anterior, podemos utilizar la siguiente sintaxis
with(medidas,{
volumen=longitud*pi*(diametro/2)^2 # Calcula el volumen de los objetos
densidad=peso/volumen # Calcula su densidad
densidad # Muestra los valores de densidad
})## [1] 0.795775 1.281099 0.707355Obsérvese que el with no modifica el contenido del dataframe medidas:
medidas## longitud peso diametro
## 1 6 240 8
## 2 4 326 9
## 3 7 315 9Si quisiéramos incluir la densidad dentro del dataframe medidas deberíamos proceder del siguiente modo:
medidas$densidad=with(medidas,{
volumen=longitud*pi*(diametro/2)^2 # Calcula el volumen de los objetos
densidad=peso/volumen # Calcula su densidad
densidad # Muestra los valores de densidad
})
medidas # Mostramos el dataframe 'medidas'. Ahora sí que contiene la densidad## longitud peso diametro densidad
## 1 6 240 8 0.795775
## 2 4 326 9 1.281099
## 3 7 315 9 0.707355Subconjuntos de un dataframe
La función subset() nos permite seleccionar una parte del data.frame. Por ejemplo, si deseamos crear dos dataframes nuevos, uno solo con los hombres y otro con las mujeres utilizaríamos:
hombres=subset(misDatos,sexo=="H")
hombres## edad tiempo sexo
## 2 34 10.36 H
## 3 29 11.89 H
## 6 33 10.99 H
## 9 25 12.29 H
## 10 25 11.92 Hmujeres=subset(misDatos,sexo=="M")
mujeres## edad tiempo sexo
## 1 22 14.21 M
## 4 25 13.81 M
## 5 30 12.03 M
## 7 31 12.48 M
## 8 27 13.37 MPodemos elaborar selecciones más complejas; por ejemplo:
• Sujetos que sean hombres y tengan más de 30 años (la condición “ y ” se especifica mediante el símbolo “ & ”):
mayores=subset(misDatos,sexo=="H" & edad>30)
mayores## edad tiempo sexo
## 2 34 10.36 H
## 6 33 10.99 H• Hombres que tengan menos de 30 años y hayan hablado por el móvil más de 12 minutos diarios:
jov_habladores=subset(misDatos,sexo=="H" & edad<30 & tiempo>12)
jov_habladores## edad tiempo sexo
## 9 25 12.29 H• Sujetos que tengan menos de 25 o más 30 años (la condición “ o ” se expresa mediante la línea vertical “ | ”):
extremos=subset(misDatos,edad<25|edad>30)
extremos## edad tiempo sexo
## 1 22 14.21 M
## 2 34 10.36 H
## 6 33 10.99 H
## 7 31 12.48 MPodemos seleccionar además un subconjunto de variables del data.frame. Por ejemplo, si nos interesan solo la edad y el tiempo de uso del móvil de los hombres de la muestra:
hombres=subset(misDatos,sexo=="H", select=c(edad, tiempo))
hombres## edad tiempo
## 2 34 10.36
## 3 29 11.89
## 6 33 10.99
## 9 25 12.29
## 10 25 11.92Construir el subconjunto de las mujeres con edad mayor a 25 y menor a 50 años. Para este subconjunto calcular el tiempo medio de uso del móvil.
Combinación de dataframes: rbind y merge
Si tenemos dos dataframes con la misma estructura (idénticas variables), pero distintos datos, podemos combinarlos pegando uno a continuación del otro mediante rbind (acrónimo de rowbind, pegar por filas):
animales1 = data.frame(animal=c("vaca","perro","rana","lagarto","mosca","jilguero"),
clase=c("mamífero","mamífero","anfibio","reptil","insecto","ave"))
animales1## animal clase
## 1 vaca mamífero
## 2 perro mamífero
## 3 rana anfibio
## 4 lagarto reptil
## 5 mosca insecto
## 6 jilguero aveanimales2 = data.frame(animal=c("atún", "cocodrilo", "gato","rana"), clase=c("pez", "reptil", "mamífero","anfibio"))
animales2## animal clase
## 1 atún pez
## 2 cocodrilo reptil
## 3 gato mamífero
## 4 rana anfibioanimales3 = rbind(animales1,animales2)
animales3## animal clase
## 1 vaca mamífero
## 2 perro mamífero
## 3 rana anfibio
## 4 lagarto reptil
## 5 mosca insecto
## 6 jilguero ave
## 7 atún pez
## 8 cocodrilo reptil
## 9 gato mamífero
## 10 rana anfibioEl comando rbind no controla la posible aparición de casos repetidos en los dos dataframes (podemos comprobar que la rana está repetida en el dataframe ‘animales3’). La función merge() evita este problema; utilizando la opción all=TRUE ó all=FALSE (valor por defecto) se consigue que se muestren todos los datos de ambos dataframes, o solo aquellos que son comunes a ambos
animales4=merge(animales1,animales2)
animales4## animal clase
## 1 rana anfibioanimales5=merge(animales1,animales2,all=TRUE)
animales5## animal clase
## 1 jilguero ave
## 2 lagarto reptil
## 3 mosca insecto
## 4 perro mamífero
## 5 rana anfibio
## 6 vaca mamífero
## 7 atún pez
## 8 cocodrilo reptil
## 9 gato mamíferoSi los dataframes tienen estructura distinta, pero contienen variables en común que permiten identificar unívocamente a los mismos objetos en ambos conjuntos, también podemos combinarlos mediante merge():
superficieAnimales=data.frame(animal=c("perro","tortuga","jilguero",
"cocodrilo","vaca","lagarto","sardina"),
superficie=c("pelo","placas óseas","plumas",
"escamas","pelo","escamas","escamas"))
superficieAnimales## animal superficie
## 1 perro pelo
## 2 tortuga placas óseas
## 3 jilguero plumas
## 4 cocodrilo escamas
## 5 vaca pelo
## 6 lagarto escamas
## 7 sardina escamasmerge(animales3,superficieAnimales) # Muestra sólo los animales comunes a ambos dataframes## animal clase superficie
## 1 cocodrilo reptil escamas
## 2 jilguero ave plumas
## 3 lagarto reptil escamas
## 4 perro mamífero pelo
## 5 vaca mamífero pelomerge(animales3,superficieAnimales, all.x=TRUE) # Muestra todos los animales del primer dataframe.## animal clase superficie
## 1 jilguero ave plumas
## 2 lagarto reptil escamas
## 3 mosca insecto <NA>
## 4 perro mamífero pelo
## 5 rana anfibio <NA>
## 6 rana anfibio <NA>
## 7 vaca mamífero pelo
## 8 atún pez <NA>
## 9 cocodrilo reptil escamas
## 10 gato mamífero <NA>merge(animales3,superficieAnimales, all.y=TRUE) # Muestra todos los animales del segundo dataframe.## animal clase superficie
## 1 jilguero ave plumas
## 2 lagarto reptil escamas
## 3 perro mamífero pelo
## 4 vaca mamífero pelo
## 5 cocodrilo reptil escamas
## 6 sardina <NA> escamas
## 7 tortuga <NA> placas óseasmerge(animales3,superficieAnimales, all=TRUE) # Muestra todos los animales de ambos dataframes.## animal clase superficie
## 1 jilguero ave plumas
## 2 lagarto reptil escamas
## 3 mosca insecto <NA>
## 4 perro mamífero pelo
## 5 rana anfibio <NA>
## 6 rana anfibio <NA>
## 7 vaca mamífero pelo
## 8 atún pez <NA>
## 9 cocodrilo reptil escamas
## 10 gato mamífero <NA>
## 11 sardina <NA> escamas
## 12 tortuga <NA> placas óseasComo vemos, cuando en la combinación de dataframes faltan datos, se rellenan los huecos con valores perdidos (NA)
Ordenación de dataframes
Para ordenar un dataframe hemos de aplicar la función order() al elemento o elementos por el que queramos ordenar, y utilizar el resultado de esta función como índice del dataframe.
Por ejemplo, si queremos ordenar el dataframe animales1 por orden alfabético de animales, haríamos:
ordenacion=order(animales1$animal)
ordenacion # Posiciones dentro del dataframe 'animales1' de los animales ordenados alfabéticamente## [1] 6 4 5 2 3 1animales1=animales1[ordenacion,] # Se reordenan las filas del dataframe animales1
animales1## animal clase
## 6 jilguero ave
## 4 lagarto reptil
## 5 mosca insecto
## 2 perro mamífero
## 3 rana anfibio
## 1 vaca mamíferode modo equivalente, en una línea de código:
animales1=animales1[order(animales1$animal),] Si queremos ordenar nuestro primer dataframe (misDatos) primero por edad y luego por tiempo utilizando el móvil:
misDatos=misDatos[order(misDatos$edad,misDatos$tiempo),]
misDatos## edad tiempo sexo
## 1 22 14.21 M
## 10 25 11.92 H
## 9 25 12.29 H
## 4 25 13.81 M
## 8 27 13.37 M
## 3 29 11.89 H
## 5 30 12.03 M
## 7 31 12.48 M
## 6 33 10.99 H
## 2 34 10.36 HLa función reshape(): cambiando la forma de un dataframe.
En ocasiones es necesario cambiar la “forma” en que los datos se encuentran dispuestos dentro de un dataframe. A modo de ejemplo, el siguiente archivo contiene los datos de temperatura en el aeropuerto de Gran Canaria medidos una vez cada hora desde el 1 de enero de 2014 (nótese que el archivo se descarga de una dirección http)
tempe=read.csv2("http://www.dma.ulpgc.es/profesores/personal/stat/cursoR4ULPGC/datos/temperatura2014.csv")
head(tempe)## mes dia temp0 temp1 temp2 temp3 temp4 temp5 temp6 temp7 temp8 temp9 temp10
## 1 1 1 16 16 16 16 15 15 15 15 15 17 19
## 2 1 2 15 15 15 14 14 14 14 14 15 18 18
## 3 1 3 16 16 16 15 15 15 15 16 16 18 19
## 4 1 4 17 17 17 17 17 18 18 18 18 20 21
## 5 1 5 19 19 19 18 18 18 17 17 17 18 20
## 6 1 6 18 17 17 16 15 15 17 17 17 18 19
## temp11 temp12 temp13 temp14 temp15 temp16 temp17 temp18 temp19 temp20 temp21
## 1 19 20 19 19 19 19 18 18 17 16 16
## 2 19 19 20 19 19 19 19 18 17 16 16
## 3 20 20 21 21 20 20 20 18 18 18 17
## 4 21 22 21 21 21 21 20 20 20 20 20
## 5 20 20 20 20 20 20 19 18 18 17 18
## 6 19 19 19 19 19 19 19 18 18 17 17
## temp22 temp23
## 1 15 16
## 2 16 16
## 3 18 18
## 4 20 19
## 5 18 18
## 6 17 17Como podemos ver, cada fila de este dataframe corresponde a un día del año; las dos primeras columnas especifican dia y mes concretos; las restantes columnas, llamadas temp0 hasta temp23 registran la temperatura medida en el aeropuerto cada hora en punto, desde las 0 a las 23 horas. En este dataframe, la variable temperatura (temp) se encuentra en formato wide (ancho).
Para convertir este dataframe en otro que tenga una única variable temperatura medida 24 veces cada día, esto es, en formato long (largo), utilizaríamos la función reshape() del siguiente modo:
temperatura=reshape(tempe, idvar = c("mes","dia"), varying = list(3:26),
v.names="temp",direction = "long", timevar="hora")
head(temperatura,10)## mes dia hora temp
## 1.1.1 1 1 1 16
## 1.2.1 1 2 1 15
## 1.3.1 1 3 1 16
## 1.4.1 1 4 1 17
## 1.5.1 1 5 1 19
## 1.6.1 1 6 1 18
## 1.7.1 1 7 1 16
## 1.8.1 1 8 1 16
## 1.9.1 1 9 1 19
## 1.10.1 1 10 1 14Aquí:
idvares la variable o variables (en este casomesydia) que se importan del archivo original y que no corresponden a una variable que se encuentra desglosada en varias columnas.varyinges una lista donde se especifican las posiciones de las columnas que corresponden a la variable que se quiere convertir de formatowidea formatolong.v.namespermite especificar el nombre que recibirá la variable que se encuentra en formatowideuna vez que se haya reescrito en formatolong(o al revés)directionespecifica la dirección en que se va a reformatear el dataframe (long, como en este caso, owide)timevarnombre de la variable que codifica el índice (número de orden) de cada observación. Normalmente corresponde a la secuencia temporal en que se han realizado las observaciones.
Como vemos, como resultado del reshape ahora tenemos una única variable temperatura y se ha creado la variable hora; por defecto, el dataframe está ordenado por esta última variable. Si queremos ordenarlo primero por mes, luego por día y por último por hora, utilizamos la función order():
ord=with(temperatura, order(mes,dia,hora))
temperatura=temperatura[ord,]
head(temperatura,10)## mes dia hora temp
## 1.1.1 1 1 1 16
## 1.1.2 1 1 2 16
## 1.1.3 1 1 3 16
## 1.1.4 1 1 4 16
## 1.1.5 1 1 5 15
## 1.1.6 1 1 6 15
## 1.1.7 1 1 7 15
## 1.1.8 1 1 8 15
## 1.1.9 1 1 9 15
## 1.1.10 1 1 10 17Además, la variable hora no corresponde, en realidad a la hora, sino al orden de cada observación dentro de cada día; si tenemos en cuenta que la primera observación se realiza a las cero horas, la segunda a la una, la tercera a las dos, etc, podemos convertir esta variable en la hora real de la observación simplemente restándole una unidad:
temperatura$hora=temperatura$hora-1
head(temperatura,10)## mes dia hora temp
## 1.1.1 1 1 0 16
## 1.1.2 1 1 1 16
## 1.1.3 1 1 2 16
## 1.1.4 1 1 3 16
## 1.1.5 1 1 4 15
## 1.1.6 1 1 5 15
## 1.1.7 1 1 6 15
## 1.1.8 1 1 7 15
## 1.1.9 1 1 8 15
## 1.1.10 1 1 9 17Representamos a continuación gráficamente la secuencia de observaciones de temperatura a lo largo del periodo observado (1 de enero a 11 de julio). Para ello creamos en primer lugar la variable fecha (ver la sección Fechas de este curso).
fecha=with(temperatura,paste(paste("2014",mes,dia,sep="-"),hora,sep=" "))
temperatura$fecha=as.POSIXlt(fecha,format="%Y-%m-%d %H")
with(temperatura,plot(fecha,temp,type="l",ylab="Temperatura (ºC)",main="Temperaturas aeropuerto GC 2014"))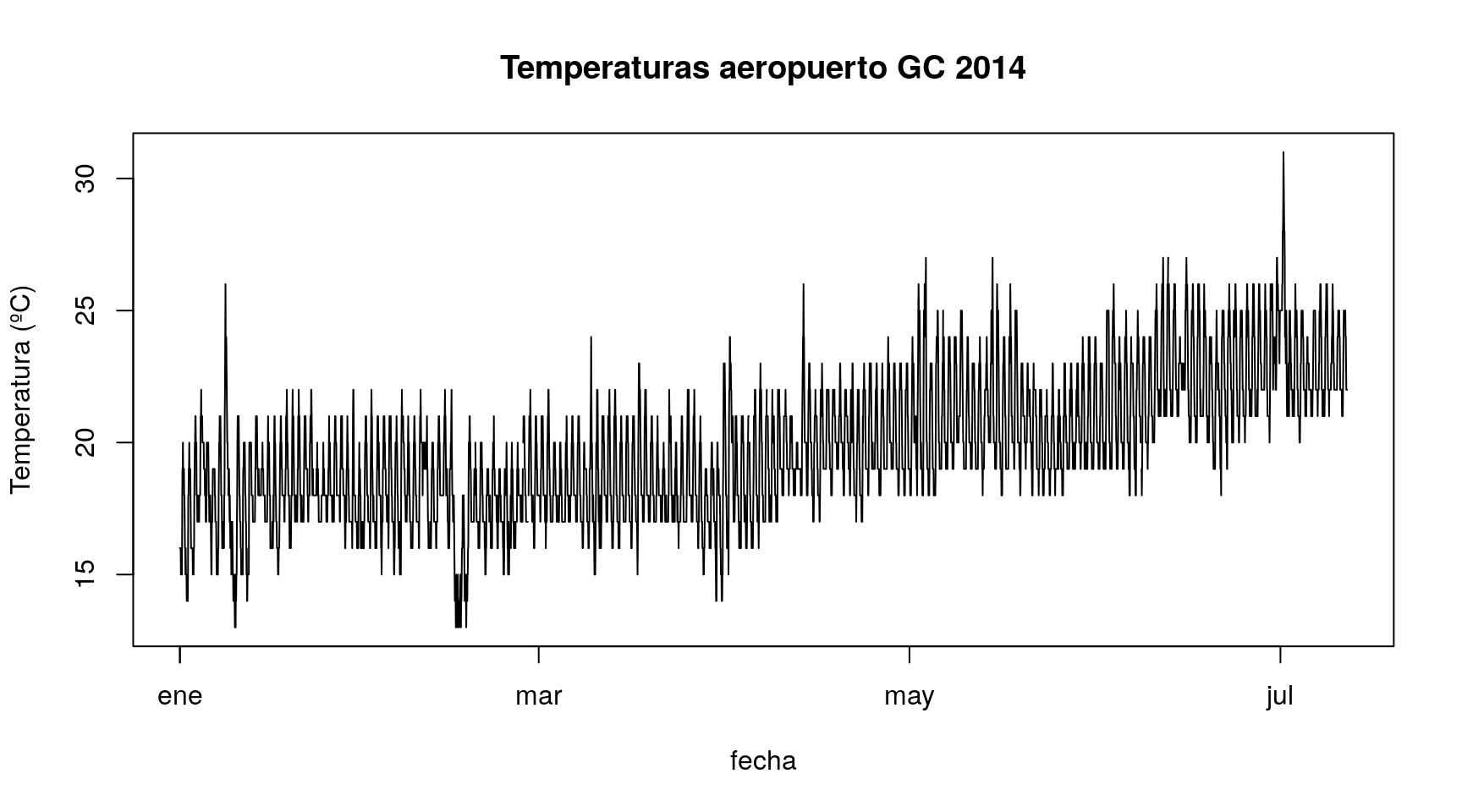
Listas
La lista representa el siguiente nivel de complejidad en las estructuras de datos que R es capaz de manejar. Podemos entender una lista como un contenedor de objetos que pueden ser de cualquier clase: números, vectores, matrices, funciones, data.frames, incluso otras listas. Una lista puede contener a la vez varios de estos objetos, que pueden ser además de distintas dimensiones.
Por ejemplo, podemos crear una lista que contenga el data.frame misDatos, la matriz A, la matrizM, el vector x=c(1,2,3,4) y la constante e=exp(1):
A=matrix(1:9,nrow=3)
M=matrix(1,4,nrow=2)
MiLista <- list(misDatos,A,M=M,x=c(1,2,3,4),e=exp(1))
MiLista## [[1]]
## edad tiempo sexo
## 1 22 14.21 M
## 10 25 11.92 H
## 9 25 12.29 H
## 4 25 13.81 M
## 8 27 13.37 M
## 3 29 11.89 H
## 5 30 12.03 M
## 7 31 12.48 M
## 6 33 10.99 H
## 2 34 10.36 H
##
## [[2]]
## [,1] [,2] [,3]
## [1,] 1 4 7
## [2,] 2 5 8
## [3,] 3 6 9
##
## $M
## [,1] [,2] [,3] [,4]
## [1,] 1 1 1 1
## [2,] 1 1 1 1
##
## $x
## [1] 1 2 3 4
##
## $e
## [1] 2.71828
Obsérvese a continuación como podemos acceder a los distintos elementos de la lista. Póngase especial atención a lo que ocurre con los elementos misDatos y A, cuyo nombre no se utilizó explícitamente en la declaración de la lista:
MiLista$misDatos## NULLMiLista[[1]]## edad tiempo sexo
## 1 22 14.21 M
## 10 25 11.92 H
## 9 25 12.29 H
## 4 25 13.81 M
## 8 27 13.37 M
## 3 29 11.89 H
## 5 30 12.03 M
## 7 31 12.48 M
## 6 33 10.99 H
## 2 34 10.36 HMiLista$A## NULLMiLista[[2]]## [,1] [,2] [,3]
## [1,] 1 4 7
## [2,] 2 5 8
## [3,] 3 6 9MiLista$M## [,1] [,2] [,3] [,4]
## [1,] 1 1 1 1
## [2,] 1 1 1 1MiLista$x## [1] 1 2 3 4MiLista$e## [1] 2.71828Como vemos, para acceder a los objetos que forman parte de una lista, basta con añadir su nombre a continuación del de la lista, separados por el símbolo $, o bien con el índice de posición dentro de la lista con doble corchete [[]]. Nótese que los objetos misDatos y A no tienen nombre dentro de la lista, por lo que hemos de referirnos a ellos como MiLista[[1]] o MiLista[[2]]. Sin embargo, el objeto M sí que tiene nombre. Para que un objeto dentro de una lista tenga nombre, éste debe declararse explícitamente en la construcción de la lista, tal como se hizo con M, x o e.
R utiliza las listas, sobre, todo como salida de los distintos procedimientos estadísticos. Así, por ejemplo, al realizar un contraste de medias de dos poblaciones, R calcula, entre otras cosas, la diferencia de medias muestrales, el valor del estadístico de contraste, el p-valor del test y el intervalo de confianza para la diferencia observada. Todos estos términos forman parte de una lista. La sintaxis para comparar, por ejemplo, el tiempo medio de uso del móvil entre hombres y mujeres a partir de nuestros datos sería:
t.test(tiempo~sexo, data=misDatos)##
## Welch Two Sample t-test
##
## data: tiempo by sexo
## t = -3.133, df = 7.854, p-value = 0.0143
## alternative hypothesis: true difference in means is not equal to 0
## 95 percent confidence interval:
## -2.937818 -0.442182
## sample estimates:
## mean in group H mean in group M
## 11.49 13.18Si guardamos el resultado de este contraste en el objeto contraste, podemos observar que tiene estructura de lista:
contraste=t.test(tiempo~sexo, data=misDatos)
str(contraste)## List of 9
## $ statistic : Named num -3.13
## ..- attr(*, "names")= chr "t"
## $ parameter : Named num 7.85
## ..- attr(*, "names")= chr "df"
## $ p.value : num 0.0143
## $ conf.int : num [1:2] -2.938 -0.442
## ..- attr(*, "conf.level")= num 0.95
## $ estimate : Named num [1:2] 11.5 13.2
## ..- attr(*, "names")= chr [1:2] "mean in group H" "mean in group M"
## $ null.value : Named num 0
## ..- attr(*, "names")= chr "difference in means"
## $ alternative: chr "two.sided"
## $ method : chr "Welch Two Sample t-test"
## $ data.name : chr "tiempo by sexo"
## - attr(*, "class")= chr "htest"Si deseamos extraer, por ejemplo, solo el intervalo de confianza de la lista anterior nos bastaría con ejecutar:
contraste$conf.int## [1] -2.937818 -0.442182
## attr(,"conf.level")
## [1] 0.95También podemos extraerlo directamente en la llamada al procedimiento t.test():
t.test(tiempo~sexo, data=misDatos)$conf.int## [1] -2.937818 -0.442182
## attr(,"conf.level")
## [1] 0.95
© 2016 Angelo Santana, Carmen N. Hernández, Departamento de Matemáticas ULPGC