Tarea 2: Importación de bases de datos
Importación de bases de datos
Bases de datos.
El primer paso para la realización de cualquier análisis estadístico es la elaboración de la base de datos que contenga la información que hemos de analizar. Para construir una base de datos existen muchas herramientas alternativas. Sin duda una de las más sencillas de utilizar (siempre y cuando la base de datos no sea excesivamente grande) es una simple hoja de cálculo, como las que ofrecen Microsoft Excel o LibreOfice Calc.
Las reglas para construir una hoja de cálculo que pueda servir para un análisis estadístico son muy simples:
Cada fila debe ser un caso (un paciente o sujeto experimental)
Cada columna debe ser una variable.
Cada columna debe estar encabezada con el nombre de la variable que represente.
Las casillas con valores perdidos deben dejarse en blanco, o al menos utilizar un código estándar que luego pueda interpretarse como tal sin posibilidad de confusion.
Debe evitarse dejar filas vacías, columnas vacías, insertar figuras o introducir decoraciones de cualquier tipo.
Hay que vigilar con especial cuidado en la introduccion de valores numéricos no mezclar puntos (.) con comas (,). Si en una misma columna escribimos el número 2,3 y más adelante 7.2, R interpretará todos los valores de la variable como caracteres no como números.
De la misma forma, en una columna donde se registran valores numéricos no deben introducirse caracteres, pues en tal caso R interpretará la variable como de tipo carácter y no como numérica.
Importacion de datos desde Excel:
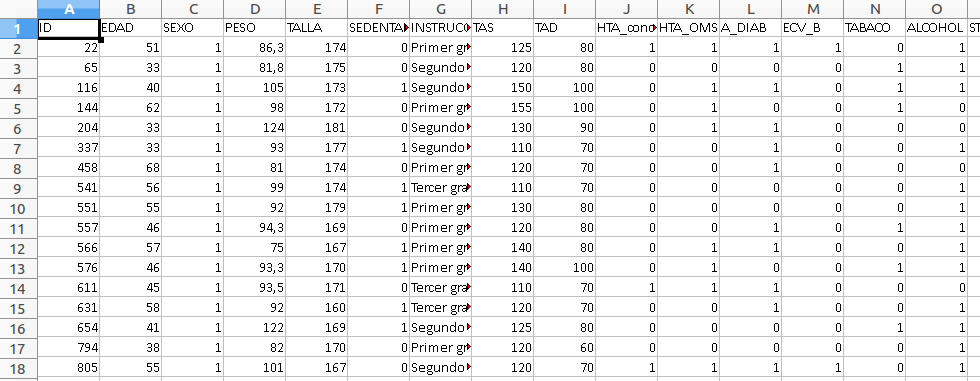
A modo de ejemplo, en el campus virtual de la asignatura, o pinchando aquí podemos descargar la hoja de cálculo endocrino.xlsx en la que se muestran parte de las variables medidas en un estudio transversal realizado, diseñado originalmente con el objetivo de estimar las prevalencias de diabetes mellitus (DM) en la población de Telde (Gran Canaria), así como para identificar los factores asociados con aquella. En el estudio se incluyeron 1030 sujetos con edades comprendidas entre los 30 y 82 años, que respondieron a un cuestionario diseñado para obtener información sobre la edad, sexo, historia personal y familiar de diabetes mellitus y estilos de vida. Se realizaron mediciones antropométricas y de presión arterial. Se tomaron asimismo muestras de sangre sobre las que se obtuvieron diversas mediciones (glucemia, lípidos, marcadores de inflamación, hemostasia, etc).
A continuación se muestran las primeras lineas de este archivo:
Para leer este archivo con R deberemos ejecutar los tres comandos siguientes:
library(openxlsx)
setwd("c:/Users/aulas/Downloads/")
telde = read.xlsx("endocrino.xlsx") - El primer comando,
library(openxlsx), carga en memoria la librería que permite leer archivos Excel. - El segundo,
setwd("c:/Users/aulas/Downloads/")indica a R la carpeta en la que se encuentra el archivo. Nótese que en R las carpetas se especifican mediante la barra “/”, y no mediante la barra inversa “\” habitual en Windows. - El tercero,
telde = read.xlsx("endocrino.xlsx")lee los datos y los guarda dentro de un objeto llamadotelde. Este objeto es la base de datos ya leída por R. En la terminología de R,teldees un objeto de tipodata.frame.
Visualización de los datos
Si la base se ha leido correctamente, su nombre debe aparecer en la ventana superior derecha de Rstudio, bajo la pestaña Environment:
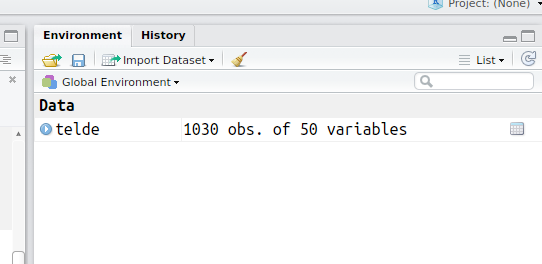
Si el nombre de la base de datos no aparece ahí es que no se ha leído correctamente.
Podemos ver las primeras líneas de esta base de datos simplemente tecleando head(telde) en la consola y pulsando Return. De esta forma podemos hacernos una primera idea de qué variables contiene y qué tipo de valores toma:
head(telde)## ID EDAD SEXO PESO TALLA SEDENTARIO INSTRUCCION TAS TAD HTA_conocida
## 1 22 51 1 86.3 174 0 Primer grado 125 80 1
## 2 65 33 1 81.8 175 0 Segundo grado 120 80 0
## 3 116 40 1 105.0 173 1 Segundo grado 150 100 0
## 4 144 62 1 98.0 172 0 Primer grado 155 100 0
## 5 204 33 1 124.0 181 0 Segundo grado 130 90 0
## 6 337 33 1 93.0 177 1 Segundo grado 110 70 0
## HTA_OMS A_DIAB ECV_B TABACO ALCOHOL STATIN CINTURA CADERA OBCENT_ATP
## 1 1 1 1 0 1 1 104 101.5 1
## 2 0 0 0 1 1 0 86 89.0 0
## 3 1 1 0 1 1 0 116 107.0 1
## 4 1 0 0 1 0 0 104 103.0 1
## 5 1 1 0 0 0 0 130 122.0 1
## 6 0 1 0 0 1 0 108 106.0 1
## COLESTEROL HDL LDL LDL_C TG CnoHDL ApoA ApoB LPA A1C hba1
## 1 190 50 123 120 - 140 86 140 138.1 78 4.78 6.1730 7.9
## 2 217 42 144 140 - 160 155 175 93.1 94 2.39 5.5820 7.2
## 3 288 44 NA <NA> 448 244 122.7 120 39.30 5.3850 7.1
## 4 225 45 153 140 - 160 132 180 115.8 101 7.63 5.3850 6.6
## 5 224 58 154 140 - 160 62 166 135.2 86 24.90 5.1880 6.3
## 6 163 40 111 100 - 120 59 123 98.5 71 2.41 4.8925 6.0
## CREATININA GLUCB SOG Tol_Glucosa DM conocida SM PCR INSULINEMIA PAI_1
## 1 0.9 101 122 IFG 0 Normal 1 0.34 16.2 47.8
## 2 1.0 103 100 IFG 0 Normal 0 0.32 19.8 57.2
## 3 1.0 103 90 IFG 0 Normal 1 0.58 11.5 38.5
## 4 0.9 109 96 IFG 0 Normal 1 0.32 17.8 32.4
## 5 0.8 107 69 IFG 0 Normal 1 0.32 11.6 32.0
## 6 0.9 103 117 IFG 0 Normal 0 0.34 22.7 71.7
## fvw fibri HMC HOMA IR ecnos ppr fibratos CETP PON_192
## 1 115.0 2.40 14.00 4.036687 1 ab pp No <NA> QR
## 2 17.8 2.89 40.72 5.031426 1 bb pp No <NA> QQ
## 3 91.0 2.71 15.34 2.922293 1 bb pp No <NA> QR
## 4 96.1 2.45 15.33 4.786689 1 bb pp No B1B1 RR
## 5 75.0 3.78 8.64 3.062178 1 <NA> <NA> No <NA> <NA>
## 6 117.0 3.53 13.20 5.768352 1 bb pp No <NA> QRPodemos ver su estructura con más detalle mediante la función str(), lo que nos permite comprobar si las variables numéricas se han leído como numéricas y si las variables tipo carácter se han leido como caracteres:
str(telde)## 'data.frame': 1030 obs. of 50 variables:
## $ ID : num 22 65 116 144 204 337 458 541 551 557 ...
## $ EDAD : num 51 33 40 62 33 33 68 56 55 46 ...
## $ SEXO : num 1 1 1 1 1 1 1 1 1 1 ...
## $ PESO : num 86.3 81.8 105 98 124 93 81 99 92 94.3 ...
## $ TALLA : num 174 175 173 172 181 177 174 174 179 169 ...
## $ SEDENTARIO : num 0 0 1 0 0 1 0 1 1 0 ...
## $ INSTRUCCION : chr "Primer grado" "Segundo grado" "Segundo grado" "Primer grado" ...
## $ TAS : num 125 120 150 155 130 110 120 110 130 120 ...
## $ TAD : num 80 80 100 100 90 70 70 70 80 80 ...
## $ HTA_conocida: num 1 0 0 0 0 0 0 0 0 0 ...
## $ HTA_OMS : num 1 0 1 1 1 0 0 0 0 0 ...
## $ A_DIAB : num 1 0 1 0 1 1 1 0 0 1 ...
## $ ECV_B : num 1 0 0 0 0 0 0 0 0 0 ...
## $ TABACO : num 0 1 1 1 0 0 0 0 0 1 ...
## $ ALCOHOL : num 1 1 1 0 0 1 0 1 1 1 ...
## $ STATIN : num 1 0 0 0 0 0 0 1 0 0 ...
## $ CINTURA : num 104 86 116 104 130 108 100 114 105 109 ...
## $ CADERA : num 102 89 107 103 122 ...
## $ OBCENT_ATP : num 1 0 1 1 1 1 0 1 1 1 ...
## $ COLESTEROL : num 190 217 288 225 224 163 217 306 283 218 ...
## $ HDL : num 50 42 44 45 58 40 49 53 62 44 ...
## $ LDL : num 123 144 NA 153 154 111 138 205 186 120 ...
## $ LDL_C : chr "120 - 140" "140 - 160" NA "140 - 160" ...
## $ TG : num 86 155 448 132 62 59 149 239 175 271 ...
## $ CnoHDL : num 140 175 244 180 166 123 168 253 221 174 ...
## $ ApoA : num 138.1 93.1 122.7 115.8 135.2 ...
## $ ApoB : num 78 94 120 101 86 71 NA 150 130 99 ...
## $ LPA : num 4.78 2.39 39.3 7.63 24.9 2.41 15.3 7.31 36.7 28.9 ...
## $ A1C : num 6.17 5.58 5.38 5.38 5.19 ...
## $ hba1 : num 7.9 7.2 7.1 6.6 6.3 6 8.1 7.7 6.8 7.3 ...
## $ CREATININA : num 0.9 1 1 0.9 0.8 0.9 1 1 1.2 1 ...
## $ GLUCB : num 101 103 103 109 107 103 104 101 115 101 ...
## $ SOG : num 122 100 90 96 69 117 166 94 162 143 ...
## $ Tol_Glucosa : chr "IFG" "IFG" "IFG" "IFG" ...
## $ DM : num 0 0 0 0 0 0 0 0 0 0 ...
## $ conocida : chr "Normal" "Normal" "Normal" "Normal" ...
## $ SM : num 1 0 1 1 1 0 0 1 1 1 ...
## $ PCR : num 0.34 0.32 0.58 0.32 0.32 0.34 0.34 0.34 0.34 0.49 ...
## $ INSULINEMIA : num 16.2 19.8 11.5 17.8 11.6 22.7 14.7 17.2 14.6 15.6 ...
## $ PAI_1 : num 47.8 57.2 38.5 32.4 32 71.7 46.3 56.8 71.1 31.6 ...
## $ fvw : num 115 17.8 91 96.1 75 117 111 75.4 150 126 ...
## $ fibri : num 2.4 2.89 2.71 2.45 3.78 3.53 3.85 3.18 3.2 2.64 ...
## $ HMC : num 14 40.72 15.34 15.33 8.64 ...
## $ HOMA : num 4.04 5.03 2.92 4.79 3.06 ...
## $ IR : num 1 1 1 1 1 1 1 1 1 1 ...
## $ ecnos : chr "ab" "bb" "bb" "bb" ...
## $ ppr : chr "pp" "pp" "pp" "pp" ...
## $ fibratos : chr "No" "No" "No" "No" ...
## $ CETP : chr NA NA NA "B1B1" ...
## $ PON_192 : chr "QR" "QQ" "QR" "RR" ...
Posibles problemas que podemos encontrar.
Con la lectura de archivos Excel
Hay dos problemas muy frecuentes con los que se encuentra en usuario novel cuando lee archivos Excel con R:
Error: no se pudo encontrar la función "read.xlsx": esto significa que queremos ejecutar la funciónread.xlsx()sin haber cargado aún el paqueteopenxlsx. Lo único que hay que hacer es ejecutar primerolibrary(openxlsx)y volver a hacertelde = read.xlsx("endocrino.xlsx").Error in library(openxlsx) : there is no package called ???openxlsx???: esto significa que no tenemos el paqueteopenxlxsinstalado en nuestro ordenador. Lo único que habrá que hacer es instalarlo medianteinstall.packages("openxlsx"), y a continuación volver a ejecutarlibrary(openxlsx).
Con el establecimiento del directorio de trabajo.
Si nos aparece el mensaje:
Error in setwd("ruta/directorio") : no es posible cambiar el directorio de trabajo
ello significa que el directorio o carpeta que hemos especificado no existe. En las aulas de informática de la ULPGC, cuando un archivo se descarga de internet lo hace automáticamente a la carpeta c:\Users\aulas\Downloads. En ordenadores Mac, la carpeta de descargas suele ser /Users/nombreUsuario/Downloads (nótese que en Mac no hay que especificar la unidad c: al principio de la ruta).
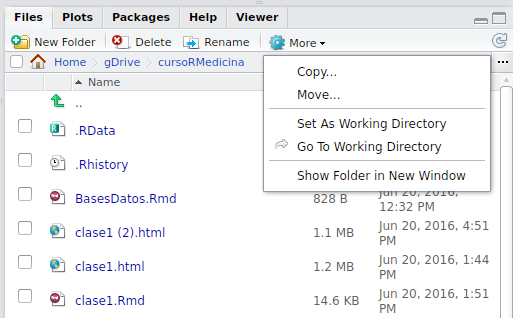
Si desconocemos la ubicación de la carpeta, podemos ir a la pestaña “Files” en la ventana inferior derecha de Rstudio y navegar por las carpetas de nuestro ordenador hasta localizar el archivo. A continuación pinchamos en “More” en la misma ventana, y elegimos la opción “Set As Working Directory”; como resultado, R se situará en dicha carpeta y se mostrará su ruta en la consola de Rstudio.
Con la no asignación de un nombre al objeto resultante de la lectura de la base de datos.
Si nos limitamos a escribir:
read.xlsx("endocrino.xlsx")el archivo se lee, y se muestra en pantalla, pero no se asigna a ningún data.frame por lo que no se conserva en la memoria del ordenador y R no puede acceder a él.