Gráficamente:
Un vector de variables aleatorias \(\mathbf{Y}= \left(y_1,y_2,\dots, y_n\right)'\) sigue un modelo lineal con variables independientes o explicativas \(X_1, X_2, \dots, X_p\) si:
\[\mathbf{Y} = \beta_{0}+\beta_{1}X_{1}+\beta_{2}X_{2}+\dots+\beta_{p}X_{p}+\varepsilon,\,\,\,\,\,\,\varepsilon\approx N \left(0,\sigma_\varepsilon\right)\]
Esto significa que si sobre los \(n\) objetos (o sujetos) de una muestra se han observado las variables \(X_1, X_2, \dots, X_p\) e \(Y\), entonces para el \(i\)-ésimo elemento de la muestra se tiene que:
\[y_{i}=\beta_{0}+\beta_{1}x_{1i}+\beta_{2}x_{2i}+\dots+\beta_{p}x_{pi}+e_{i}\] siendo \(y_i\) el valor de \(\mathbf{Y}\), y \(x_{ji}\) el valor de la variable \(X_j\) medidos en dicho objeto.
El valor de \(e_i\) es la diferencia entre \(y_i\) y el valor que se puede predecir a partir de las \(X_j\), esto es, \(\hat{y_i}=\beta_{0}+\beta_{1}x_{1i}+\beta_{2}x_{2i}+\dots+\beta_{p}x_{pi}\).
Asumiremos que los valores de \(e_i\) siguen una distribución \(N\left(0,\sigma_\varepsilon\right)\)
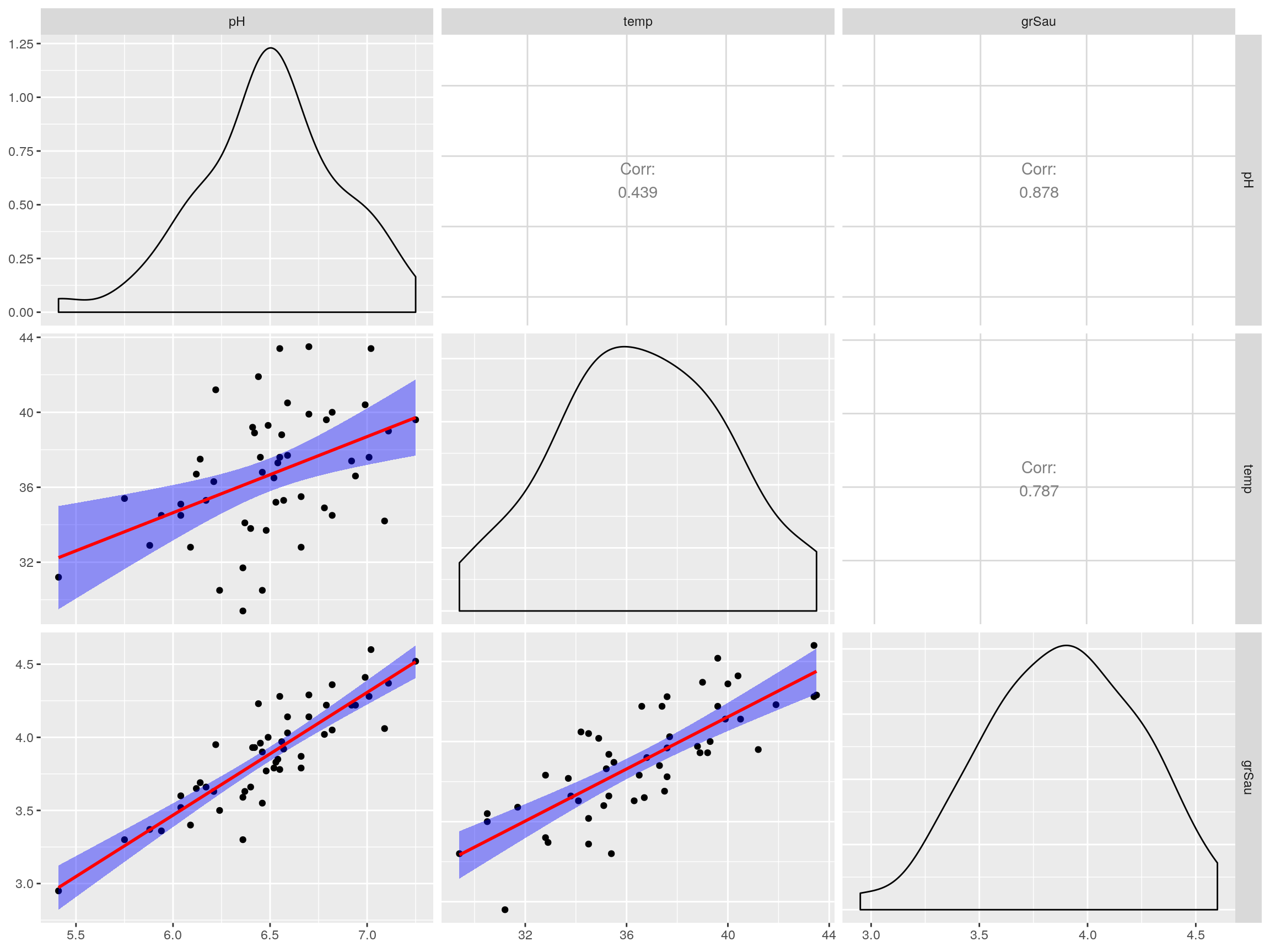
A continuación se muestran algunos de los datos obtenidos en un experimento para medir la velocidad de crecimiento de la bacteria S. aurea sobre un sustrato vegetal (verduras de ensalada) bajo diversas condiciones de pH y temperatura:
##
## ---------------------
## pH temp grSau
## ------ ------ -------
## 6.37 34.1 3.63
##
## 6.66 35.5 3.87
##
## 6.46 36.8 3.9
##
## 6.94 36.6 4.22
##
## 6.17 35.3 3.66
##
## 6.55 37.6 3.78
##
## 5.94 34.5 3.36
##
## 6.54 37.3 3.85
##
## 6.14 37.5 3.69
##
## 6.7 39.9 4.14
## ---------------------La variable grSau es la velocidad de crecimiento de la bacteria (en cels/ml/min)
Gráficamente:

El modelo lineal puede estimarse por Mínimos cuadrados: Este método consiste en encontrar los valores de los parámetros \(\beta_i\) que minimizan la distancia entre los valores observados y los predichos:
\[\underset{\beta_{0},\beta_{1},\dots,\beta_{p}}{min}\sum_{i=1}^{n}\left(y_{i}-\hat{y}_{i}\right)^{2}=\underset{\beta_{0},\beta_{1},\dots,\beta_{p}}{min}\sum_{i=1}^{n}\left(y_{i}-\beta_{0}-\beta_{1}x_{1i}+\beta_{2}x_{2i}+\dots+\beta_{p}x_{pi}\right)^{2}\]
Sólo en el caso de que los residuos del modelo sean normales e independientes puede llevarse a cabo adecuadamente la inferencia clásica sobre los parámetros del modelo.
En R la estimación del modelo se realiza mediante:
lm(grSau~pH,data=growth)##
## Call:
## lm(formula = grSau ~ pH, data = growth)
##
## Coefficients:
## (Intercept) pH
## -1.5682 0.8393lm(grSau~temp,data=growth)##
## Call:
## lm(formula = grSau ~ temp, data = growth)
##
## Coefficients:
## (Intercept) temp
## 0.90165 0.08131lm(grSau~pH+temp,data=growth)##
## Call:
## lm(formula = grSau ~ pH + temp, data = growth)
##
## Coefficients:
## (Intercept) pH temp
## -2.09547 0.63082 0.05133y de manera más completa:
modelo=lm(grSau~pH+temp,data=growth)
summary(modelo)##
## Call:
## lm(formula = grSau ~ pH + temp, data = growth)
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.186542 -0.037014 0.002805 0.044446 0.112115
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -2.095468 0.158264 -13.24 <2e-16 ***
## pH 0.630820 0.026616 23.70 <2e-16 ***
## temp 0.051333 0.002879 17.83 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.06187 on 47 degrees of freedom
## Multiple R-squared: 0.9706, Adjusted R-squared: 0.9693
## F-statistic: 775.1 on 2 and 47 DF, p-value: < 2.2e-16En esta salida de R podemos apreciar varios elementos importantes del ajuste de un modelo de regresión:
\[H_0:\beta_{1}=\beta_{2}=\dots=\beta_{p}=0\] Si p-valor<0.05 se acepta esta hipótesis, en caso contrario se rechaza. La aceptación de esta hipótesis implica que el modelo ajustado no tiene valor predictivo ni explicativo.
confint(modelo)## 2.5 % 97.5 %
## (Intercept) -2.41385336 -1.77708196
## pH 0.57727519 0.68436502
## temp 0.04554181 0.05712507x0=data.frame(pH=6.75,temp=40)predict(modelo,newdata=x0,interval="confidence")## fit lwr upr
## 1 4.215906 4.190548 4.241263predict(modelo,newdata=x0,interval="prediction")## fit lwr upr
## 1 4.215906 4.088891 4.34292Se puede “visualizar” el grado de ajuste alcanzado presentando gráficamente los valores predichos frente a los observados. Si el modelo es adecuado, los puntos así obtenidos deben disponerse en torno a la diagonal del primer cuadrante.
ggplot(data.frame(obs=growth$grSau,pred=fitted(modelo)), aes(x=obs,y=pred)) + geom_point() +
geom_abline(intercept = 0,slope=1,color="red3")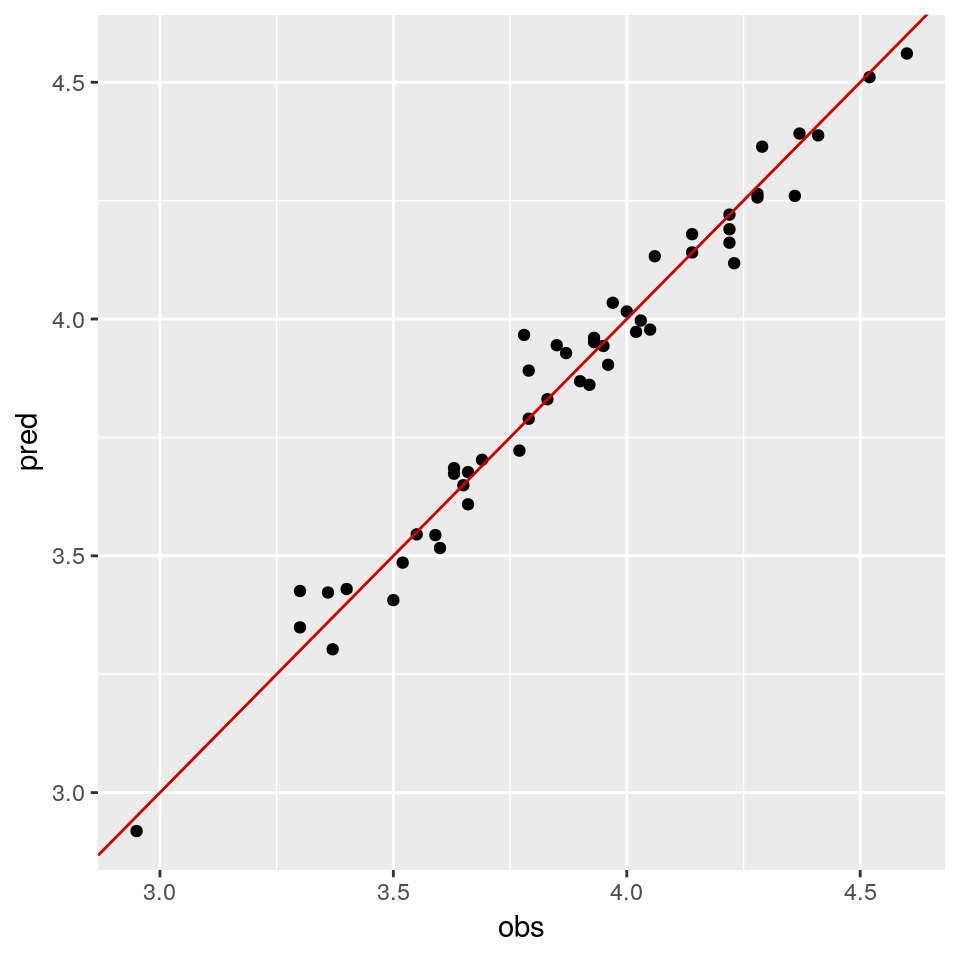
Podemos visualizar la normalidad mediante un histograma de los residuos:
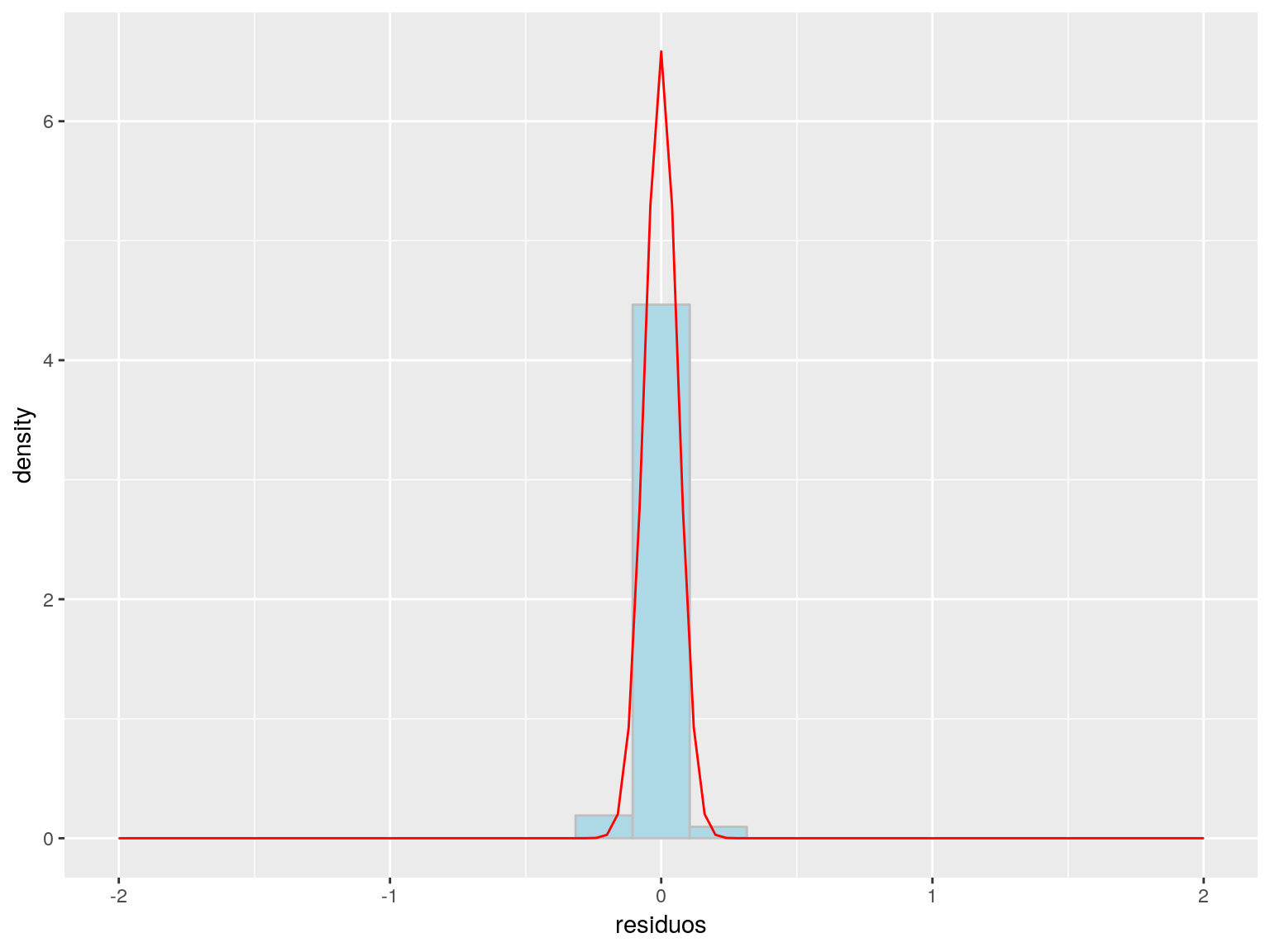
O mejor con un qqplot:
## Loading required package: carData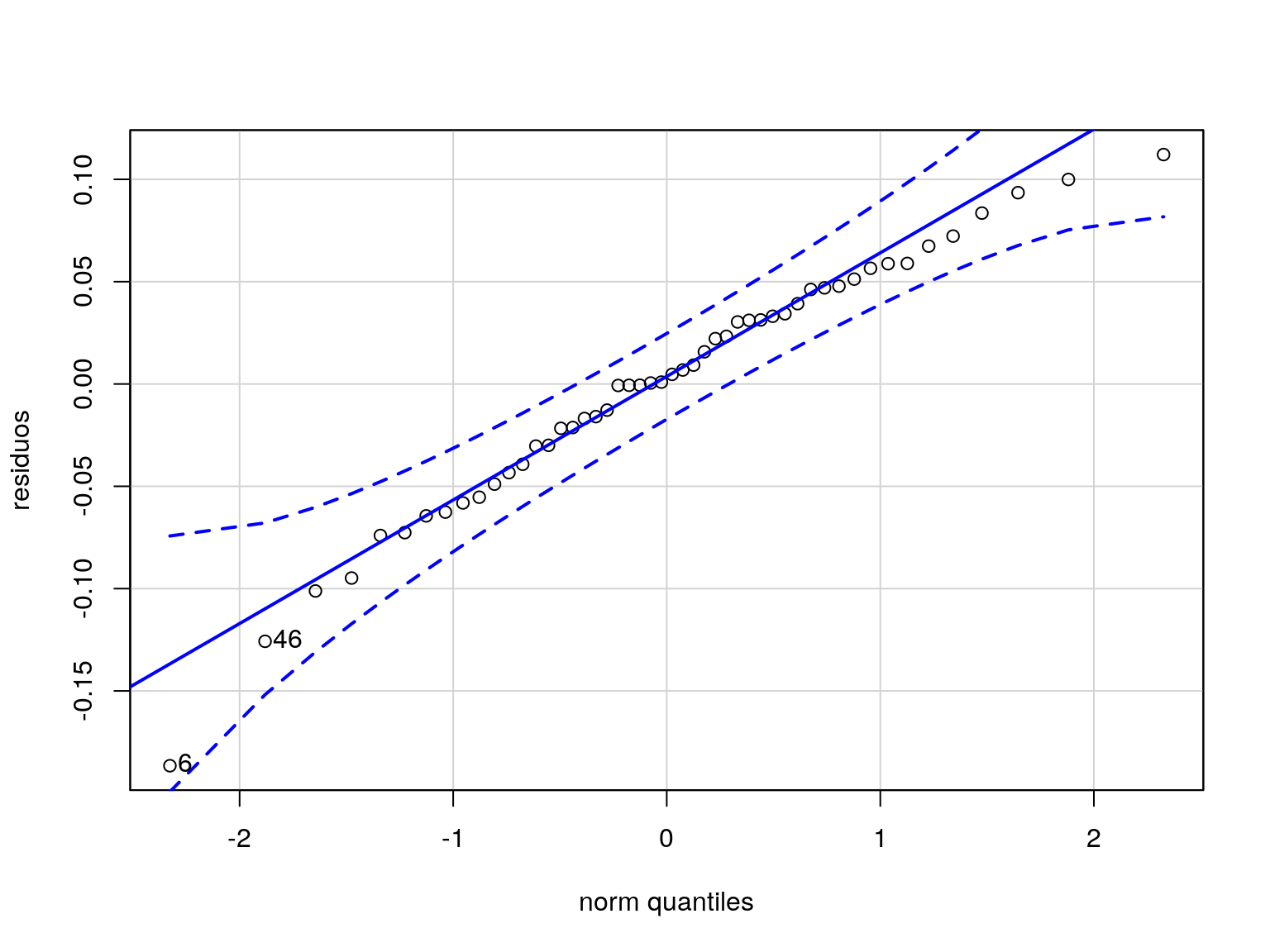
## [1] 6 46El test de Shapiro-Wilk contrasta la normalidad de los residuos:
##
## --------------------------
## Test statistic P value
## ---------------- ---------
## 0.9756 0.3853
## --------------------------
##
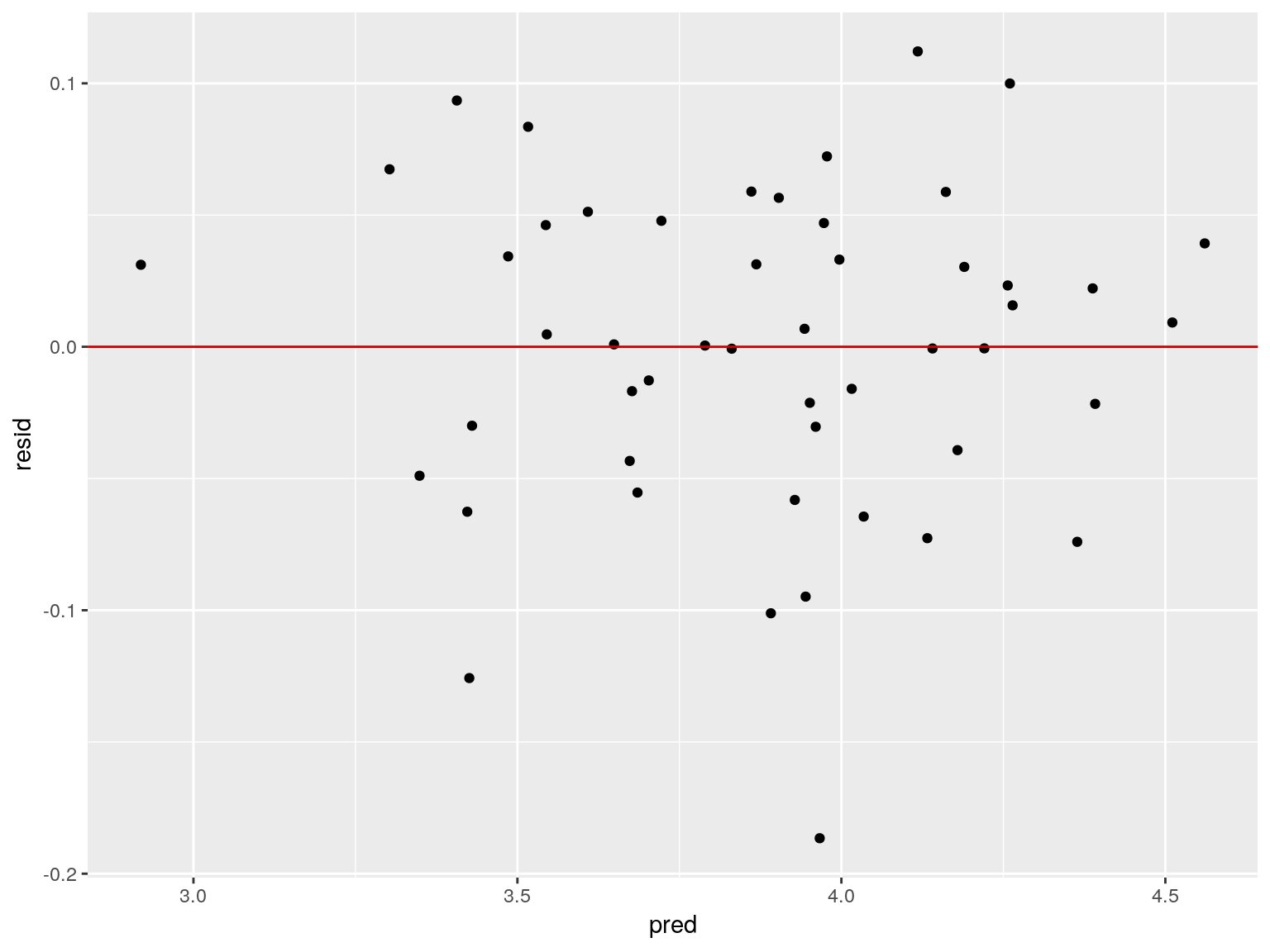
## Table: Shapiro-Wilk normality test: `residuos`Podemos valorar la homoscedasticidad de los residuos graficando los residuos frente a los valores ajustados:
ggplot(data.frame(resid=residuos,pred=fitted(modelo)), aes(x=pred,y=resid)) + geom_point() +
geom_abline(intercept = 0,slope=0,color="red3")