ANOVA
Angelo Santana
2 de octubre de 2017
Análisis de la varianza.
Para este tema utilizaremos los datos del archivo coast.xlsx.
# Lectura de datos
library(readxl)
coast=read_excel("datos/coast.xlsx")Estimación del Modelo de análisis de la varianza para evaluar el efecto de la localización sobre el NH4
Representación gráfica:
- Podemos representar los datos mediante un boxplot: (la variable
locationdebe convertirse primero en factor, ya que es numérica)
coast$location=factor(coast$location)
ggplot(coast,aes(x=location,y=NH4, fill=location))+geom_boxplot() + guides(fill=FALSE)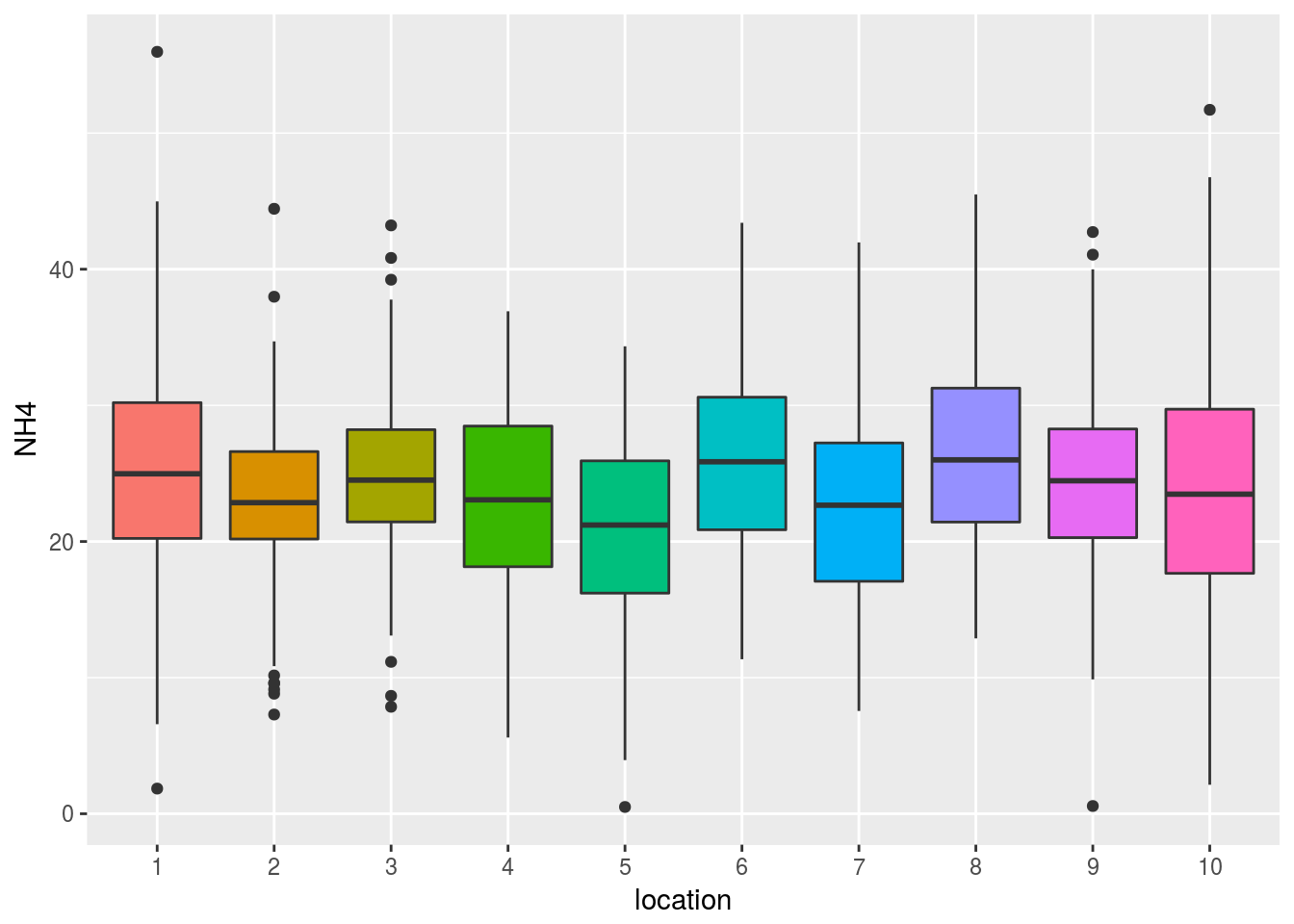
- También podemos representarlos mediante un stripchart:
ggplot(coast,aes(x=location,y=NH4, col=location)) +
geom_jitter(position=position_jitter(0.1)) + guides(col=FALSE)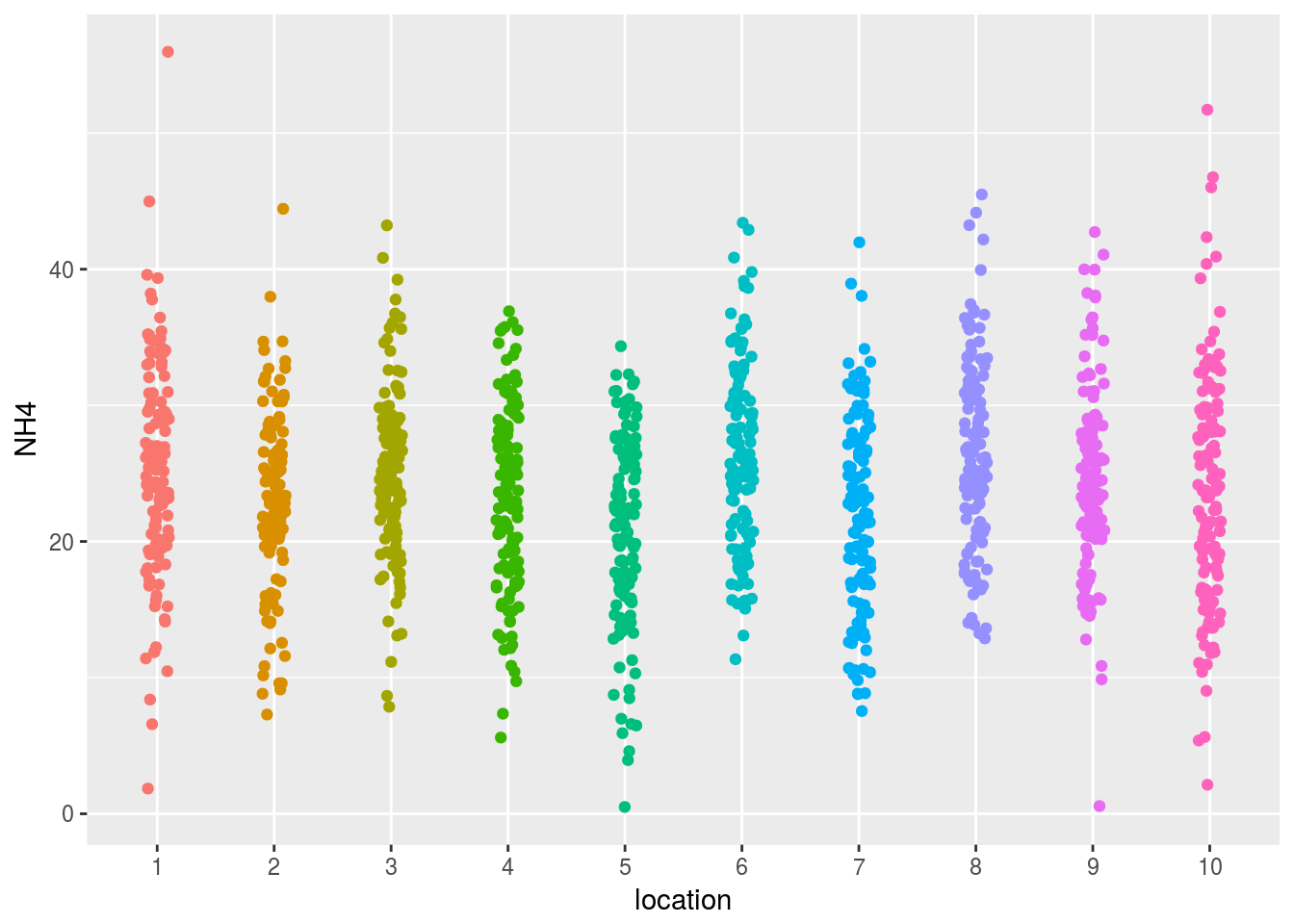
- O mediante ambas figuras combinadas:
ggplot(coast,aes(x=location,y=NH4, col=location)) + geom_boxplot() +
geom_jitter(position=position_jitter(0.1)) + guides(col=FALSE)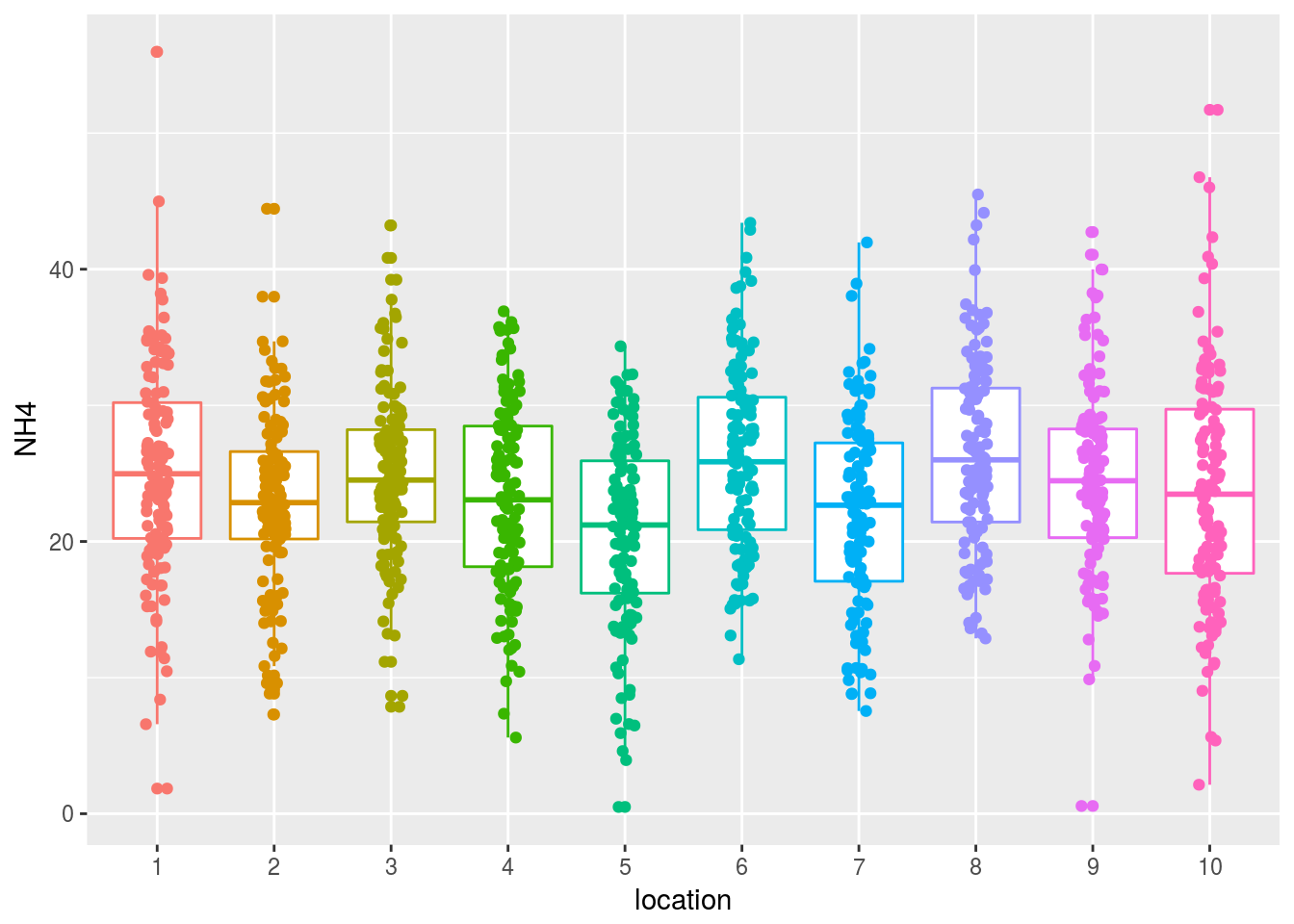
Ajustamos el modelo de análisis de la varianza:
aov1=aov(NH4~location,data=coast)
summary(aov1)## Df Sum Sq Mean Sq F value Pr(>F)
## location 9 3835 426.1 8.27 4.99e-12 ***
## Residuals 1230 63372 51.5
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1El p-valor tan pequeño (<0.05) indica que existen diferencias significativas en NH4 entre las distintas localizaciones. No obstante la validez de este análisis depende de que los residuos del modelo sean normales, y las varianzas en las 10 localizaciones sean iguales.
La normalidad de los residuos podemos comprobarla mediante el test de Shapiro, o gráficamente mediante un qqPlot:
shapiro.test(residuals(aov1))##
## Shapiro-Wilk normality test
##
## data: residuals(aov1)
## W = 0.99749, p-value = 0.05038library(car)
qqPlot(residuals(aov1))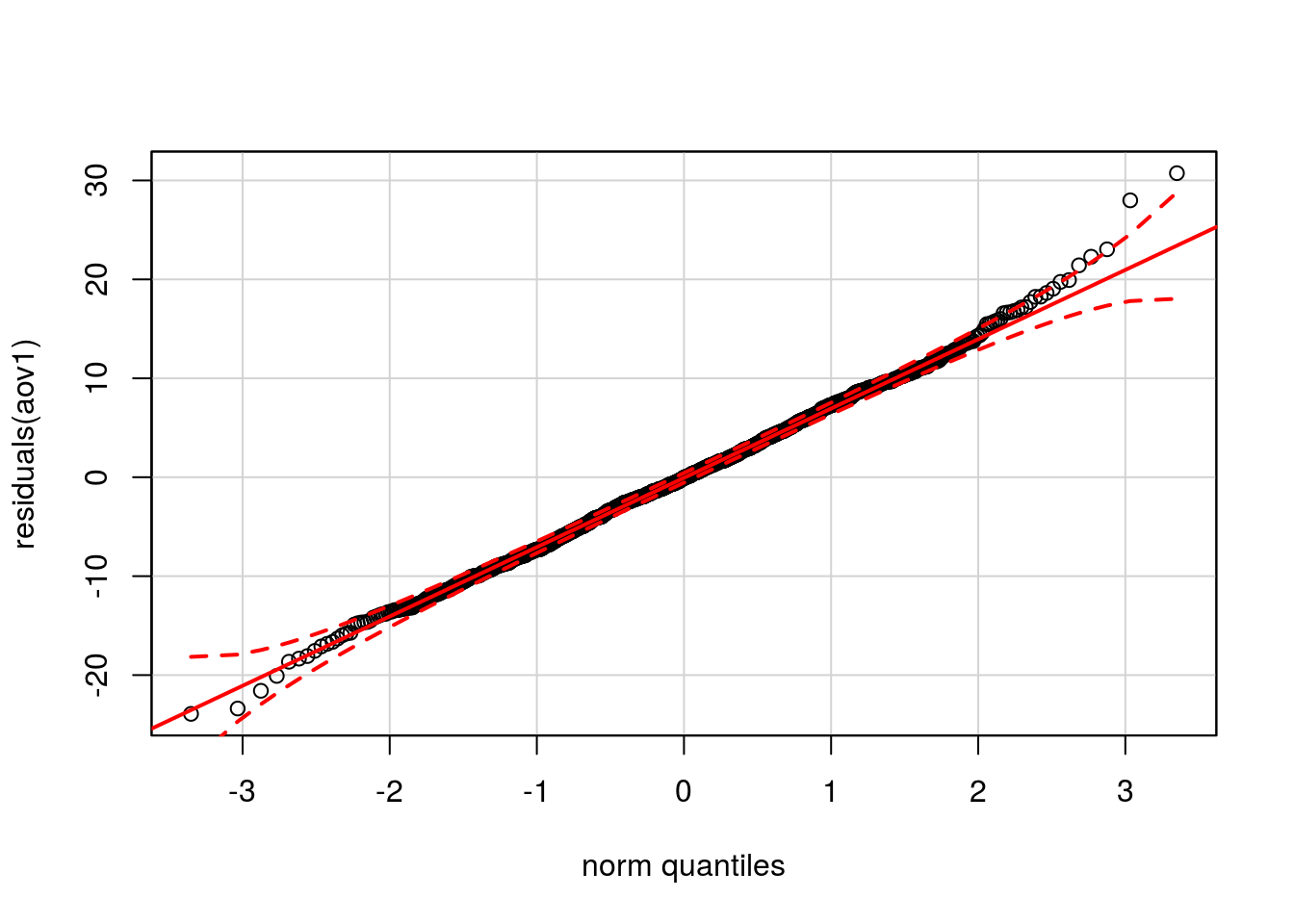
El p-valor es ligeramente mayor que 0.05, por tanto podemos aceptar la normalidad; vemos además que los puntos del gráfico Q-Q se ajustan a la bisectriz del primer cuadrante, lo que indica un buen ajuste entre los residuos y la normalidad; prácticamente todos los puntos (salvo alguno en el extremo superior, lo cual es siempre esperable) caen dentro de la banda de confianza.
Para comprobar la homoscedasticidad (homogeneidad de la varianza) aplicamos el test de Levene:
leveneTest(NH4~location,data=coast)## Levene's Test for Homogeneity of Variance (center = median)
## Df F value Pr(>F)
## group 9 2.6314 0.005121 **
## 1230
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Este test nos indica la presencia de heteroscedasticidad (el p-valor es menor que 0.05), lo que invalida el resultado del análisis de la varianza. Podemos intentar transformar los datos mediante una transformación de Box-Cox:
library(MASS)
boxcox(aov1)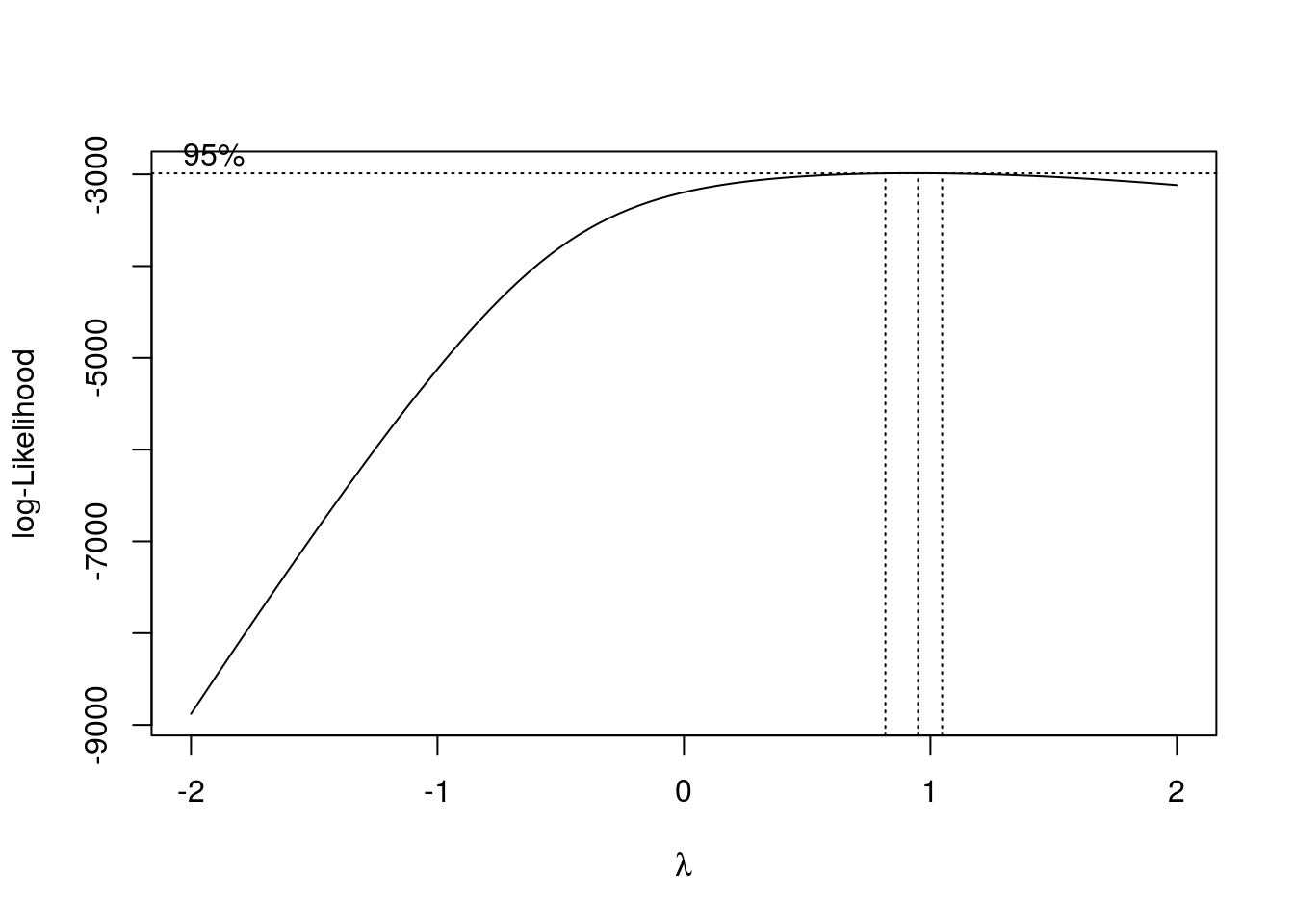
pero el estimador máximo verosímil del parámetro de transformación está en torno a 1, lo que indica que no hay ninguna transformación de esta familia que permita que los residuos sean normales. La transformación Box-Cox es de la forma:
\[y=\frac{x^{\lambda}-1}{\lambda}\] (por tanto \(\lambda=1\) no cambia la escala de \(x\)). Aunque sea posible encontrar valores de \(\lambda\) que permiten alcanzar homoscedasticidad (en este caso por ejemplo \(\lambda=-1\))
lambda=-1
leveneTest((NH4^(lambda)-1)/lambda~location,data=coast)## Levene's Test for Homogeneity of Variance (center = median)
## Df F value Pr(>F)
## group 9 1.296 0.2342
## 1230sin embargo, para dicho valor se pierde la normalidad:
aov2=aov((NH4^(lambda)-1)/lambda~location,data=coast)
shapiro.test(residuals(aov2))##
## Shapiro-Wilk normality test
##
## data: residuals(aov2)
## W = 0.14862, p-value < 2.2e-16Visto que los datos no son homoscedásticos y no es posible transformarlos para conseguir homoscedasticidad y normalidad, procedemos a aplicar un análisis de la varianza no paramétrico (test de Kruskal-Wallis):
kruskal.test(NH4~location,data=coast)##
## Kruskal-Wallis rank sum test
##
## data: NH4 by location
## Kruskal-Wallis chi-squared = 63.872, df = 9, p-value = 2.39e-10El test nos indica que existen diferencias significativas entre localizaciones. Podemos detectar entre qué localizaciones se producen dichas diferencias utilizando el test de Conover (en el paquete PMCMR):
library(PMCMR)
posthoc.kruskal.conover.test(NH4~location,data=coast)## Warning in posthoc.kruskal.conover.test.default(c(16.03, 11.41, 26.08,
## 21.57, : Ties are present. Quantiles were corrected for ties.##
## Pairwise comparisons using Conover's-test for multiple
## comparisons of independent samples
##
## data: NH4 by location
##
## 1 2 3 4 5 6 7 8 9
## 2 0.53907 - - - - - - - -
## 3 1.00000 0.82060 - - - - - - -
## 4 0.82060 1.00000 1.00000 - - - - - -
## 5 0.00011 0.43119 0.00024 0.27231 - - - - -
## 6 1.00000 0.02540 1.00000 0.04552 3.4e-07 - - - -
## 7 0.02647 1.00000 0.04667 1.00000 1.00000 0.00035 - - -
## 8 1.00000 0.01391 1.00000 0.02647 1.3e-07 1.00000 0.00017 - -
## 9 1.00000 1.00000 1.00000 1.00000 0.00450 1.00000 0.33410 0.82060 -
## 10 1.00000 1.00000 1.00000 1.00000 0.10900 0.11677 1.00000 0.07189 1.00000
##
## P value adjustment method: holm
Efecto combinado de la época del año y la localización.
Podemos preguntarnos si las diferencias en varianza que hemos detectado entre las distintas localizaciones podrían deberse a que los datos han sido tomados en momentos distintos (mayo y noviembre). El gráfico siguiente indica que existen diferencias entre los valores de NH4 de esos dos meses:
ggplot(coast,aes(x=month,y=NH4, col=month)) + geom_boxplot() +
geom_jitter(position=position_jitter(0.1)) + guides(col=FALSE)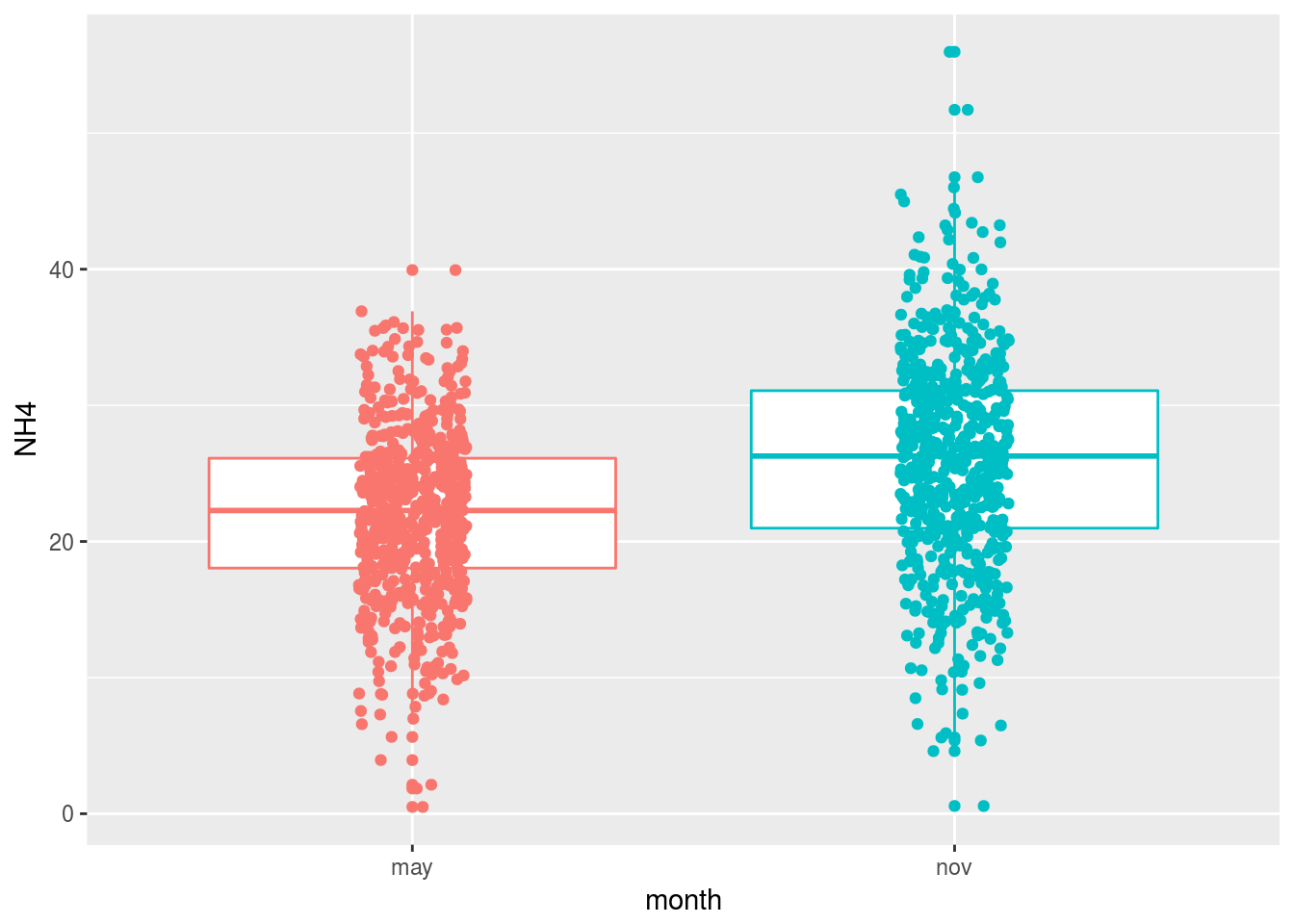
Podemos analizar la diferencia mediante análisis de la varianza:
aov3=aov(NH4~month,coast)
summary(aov3)## Df Sum Sq Mean Sq F value Pr(>F)
## month 1 4878 4878 96.89 <2e-16 ***
## Residuals 1238 62328 50
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1shapiro.test(residuals(aov3))##
## Shapiro-Wilk normality test
##
## data: residuals(aov3)
## W = 0.99736, p-value = 0.03892leveneTest(NH4~month,coast)## Warning in leveneTest.default(y = y, group = group, ...): group coerced to
## factor.## Levene's Test for Homogeneity of Variance (center = median)
## Df F value Pr(>F)
## group 1 25.428 5.276e-07 ***
## 1238
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Vemos que no hay normalidad ni homoscedasticidad. Probamos una transformación Box-Cox:
boxcox(aov3)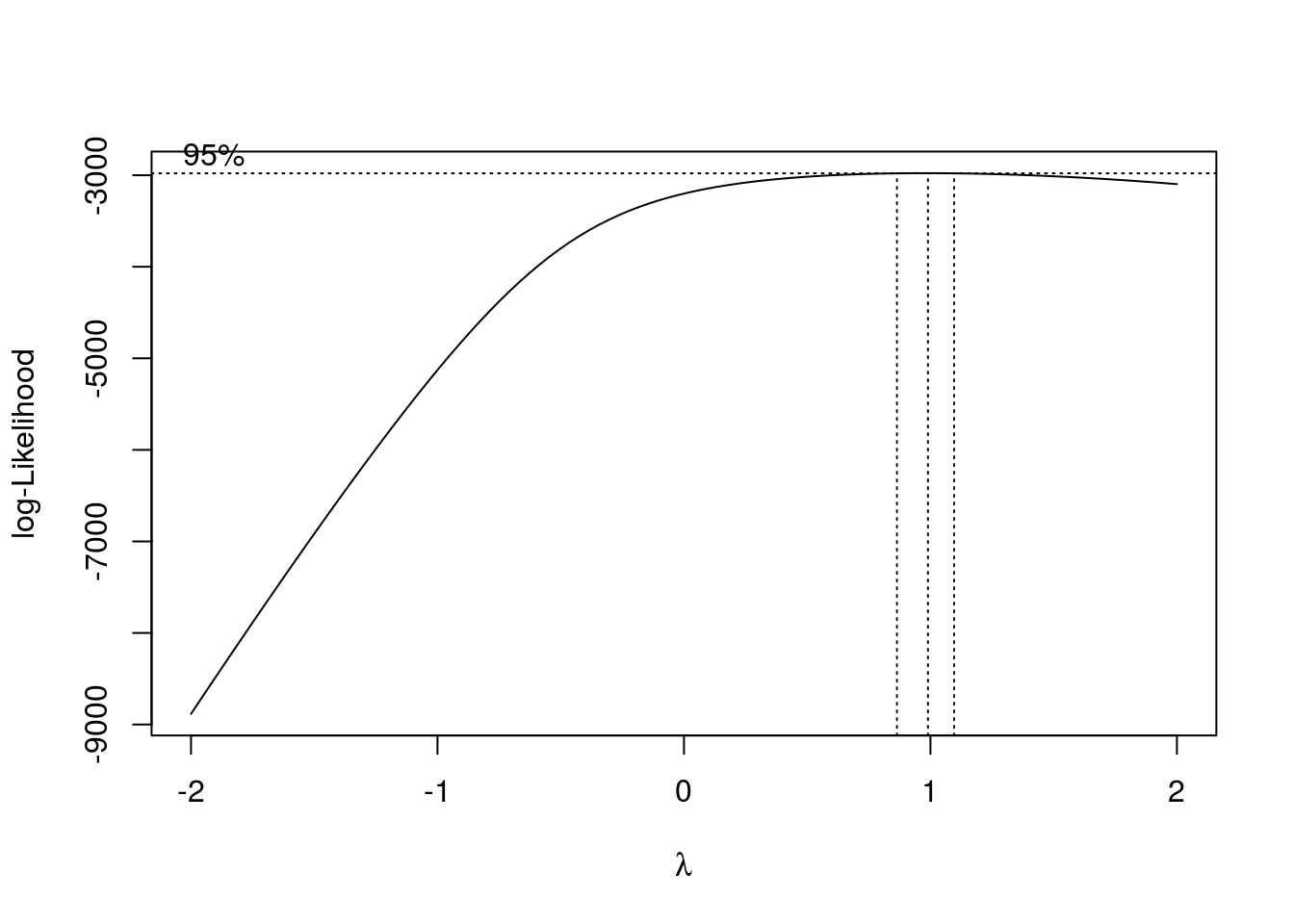
Nuevamente, el estimador de \(\lambda\) es 1 lo que indica que no hay transformación posible. Utilizamos el método de Kruskal-Wallis (convertimos month a factor):
kruskal.test(NH4~factor(month),data=coast)##
## Kruskal-Wallis rank sum test
##
## data: NH4 by factor(month)
## Kruskal-Wallis chi-squared = 89.732, df = 1, p-value < 2.2e-16que nos indica que la diferencia es significativa. Para determinar si existe efecto combinado de localización y mes del año visualizamos primero los datos:
ggplot(coast,aes(x=location,y=NH4, fill=month)) +
geom_jitter(position=position_jitter(0.1),aes(col=month)) +
geom_boxplot() + guides(col=FALSE)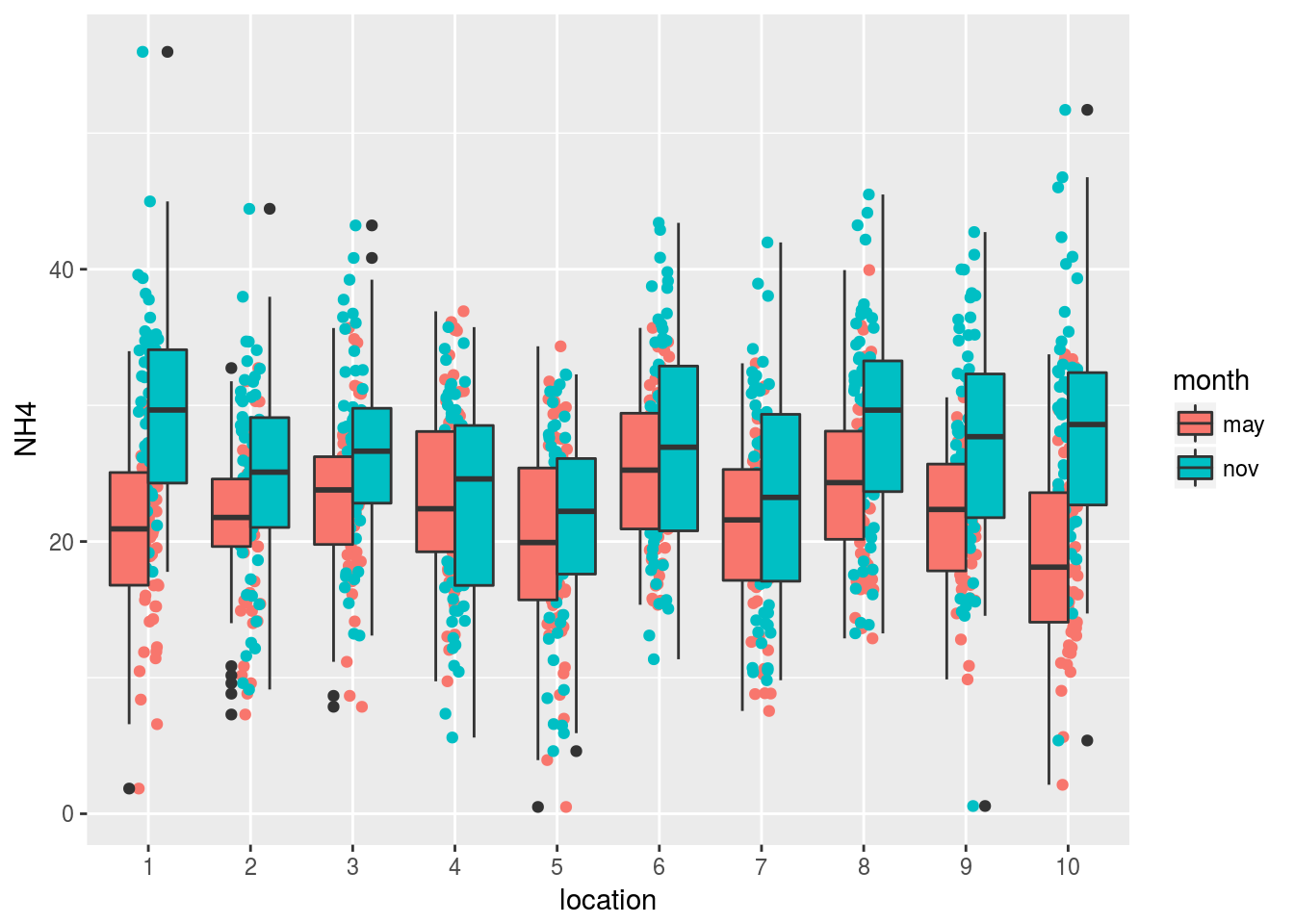
o también:
with(coast,interaction.plot(location,factor(month),NH4,col=2:3))
El análisis conjunto de ambos factores (mes y localización) se lleva a cabo del siguiente modo:
aov4=aov(NH4~month*location,data=coast)
summary(aov4)## Df Sum Sq Mean Sq F value Pr(>F)
## month 1 4878 4878 107.024 < 2e-16 ***
## location 9 3835 426 9.348 7.81e-14 ***
## month:location 9 2885 321 7.033 5.79e-10 ***
## Residuals 1220 55608 46
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Este resultado indica la presencia de interacción, esto es, la diferencia entre meses no es la misma para todas las localizaciones. No obstante, para validar este resultado, comprobamos si se cumplen las condiciones de aplicación del análisis de la varianza:
shapiro.test(residuals(aov4))##
## Shapiro-Wilk normality test
##
## data: residuals(aov4)
## W = 0.9984, p-value = 0.3039leveneTest(NH4~month*location,data=coast)## Levene's Test for Homogeneity of Variance (center = median)
## Df F value Pr(>F)
## group 19 2.3869 0.0007224 ***
## 1220
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Como los residuos son ya normales y el problema es la heteroscedasticidad, la transformación de Box-Cox no ayuda a resolverlo. Por tanto es preciso recurrir a algún test no paramétrico; aunque hay varias opciones según el tipo de diseño del estudio (https://journal.r-project.org/archive/2016/RJ-2016-027/RJ-2016-027.pdf), mostramos aquí una opción sencilla (aunque no la más eficiente). La aplicación directa del método de Kruskal-Wallis no es posible:
kruskal.test(NH4~month*location,data=coast)Es preciso definir una variable con la interacción de los factores localización y mes:
coast$locmes=interaction(coast$location,coast$month)
kruskal.test(NH4~locmes,coast)##
## Kruskal-Wallis rank sum test
##
## data: NH4 by locmes
## Kruskal-Wallis chi-squared = 201.45, df = 19, p-value < 2.2e-16Por tanto existen diferencias significativas. Para localizarlas utilizamos el test de Conover:
posthoc.kruskal.conover.test(NH4~locmes,data=coast)## Warning in posthoc.kruskal.conover.test.default(c(16.03, 11.41, 26.08,
## 21.57, : Ties are present. Quantiles were corrected for ties.##
## Pairwise comparisons using Conover's-test for multiple
## comparisons of independent samples
##
## data: NH4 by locmes
##
## 1.may 2.may 3.may 4.may 5.may 6.may 7.may 8.may
## 2.may 1.00000 - - - - - - -
## 3.may 1.00000 1.00000 - - - - - -
## 4.may 1.00000 1.00000 1.00000 - - - - -
## 5.may 1.00000 1.00000 1.00000 1.00000 - - - -
## 6.may 0.00970 0.04441 1.00000 1.00000 0.00267 - - -
## 7.may 1.00000 1.00000 1.00000 1.00000 1.00000 0.02216 - -
## 8.may 0.21715 0.67638 1.00000 1.00000 0.08036 1.00000 0.41458 -
## 9.may 1.00000 1.00000 1.00000 1.00000 1.00000 0.17269 1.00000 1.00000
## 10.may 1.00000 1.00000 0.06774 0.12440 1.00000 3.1e-05 1.00000 0.00208
## 1.nov 3.6e-11 5.9e-10 3.2e-05 1.3e-05 3.6e-12 0.07032 1.7e-10 0.00226
## 2.nov 0.04706 0.17809 1.00000 1.00000 0.01462 1.00000 0.09871 1.00000
## 3.nov 0.00013 0.00083 0.60162 0.36880 2.6e-05 1.00000 0.00035 1.00000
## 4.nov 1.00000 1.00000 1.00000 1.00000 1.00000 1.00000 1.00000 1.00000
## 5.nov 1.00000 1.00000 1.00000 1.00000 1.00000 0.10949 1.00000 1.00000
## 6.nov 6.7e-05 0.00046 0.42466 0.24727 1.4e-05 1.00000 0.00019 1.00000
## 7.nov 1.00000 1.00000 1.00000 1.00000 1.00000 1.00000 1.00000 1.00000
## 8.nov 1.2e-07 1.3e-06 0.00878 0.00407 1.7e-08 1.00000 4.4e-07 0.20589
## 9.nov 1.8e-05 0.00013 0.19841 0.10949 3.3e-06 1.00000 5.3e-05 1.00000
## 10.nov 4.8e-07 4.7e-06 0.02151 0.01048 7.3e-08 1.00000 1.7e-06 0.41458
## 9.may 10.may 1.nov 2.nov 3.nov 4.nov 5.nov 6.nov
## 2.may - - - - - - - -
## 3.may - - - - - - - -
## 4.may - - - - - - - -
## 5.may - - - - - - - -
## 6.may - - - - - - - -
## 7.may - - - - - - - -
## 8.may - - - - - - - -
## 9.may - - - - - - - -
## 10.may 1.00000 - - - - - - -
## 1.nov 8.8e-09 2.1e-15 - - - - - -
## 2.nov 0.57591 0.00024 0.01517 - - - - -
## 3.nov 0.00480 1.2e-07 1.00000 1.00000 - - - -
## 4.nov 1.00000 0.06495 3.5e-05 1.00000 0.62055 - - -
## 5.nov 1.00000 1.00000 3.5e-09 0.39439 0.00267 1.00000 - -
## 6.nov 0.00281 5.8e-08 1.00000 1.00000 1.00000 0.43887 0.00155 -
## 7.nov 1.00000 0.08036 2.5e-05 1.00000 0.52505 1.00000 1.00000 0.37520
## 8.nov 1.2e-05 2.7e-11 1.00000 0.77500 1.00000 0.00922 5.6e-06 1.00000
## 9.nov 0.00089 1.1e-08 1.00000 1.00000 1.00000 0.20589 0.00047 1.00000
## 10.nov 4.0e-05 1.4e-10 1.00000 1.00000 1.00000 0.02236 1.9e-05 1.00000
## 7.nov 8.nov 9.nov
## 2.may - - -
## 3.may - - -
## 4.may - - -
## 5.may - - -
## 6.may - - -
## 7.may - - -
## 8.may - - -
## 9.may - - -
## 10.may - - -
## 1.nov - - -
## 2.nov - - -
## 3.nov - - -
## 4.nov - - -
## 5.nov - - -
## 6.nov - - -
## 7.nov - - -
## 8.nov 0.00713 - -
## 9.nov 0.17113 1.00000 -
## 10.nov 0.01768 1.00000 1.00000
##
## P value adjustment method: holmEste método lleva a cabo un número elevado de contrastes posiblemente sin interés (por ejemplo comparar la localización 1 en mayo con la 5 en noviembre), por lo que tras aplicar la corrección por la repetición de contrastes es posible que resulten no significativas algunas diferencias que sí lo sean. Otra alternativa es utilizar tests de Mann-Whitney previamente planificados aplicándoles una corrección de Bonferroni.