El modelo lineal
Angelo Santana
25 de septiembre de 2017
Introducción
Para este tema utilizaremos los datos del archivo coast.xlsx.
# Lectura de datos
library(readxl)
costa=read_excel("datos/coast.xlsx")Este archivo contiene datos de variables físico-químicas medidas en 10 puntos distintos de la costa durante los meses de mayo y noviembre. Estas medidas se tomaron cada doce horas en los puntos elegidos.
Exploración de datos.
Antes de ajustar ningún modelo, conviene explorar los datos para detectar particularidades, outliers, etc.
Diferencias entre noviembre y mayo
La siguiente gráfica muestra las posibles diferencias entre los meses del año para las distintas variables del archivo.
library(ggplot2)
library(gridExtra)
costa$obs=1:nrow(costa)
costa=data.frame(costa)
costaRes=reshape(costa,idvar="obs",varying=list(1:17), timevar="variable",
times=names(costa)[1:17],v.names="value",direction="long")
vbles=names(costa)[1:17]
plots=lapply(1:17,function(k) {
ggplot(subset(costaRes,variable==vbles[k]),
aes(x=month,y=value,fill=month)) + geom_boxplot() +
guides(fill=FALSE) + ylab(vbles[k])
})
grid.arrange(grobs=plots[1:6],nrow=3,ncol=2)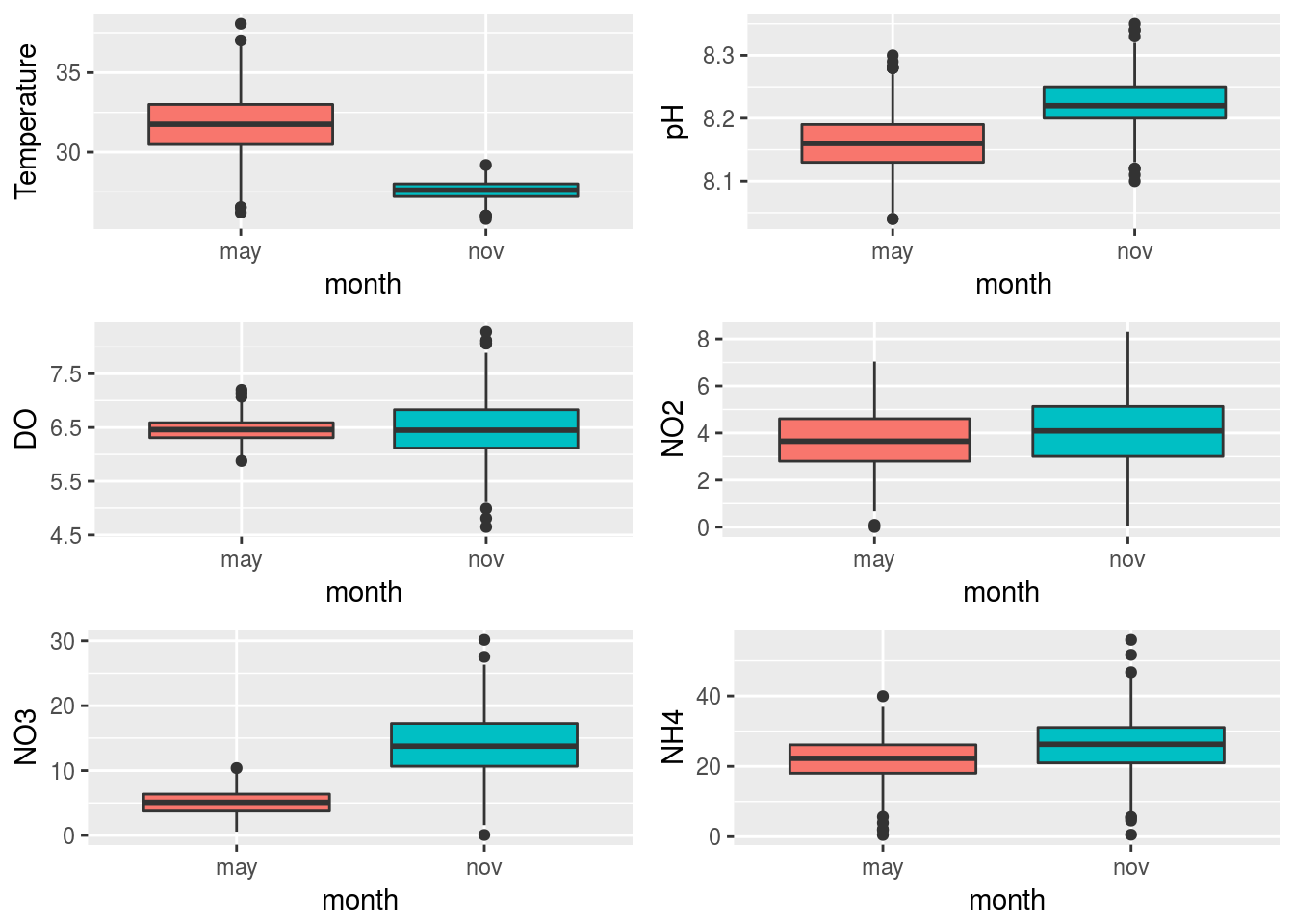
grid.arrange(grobs=plots[7:12],nrow=3,ncol=2)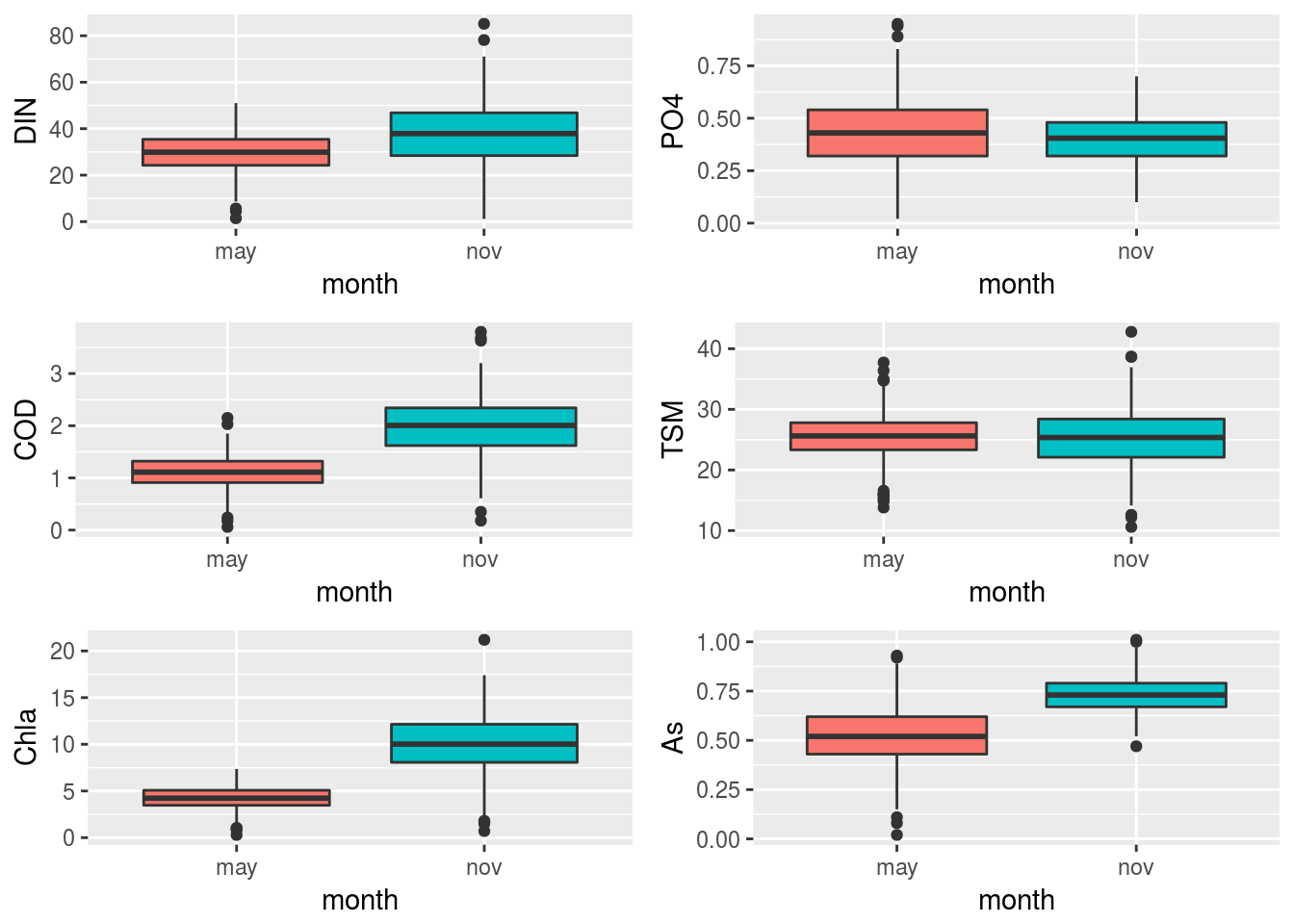
grid.arrange(grobs=plots[13:17],nrow=3,ncol=2)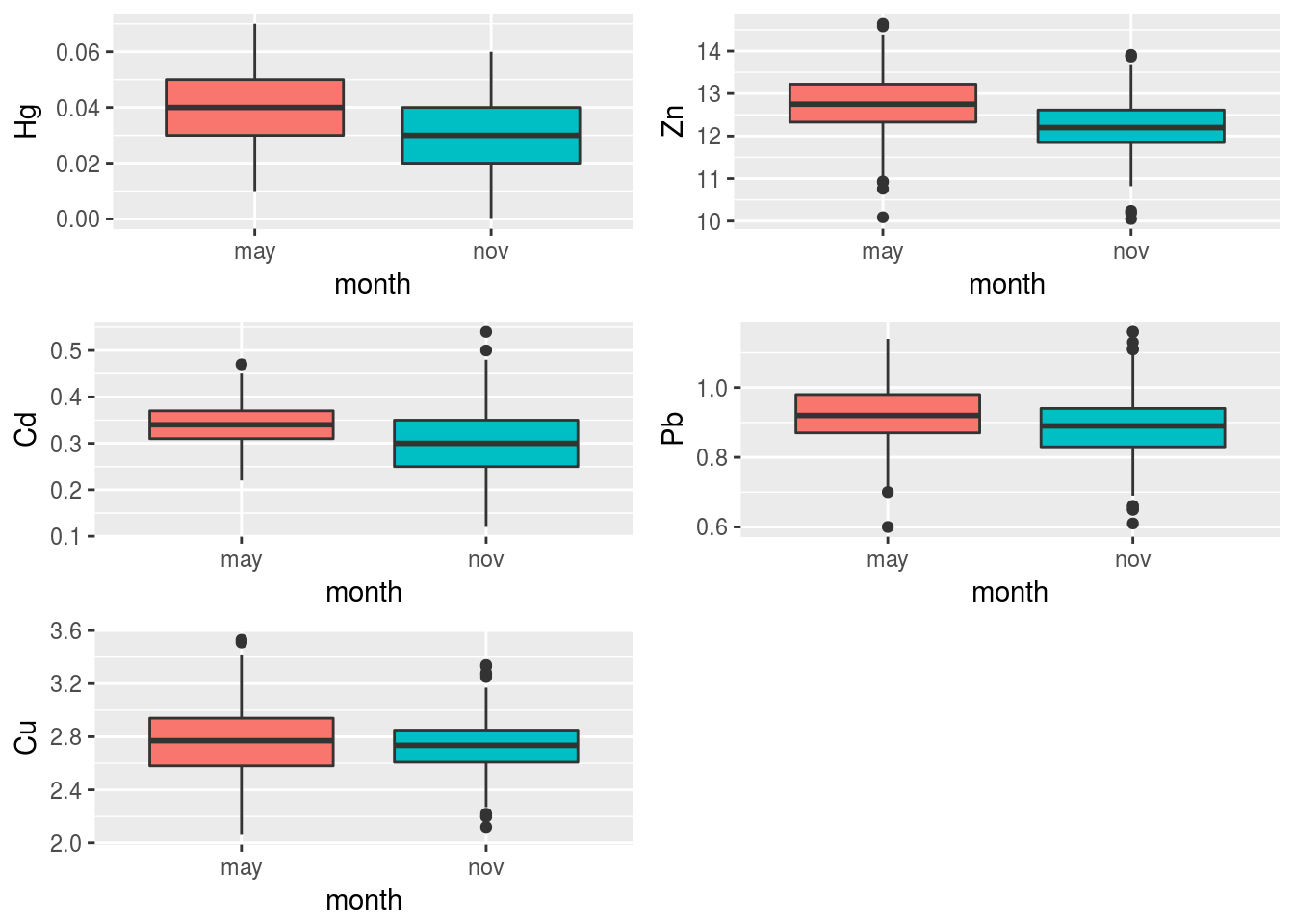
Puede apreciarse que prácticamente todas las variables manifiestan comportamientos diferentes en los dos meses de observación. Por tanto, en el análisis conjunto de las mismas habrá de tenerse el mes en cuenta.
Asociación Temperatura - Clorofila
El siguiente gráfico muestra, a modo de ejemplo, como varía la clorofila en función de la temperatura. Cada uno de los meses se ha señalado en un color distinto:
# Gráfica con el plot básico de R
plot(Chla~Temperature,costa,col=factor(month))
legend("topright",title="Month",c("May","November"),col=c(1,2),pch=1)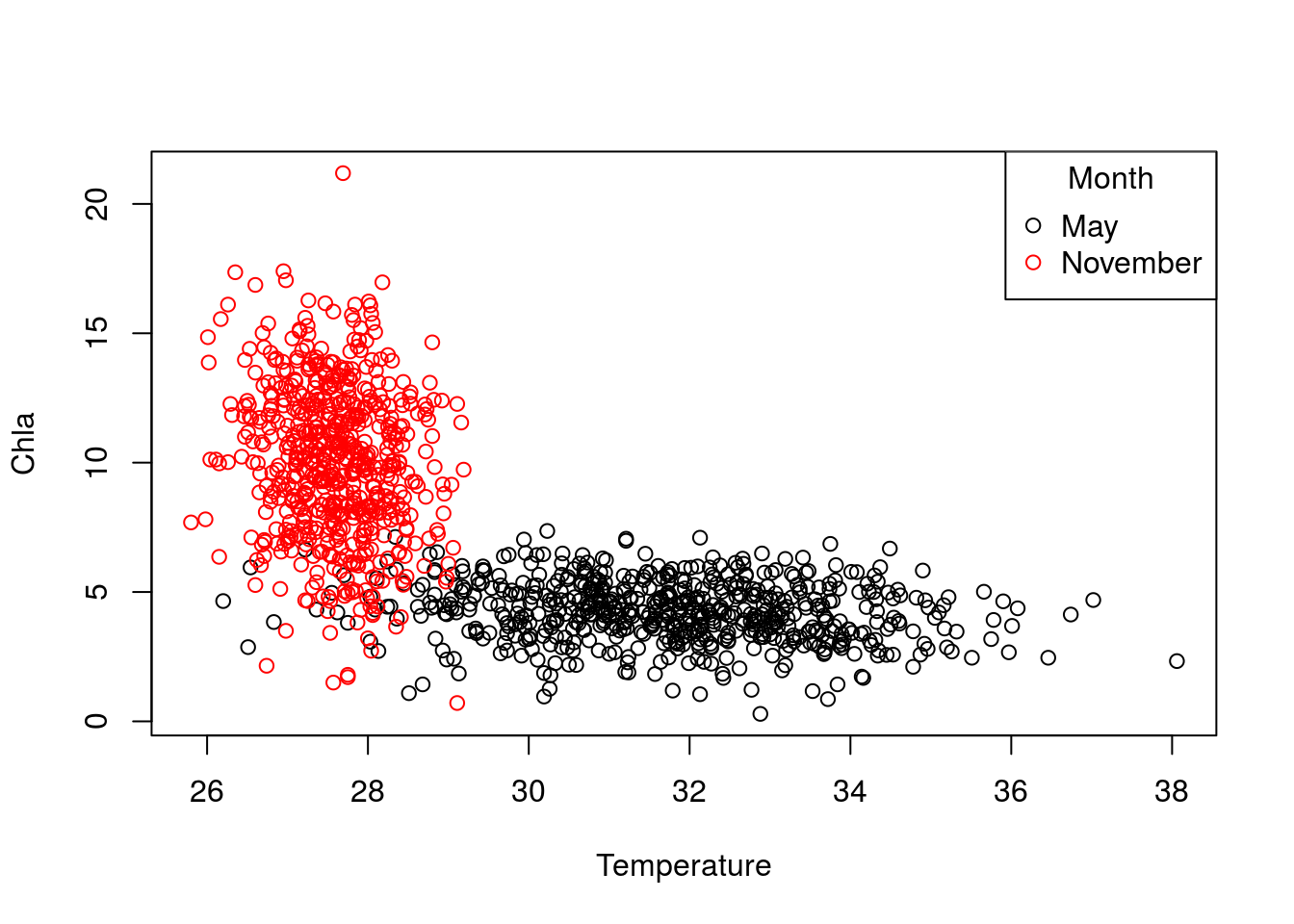
# Misma gráfica con ggplot
ggplot(costa,aes(x=Temperature,y=Chla, col=month)) + geom_point()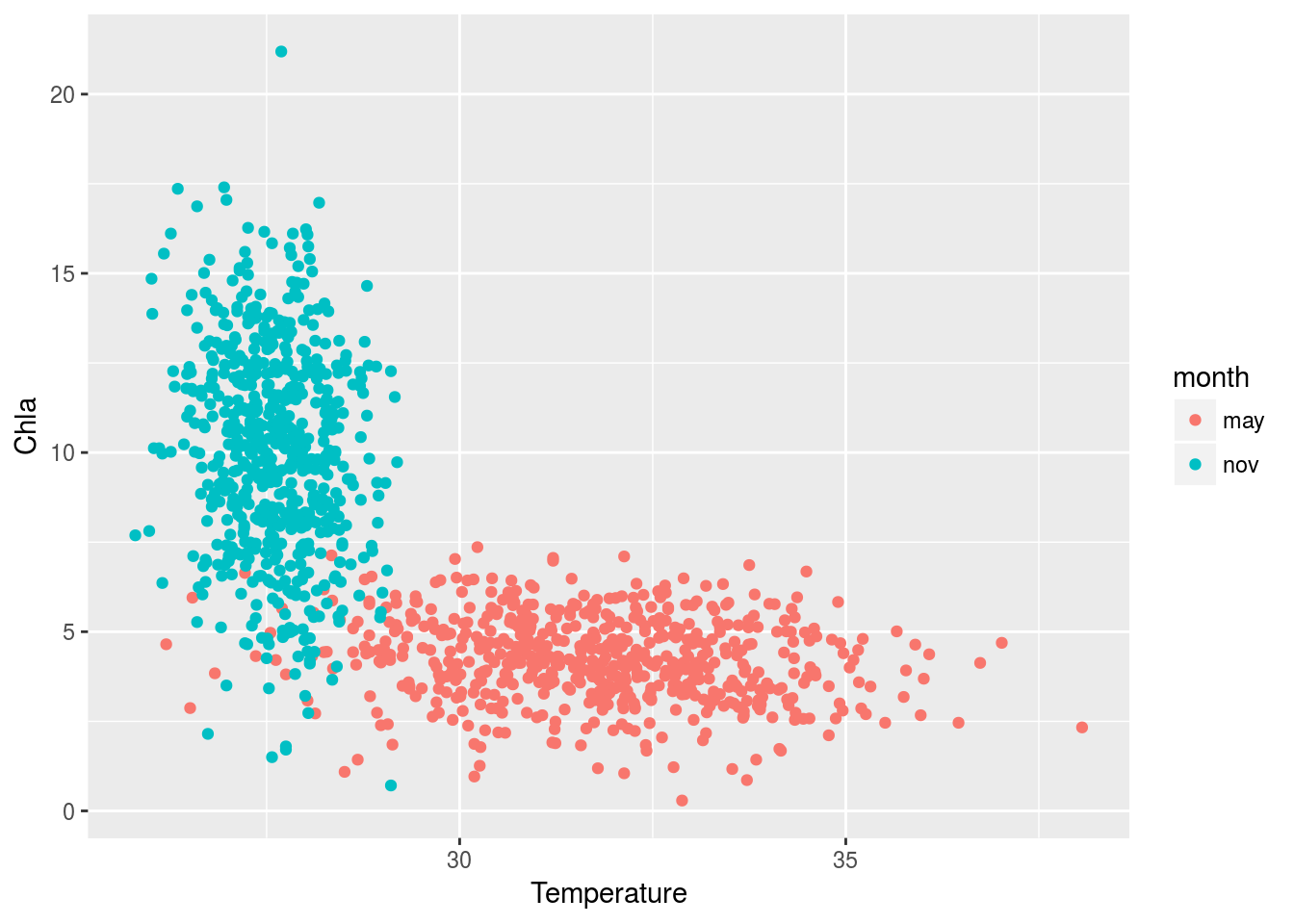
Si calculásemos la correlación entre ambas variables sin tener en cuenta el mes obtenemos un valor de correlación relativamente alto:
with(costa,cor(Temperature,Chla))## [1] -0.6958878lo que significaría la existencia de una fuerte asociación entre clorofila y temperatura.
Sin embargo, la correlación entre ambas variables cada mes es sensiblemente inferior:
may=subset(costa,month=="may")
nov=subset(costa,month=="nov")
with(may,cor(Temperature,Chla))## [1] -0.1712265with(nov,cor(Temperature,Chla))## [1] -0.1617771Esto significa que la correlación detectada inicialmente entre clorofila y temperatura en realidad se debía al efecto del mes (en noviembre hay más clorofila que en mayo, sin que un incremento de temperatura en un mes concreto signifique una variación notable de la clorofila). Podemos ver estos efectos dibujando las correspondientes rectas de regresión:
# Con los gráficos básicos de R
plot(Chla~Temperature,costa,col=factor(month))
legend("topright",title="Month",c("May","November"),col=c(1,2),pch=1)
rectaGlobal=lm(Chla~Temperature,costa)
rectaNov=lm(Chla~Temperature,nov)
rectaMay=lm(Chla~Temperature,may)
abline(rectaGlobal,col=4,lwd=3)
abline(rectaMay,col=1,lwd=2)
abline(rectaNov,col=2,lwd=2)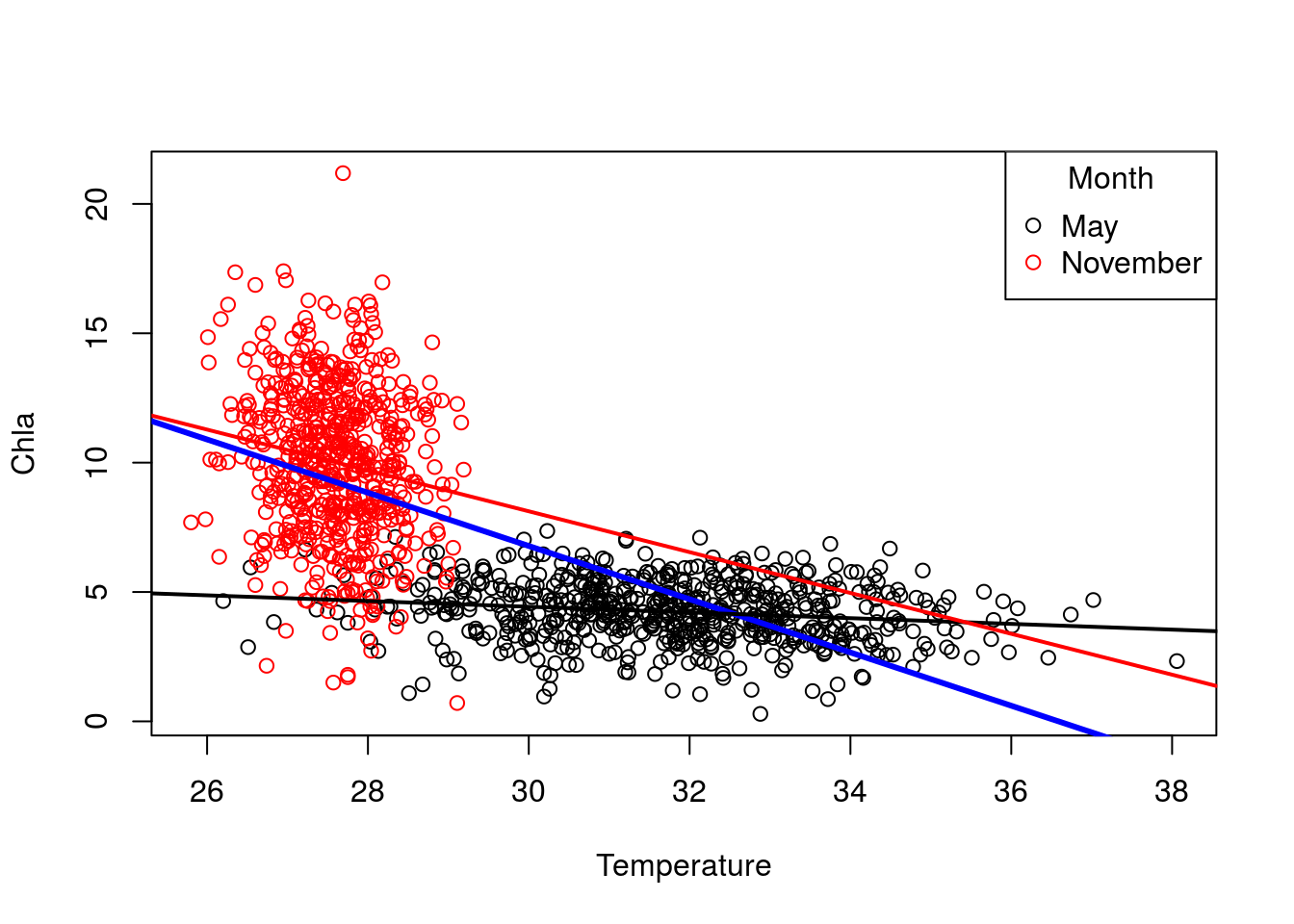
# Con ggplot
ggplot(costa,aes(x=Temperature,y=Chla)) +
geom_point(aes(colour=month)) +
geom_smooth(method="lm", aes(color="all data"), size=1.5) +
geom_smooth(method="lm",aes(color=month)) +
scale_colour_manual(name="month",values=1:3) +
guides(colour = guide_legend(override.aes = list(alpha = 0)))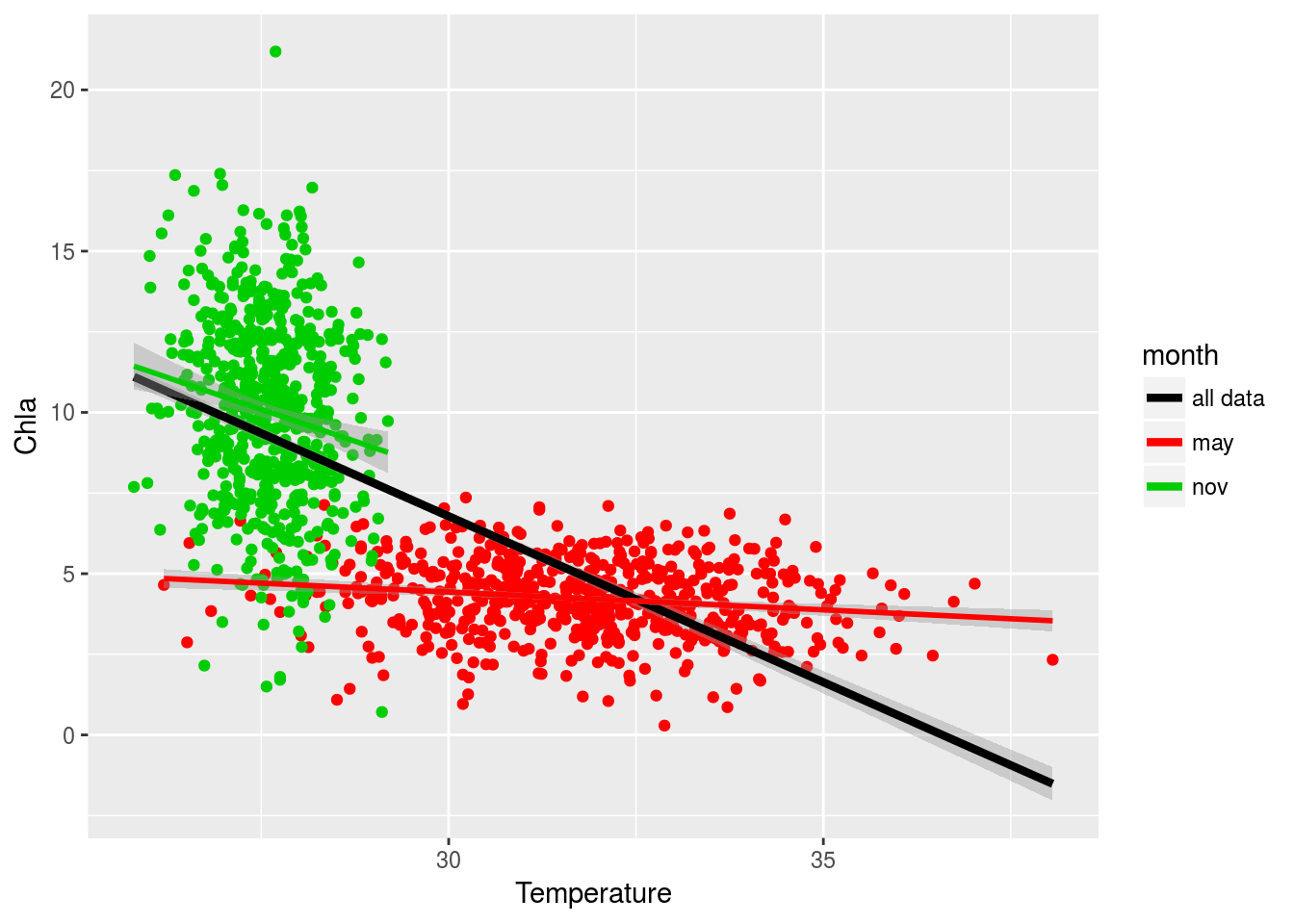
Asociación DIN - NO2
Evaluamos ahora la asociación entre Nitrógeno disuelto (DIN) y dióxido de nitrógeno:
ggplot(costa,aes(x=NO2,y=DIN)) +
geom_point(aes(colour=month,shape=month)) +
geom_smooth(method="lm", size=1.5) +
geom_smooth(method="lm",aes(color=month))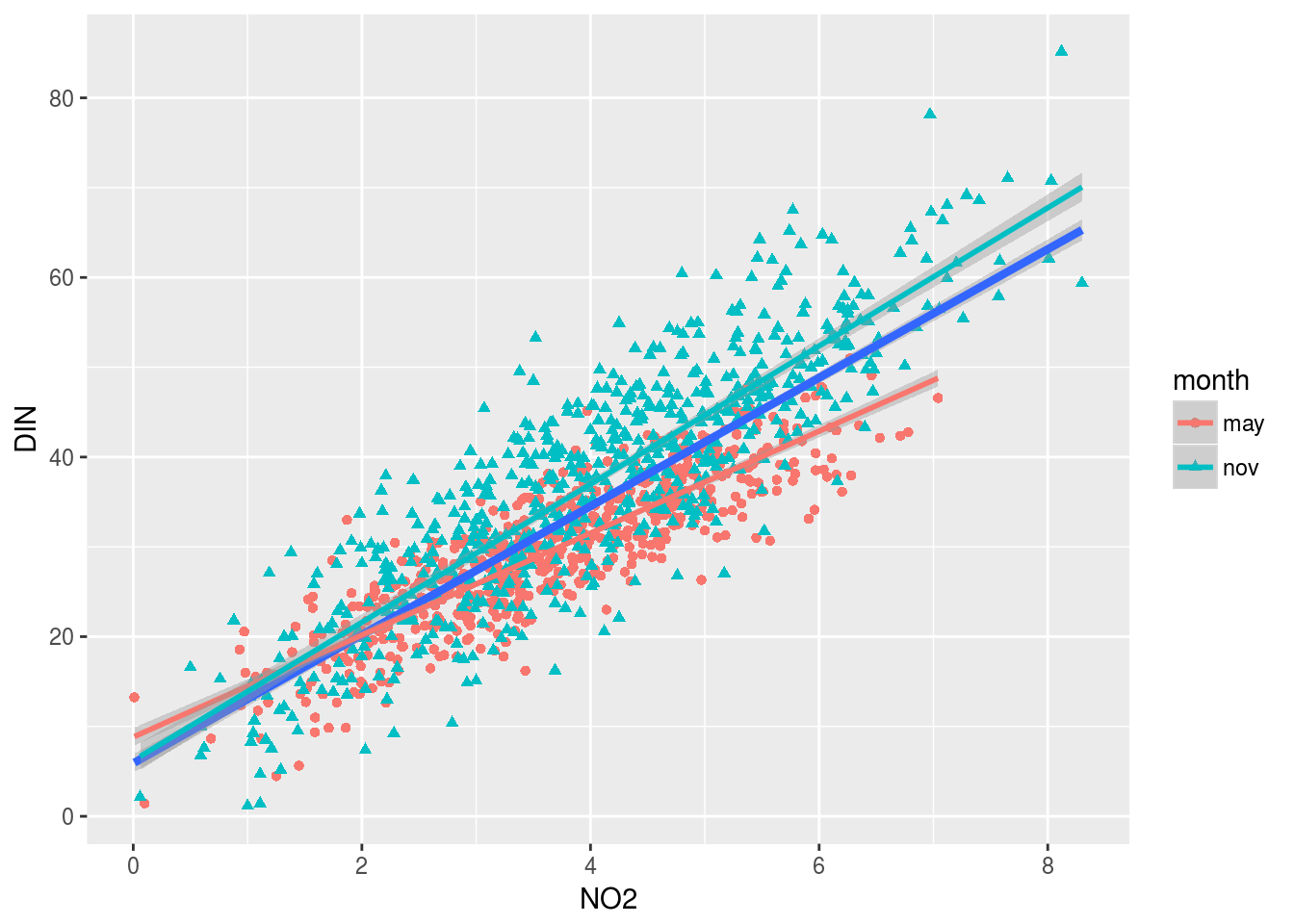
En este caso las dos rectas son muy parecidas:
Para los datos de mayo:
lm(DIN~NO2,may)##
## Call:
## lm(formula = DIN ~ NO2, data = may)
##
## Coefficients:
## (Intercept) NO2
## 8.824 5.677Para los datos de noviembre:
lm(DIN~NO2,nov)##
## Call:
## lm(formula = DIN ~ NO2, data = nov)
##
## Coefficients:
## (Intercept) NO2
## 6.210 7.693Por tanto cabe hacerse la pregunta de si la diferencia observada entre ambas rectas obedece a que la relación entre las variables cambia de mayo a noviembre, o si se debe simplemente al azar del muestreo, y en realidad el comportamiento ambos meses es análogo y se debería ajustar un único modelo.
Diferencias entre localizaciones.
Los datos han sido muestreados en 10 localizaciones distintas. Los siguientes gráficos nos informan del comportamiento de cada variable en las distintas localizaciones:
costaRes$location=factor(costaRes$location)
plots=lapply(1:17,function(k) {
ggplot(subset(costaRes,variable==vbles[k]),
aes(x=location,y=value,fill=location)) + geom_boxplot() +
guides(fill=FALSE) + ylab(vbles[k])
})
grid.arrange(grobs=plots[1:6],nrow=3,ncol=2)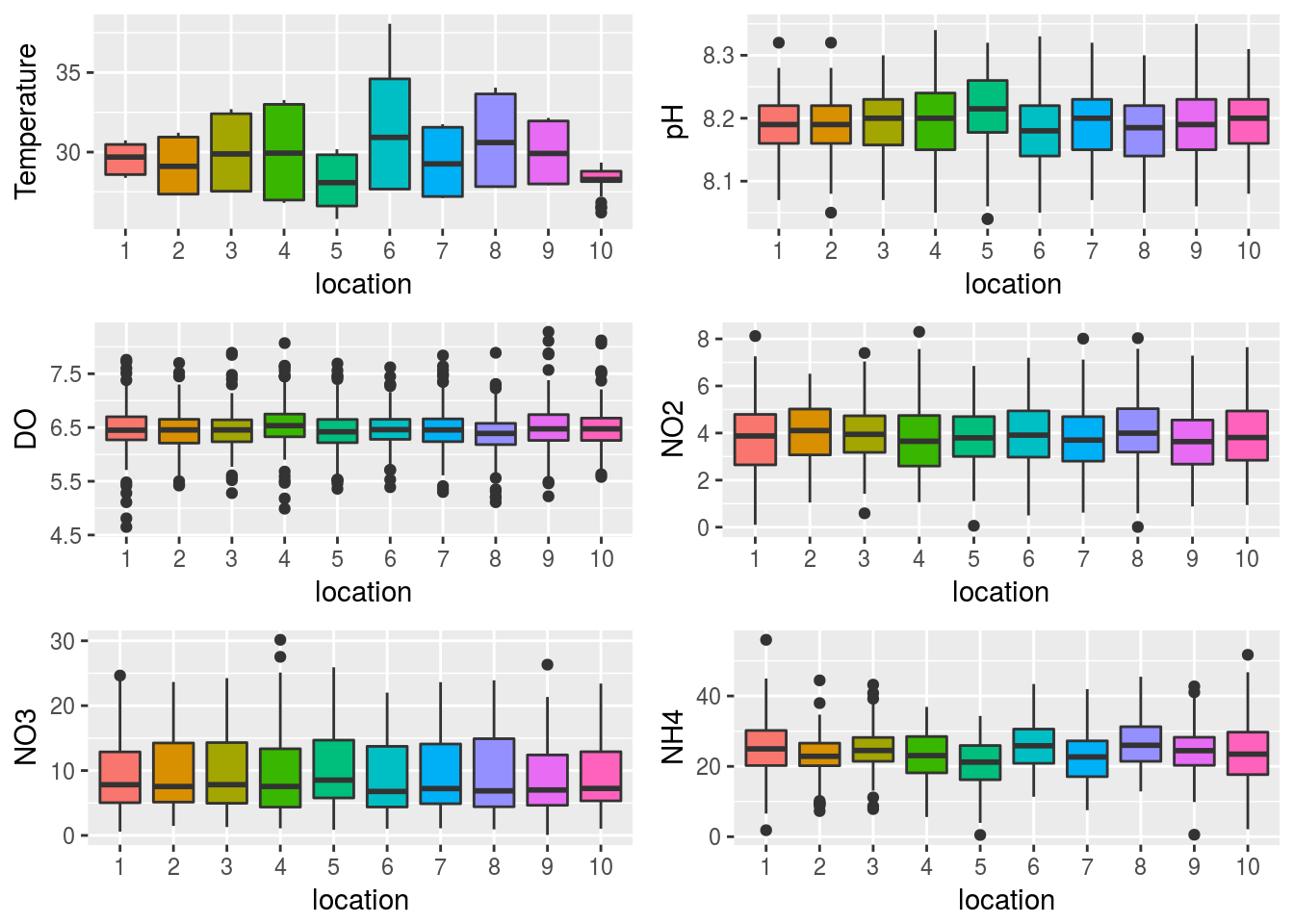
grid.arrange(grobs=plots[7:12],nrow=3,ncol=2)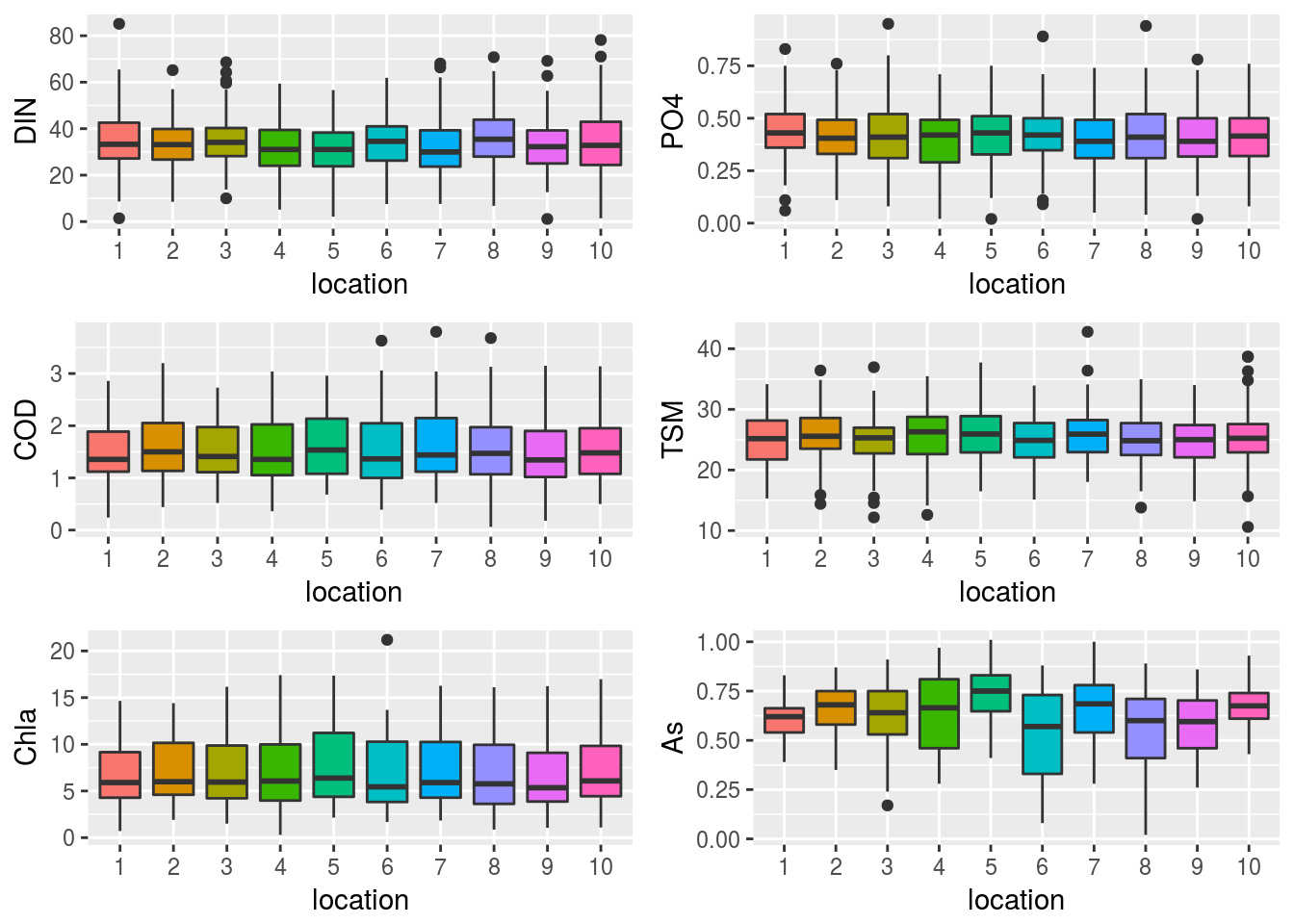
grid.arrange(grobs=plots[13:17],nrow=3,ncol=2)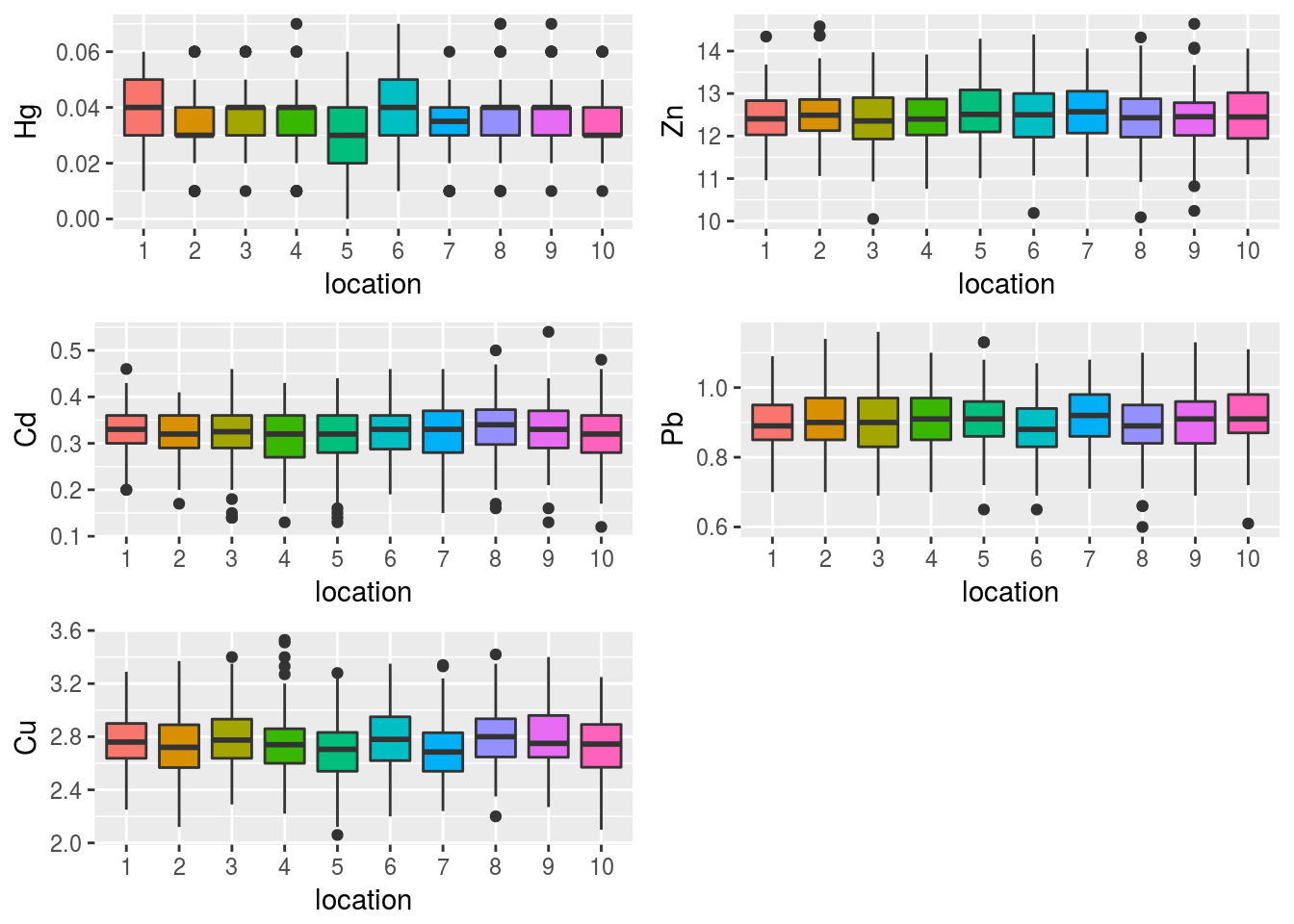
Fijémonos con detalle en la primera de estas gráficas (la correspondiente a la temperatura):
costa$location=factor(costa$location)
ggplot(costa,aes(x=location,y=Temperature,fill=location)) +geom_boxplot()+guides(fill=FALSE)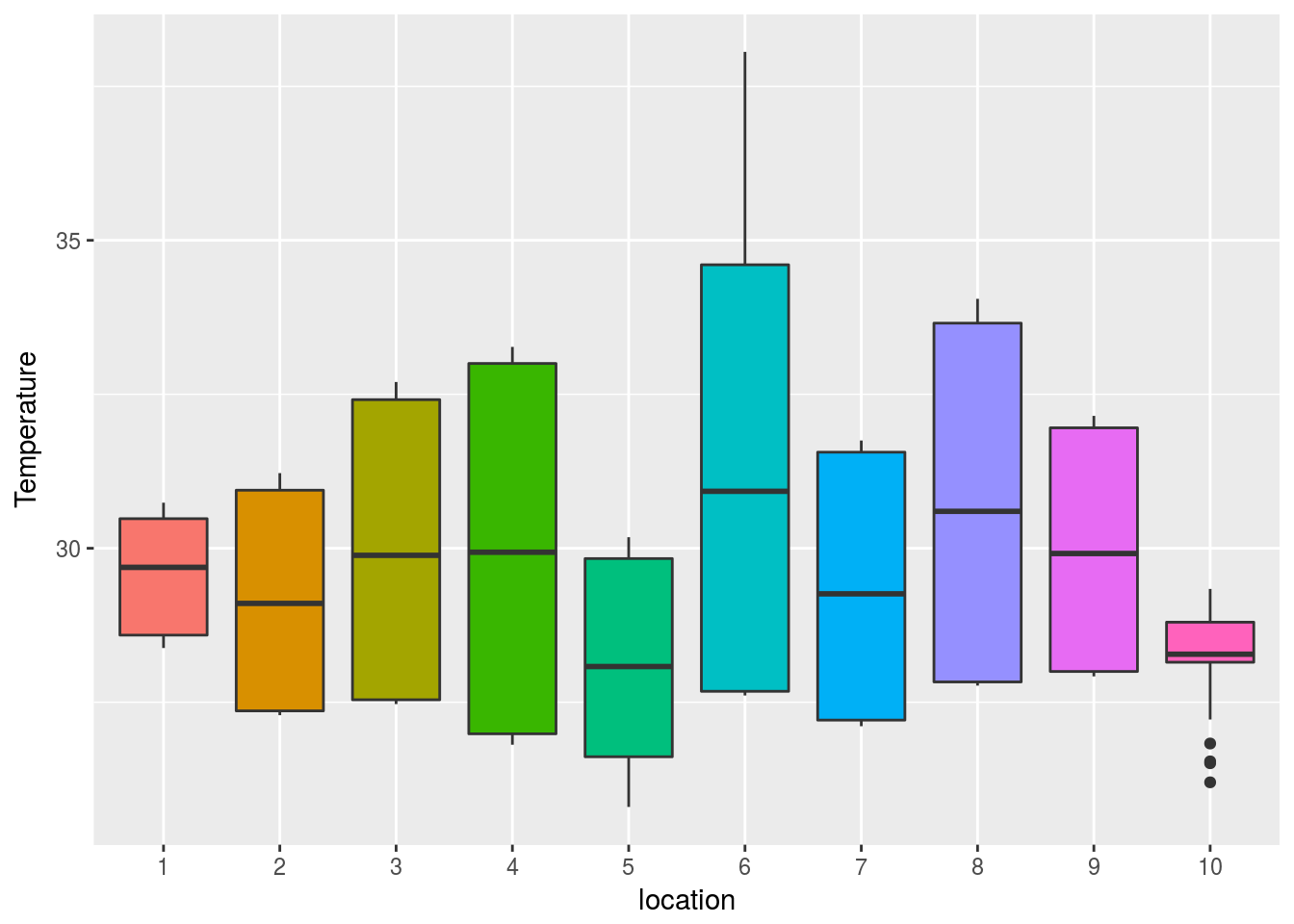
El gráfico indica que aparentemente las localizaciones 5 y 10 presentan una temperatura más baja que el resto. La tabla siguiente nos proporciona los valores medios (y sus correspondientes desviaciones típicas) de la temperatura en cada sitio:
library(pander)
pander(aggregate(Temperature~location,costa,
function(x) sprintf("%.2f (%.2f)",mean(x),sd(x))))| location | Temperature |
|---|---|
| 1 | 29.57 (0.94) |
| 2 | 29.17 (1.81) |
| 3 | 29.98 (2.45) |
| 4 | 29.98 (3.02) |
| 5 | 28.17 (1.66) |
| 6 | 31.29 (3.67) |
| 7 | 29.36 (2.17) |
| 8 | 30.74 (2.92) |
| 9 | 29.98 (1.99) |
| 10 | 28.38 (0.55) |
Al igual que en el caso anterior podemos preguntarnos si las diferencias que se aprecian entre los valores medios de temperatura se deben a que existen diferencias reales en temperatura entre los distintos lugares, o si simplemente dichas diferencias pueden atribuirse al azar del muestreo.
Efecto combinado de localización y temperatura.
Los gráficos que mostramos a continuación revelan el comportamiento conjunto de cada variable frente a la localización y el mes del año en que se ha evaluado:
plots=lapply(1:17,function(k) {
ggplot(subset(costaRes,variable==vbles[k]),
aes(x=location,y=value,fill=month)) +
geom_boxplot() + ylab(vbles[k]) + ggtitle(vbles[k])
})
for (k in 1:17) print(plots[[k]])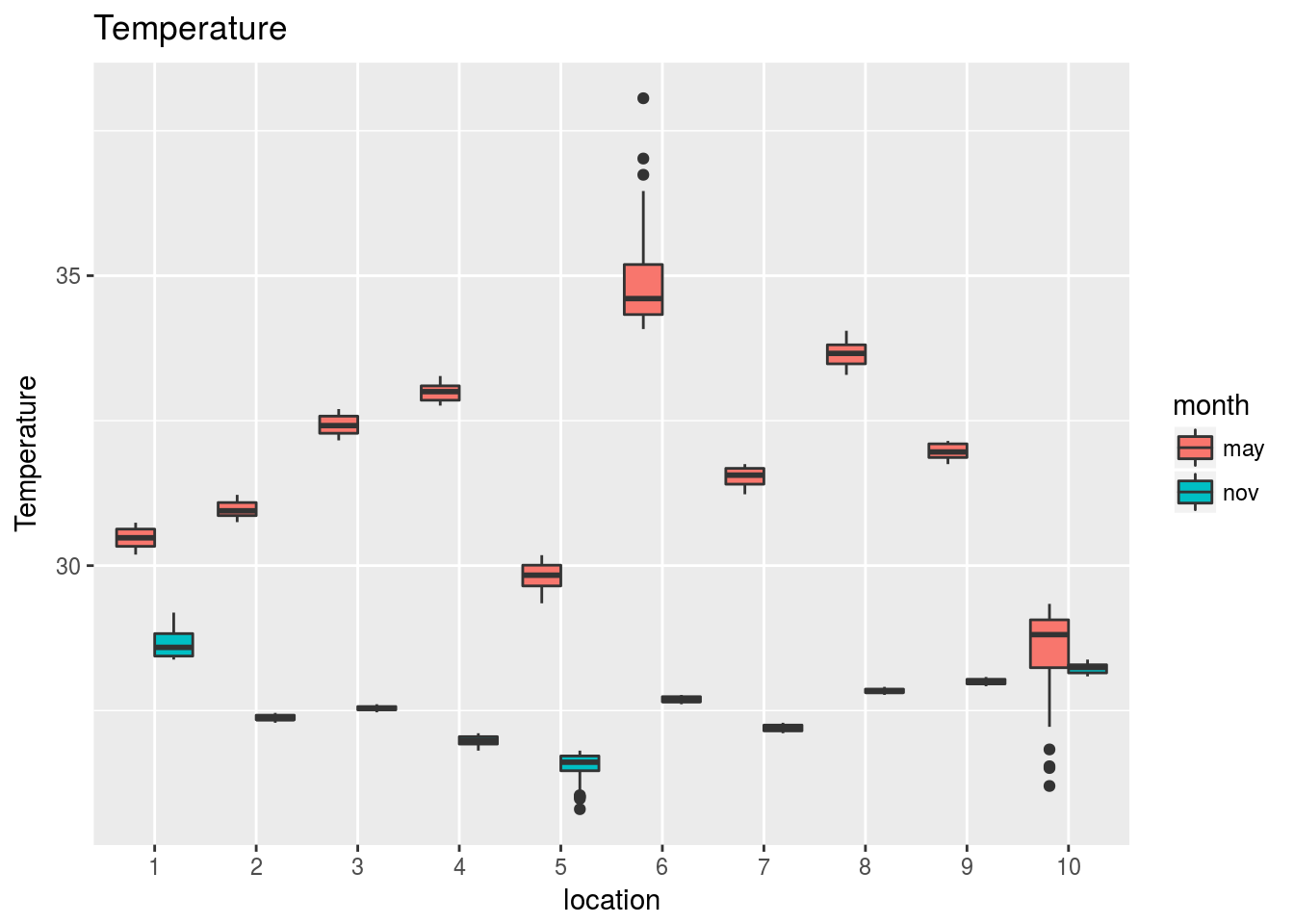
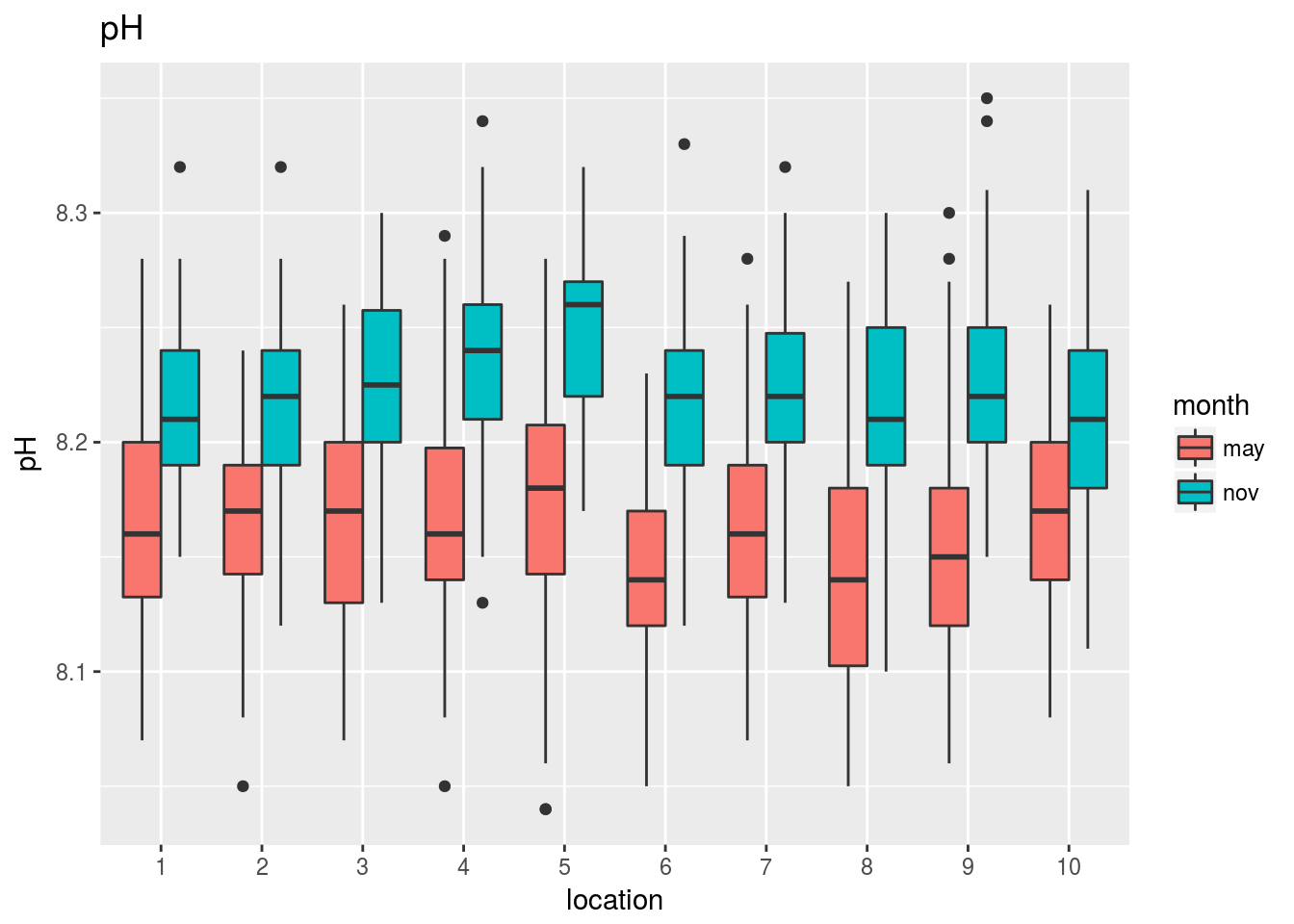
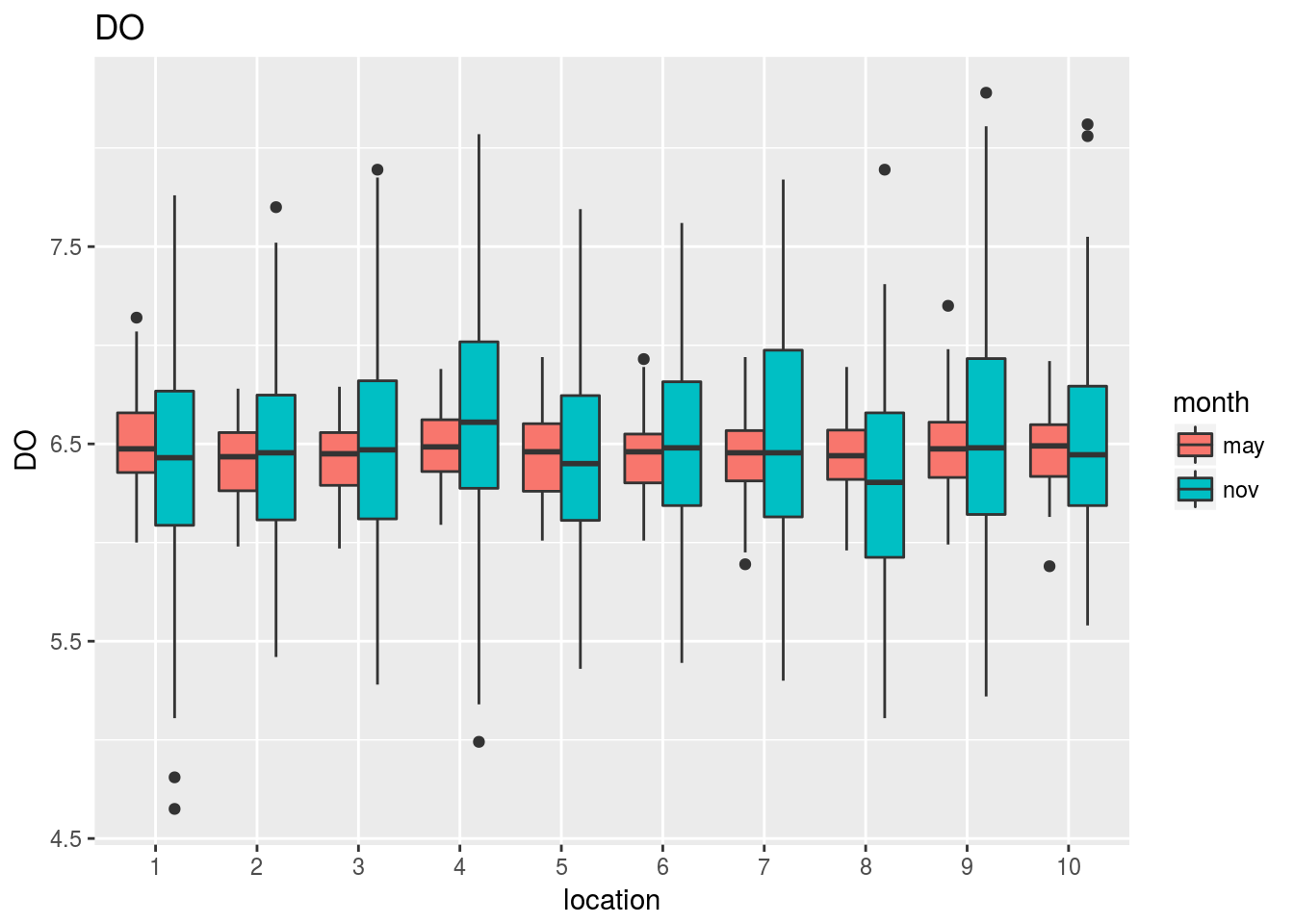
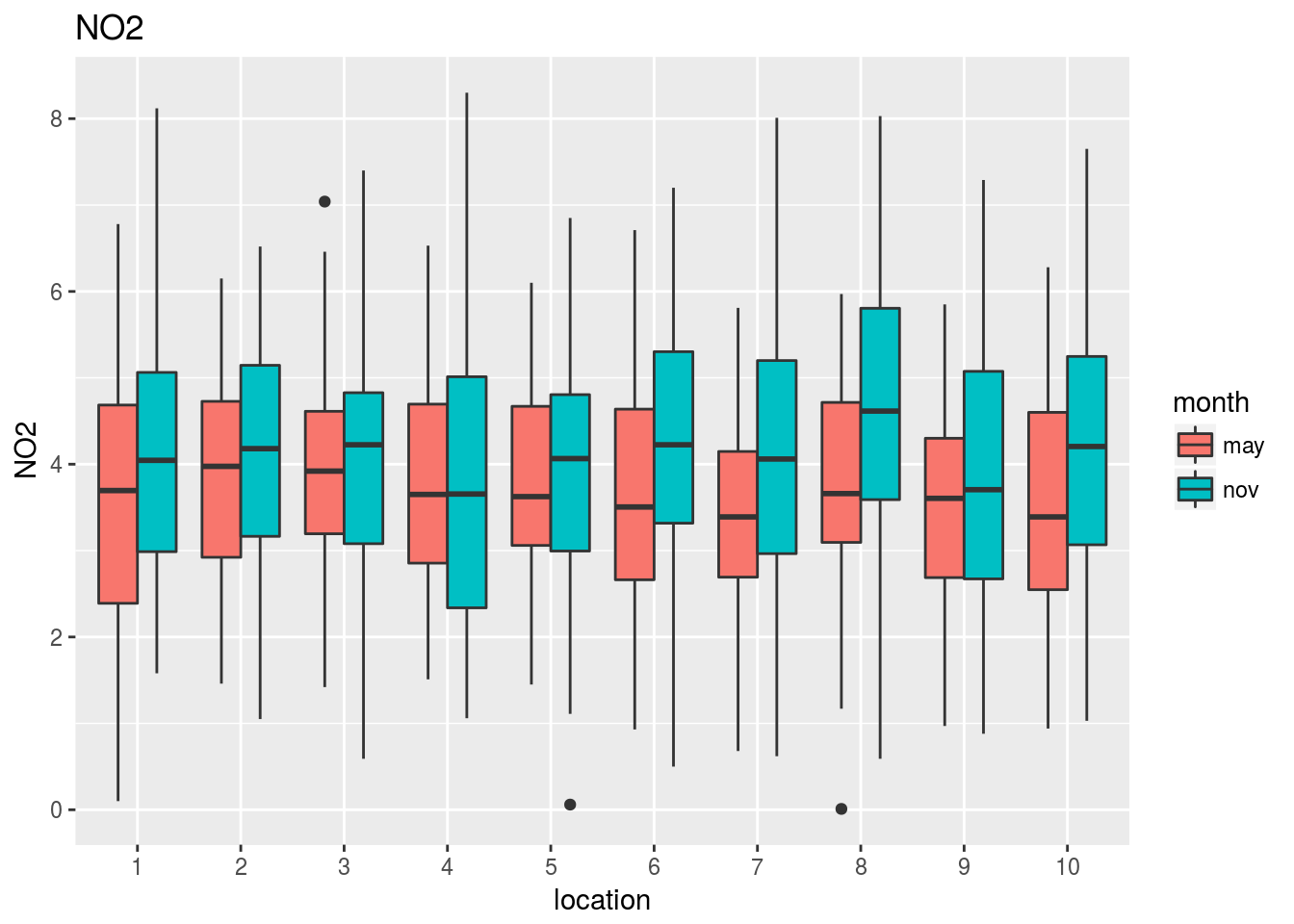
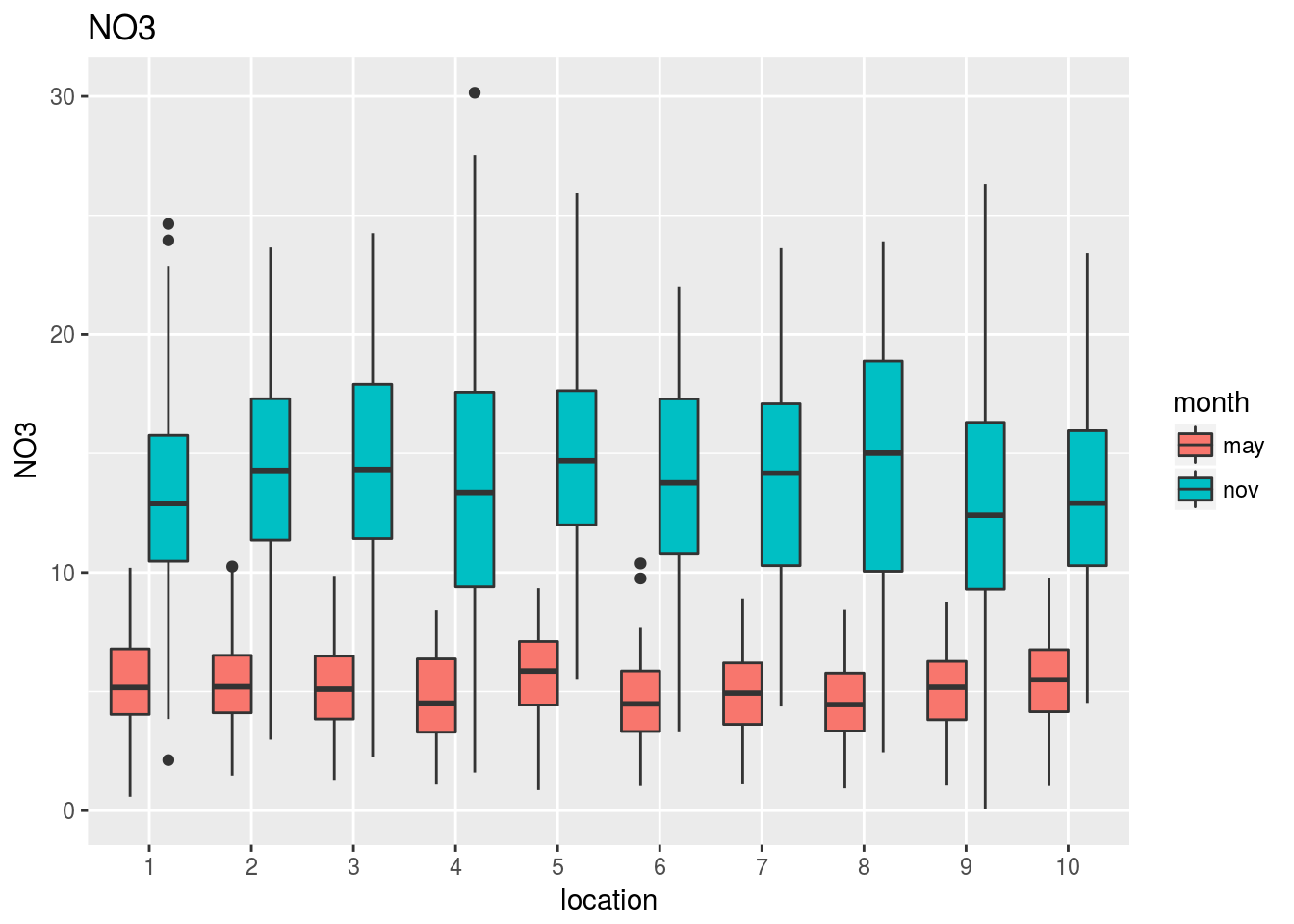
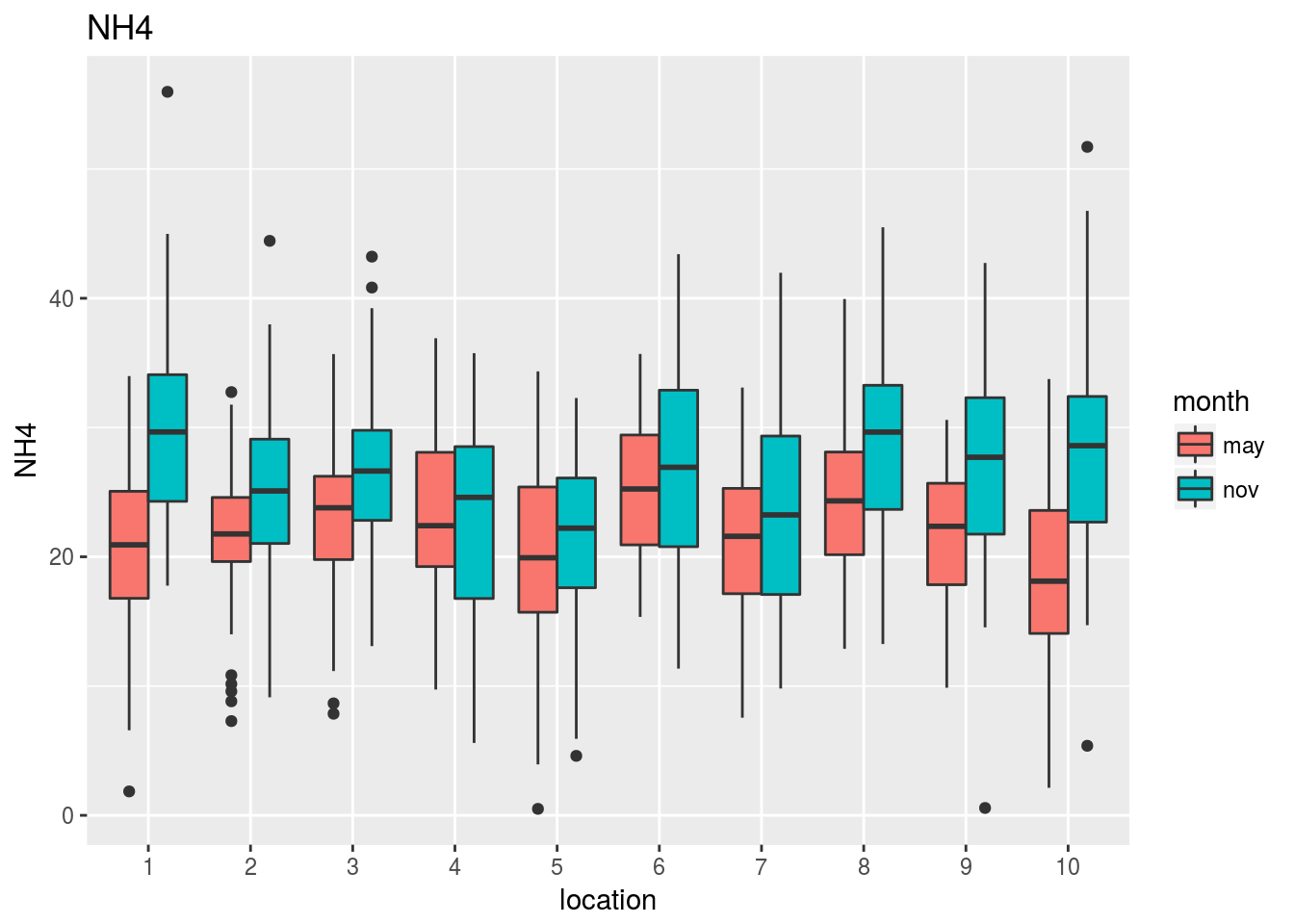
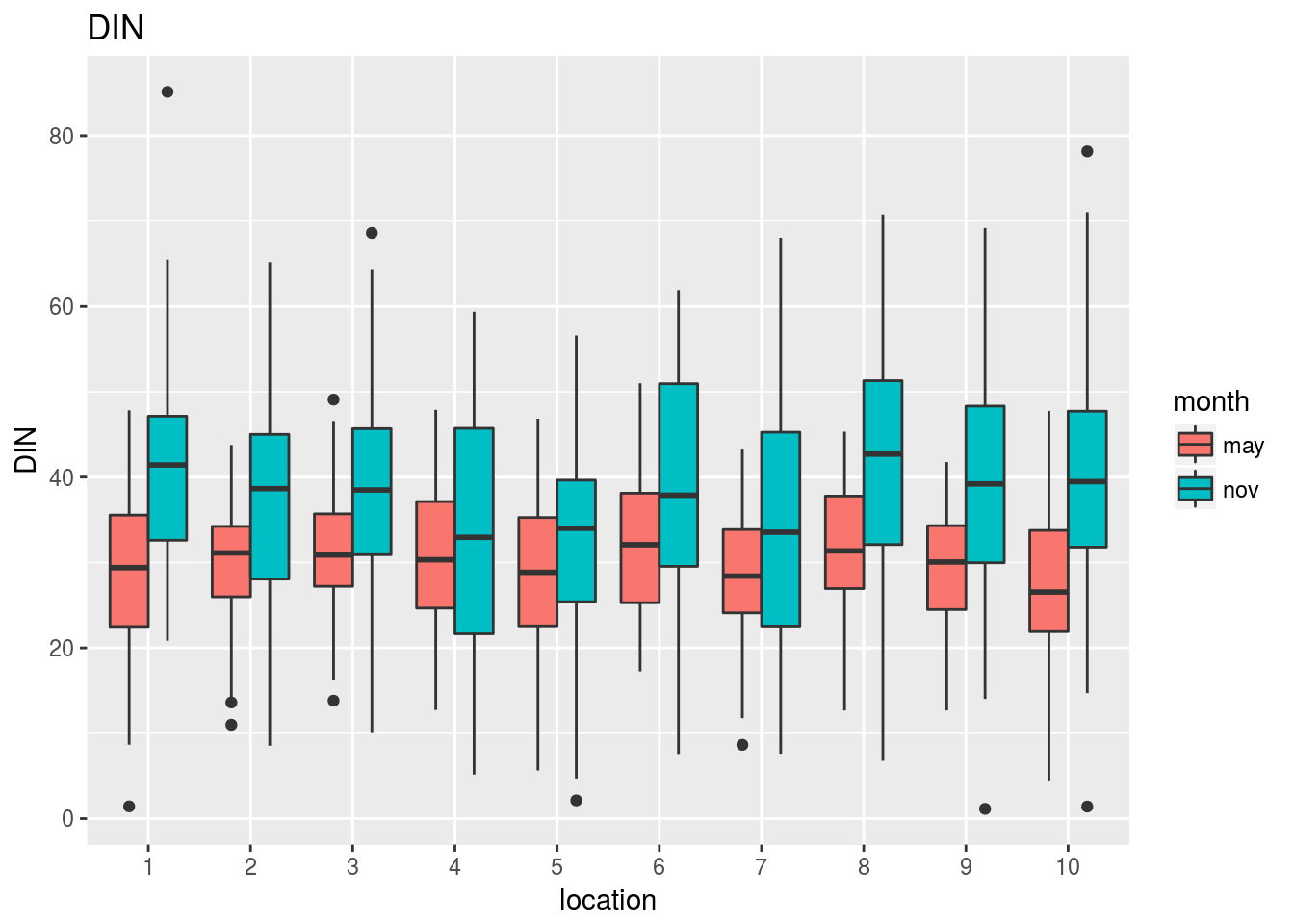
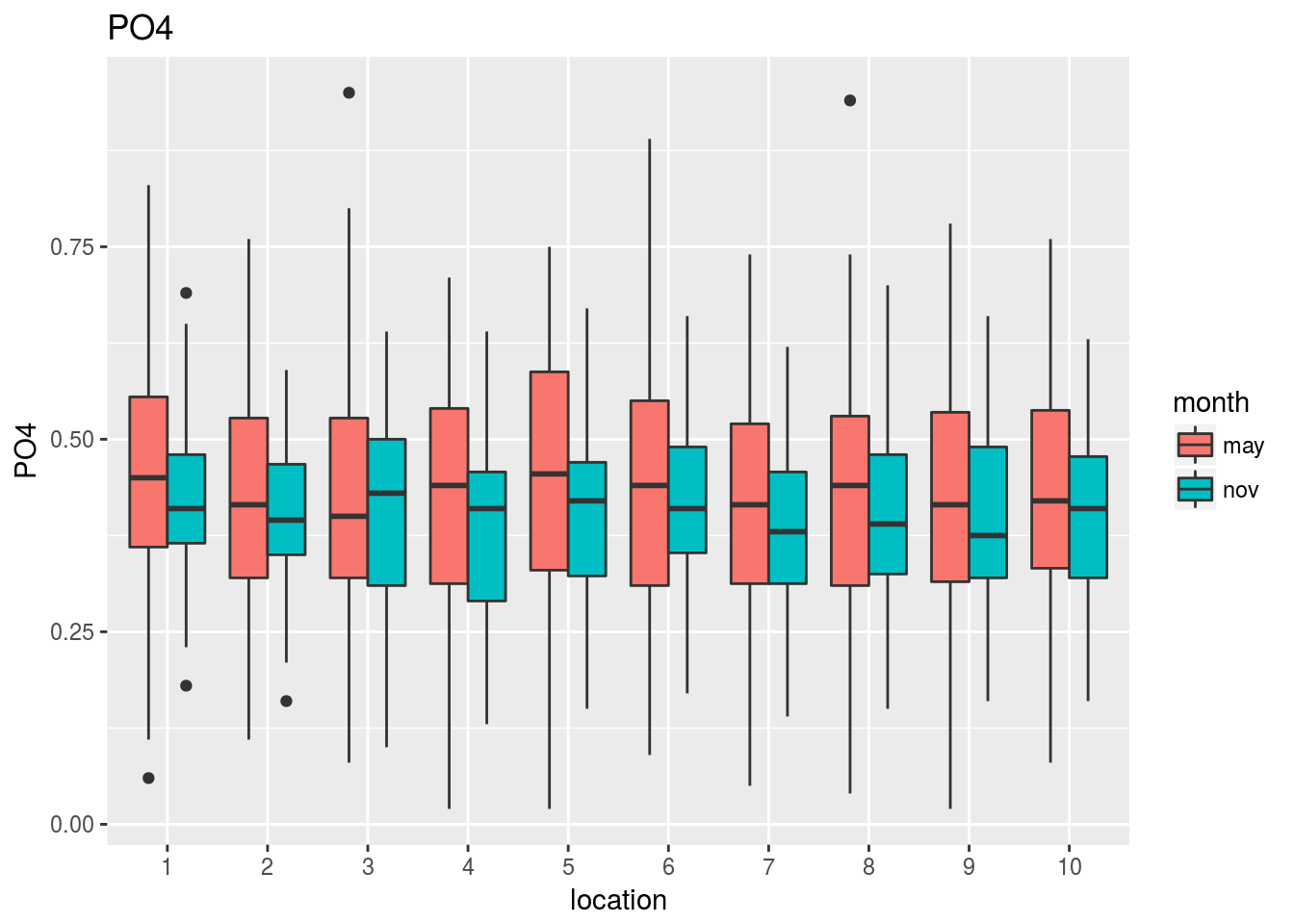
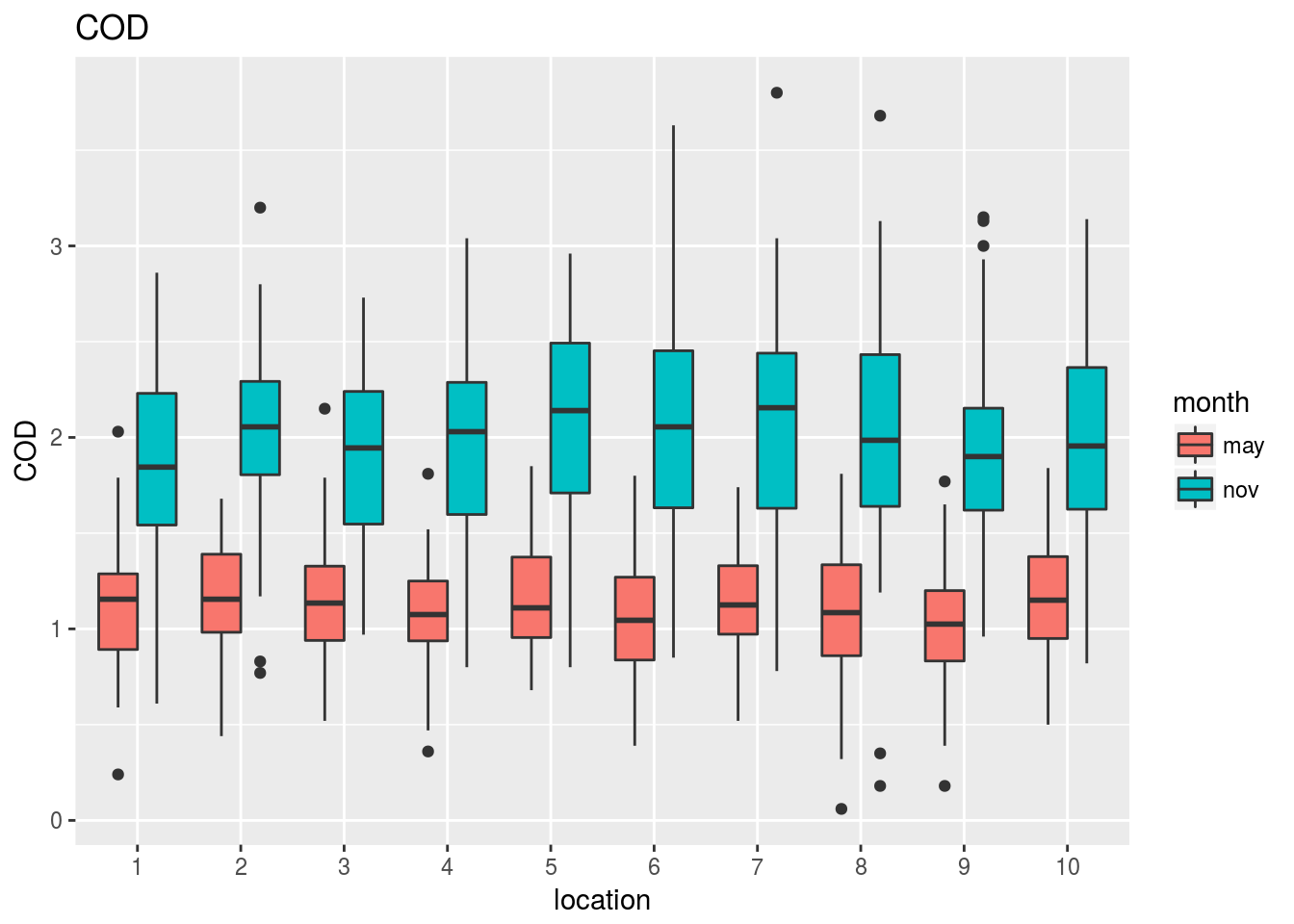
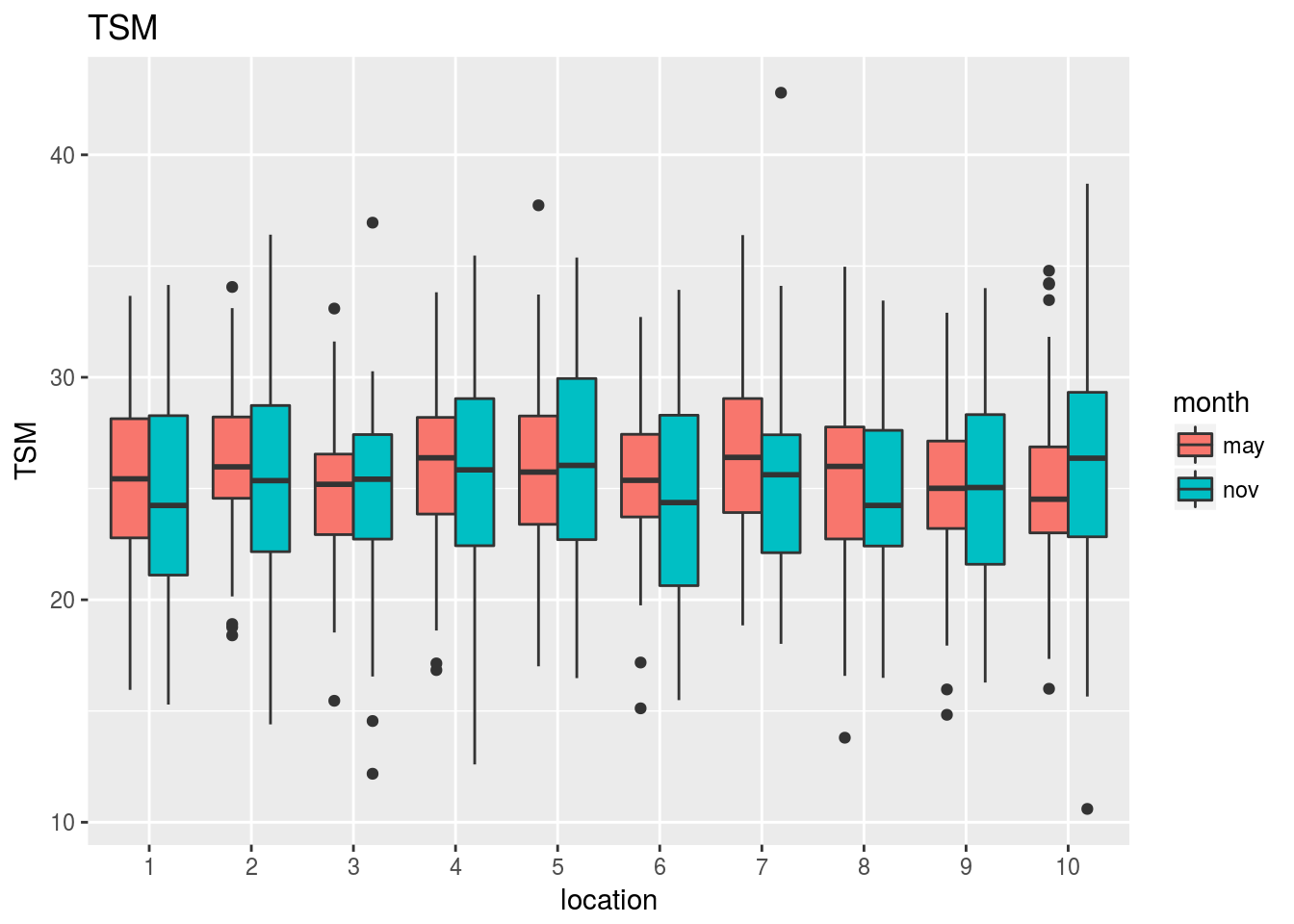
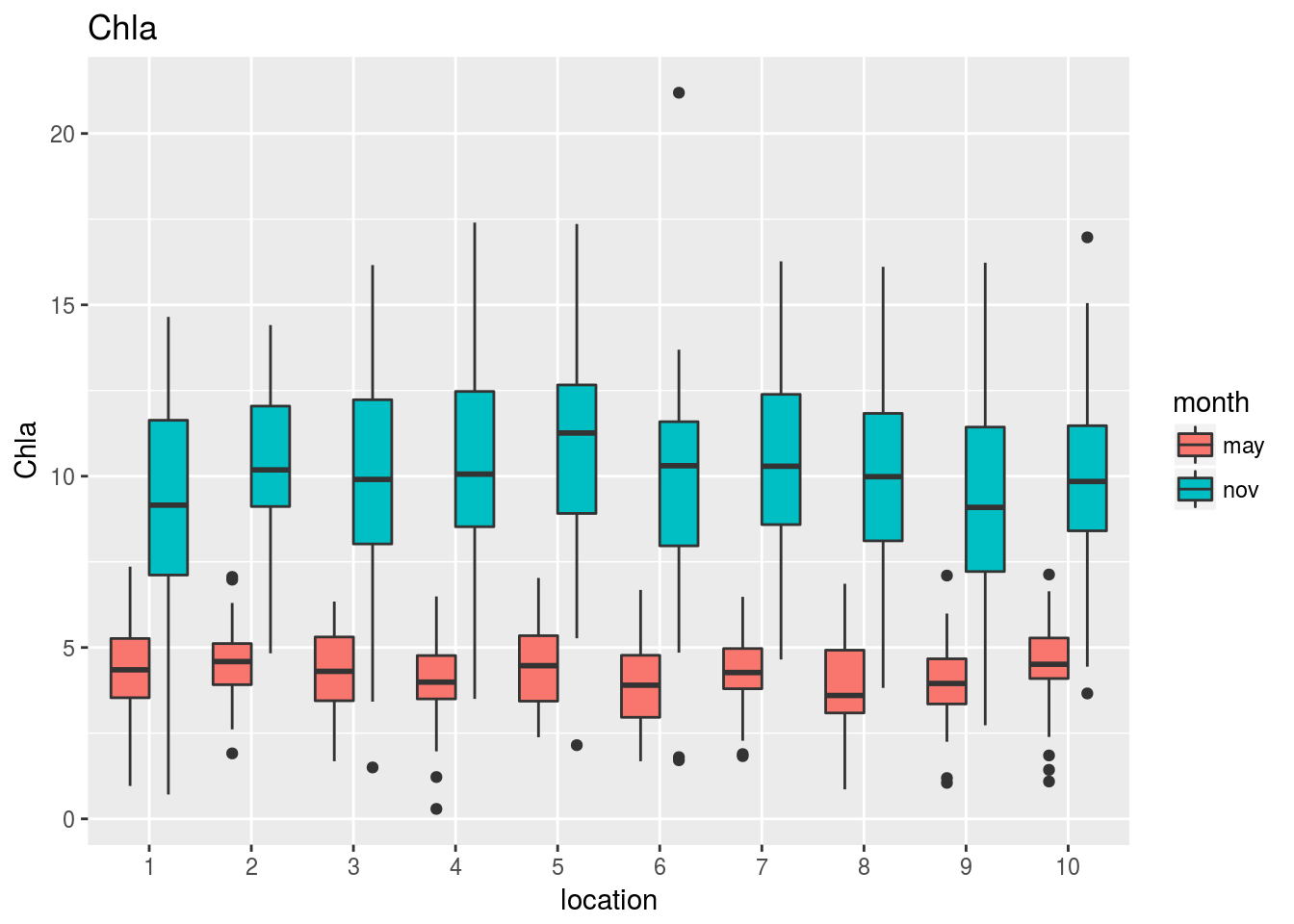
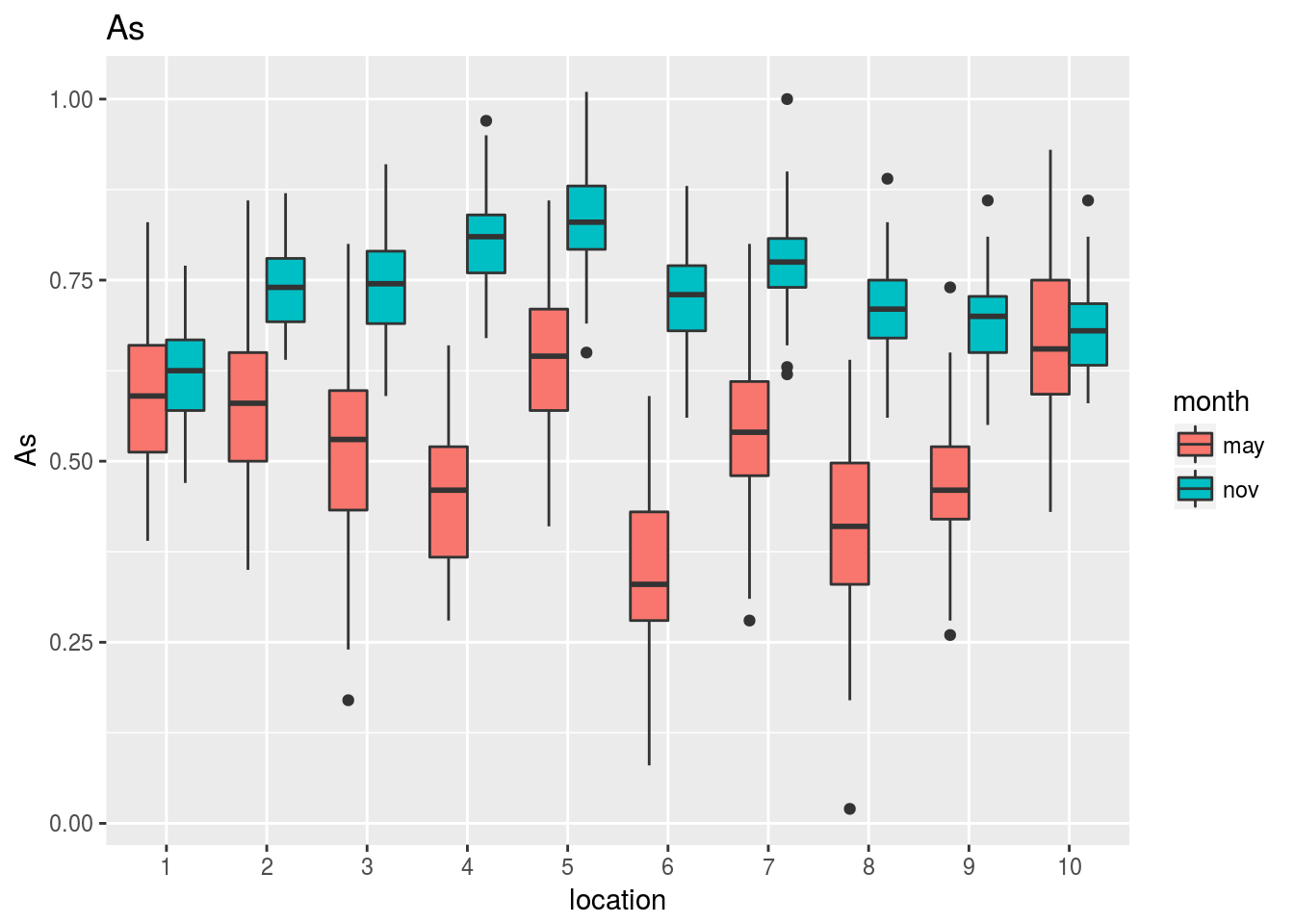
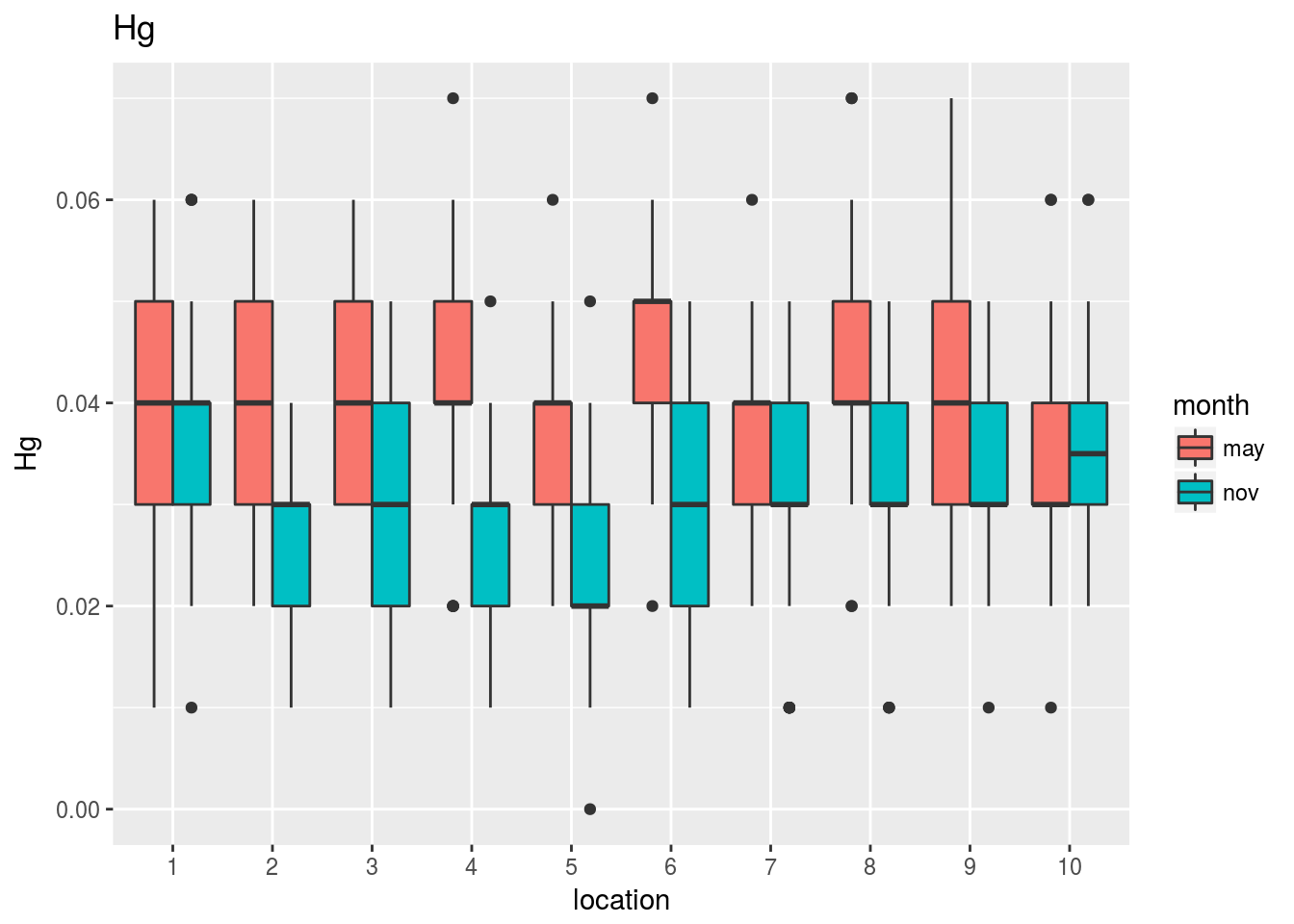
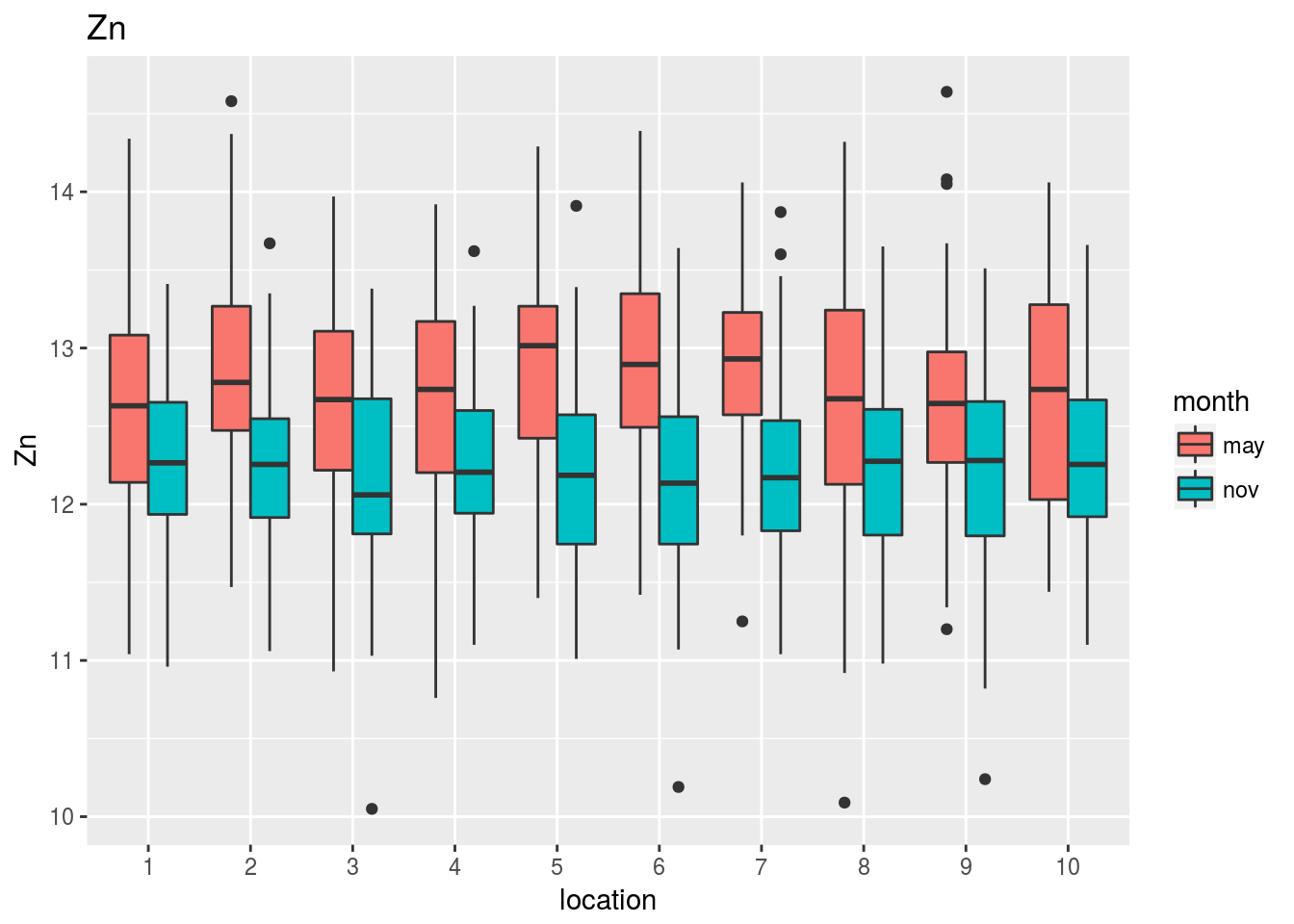
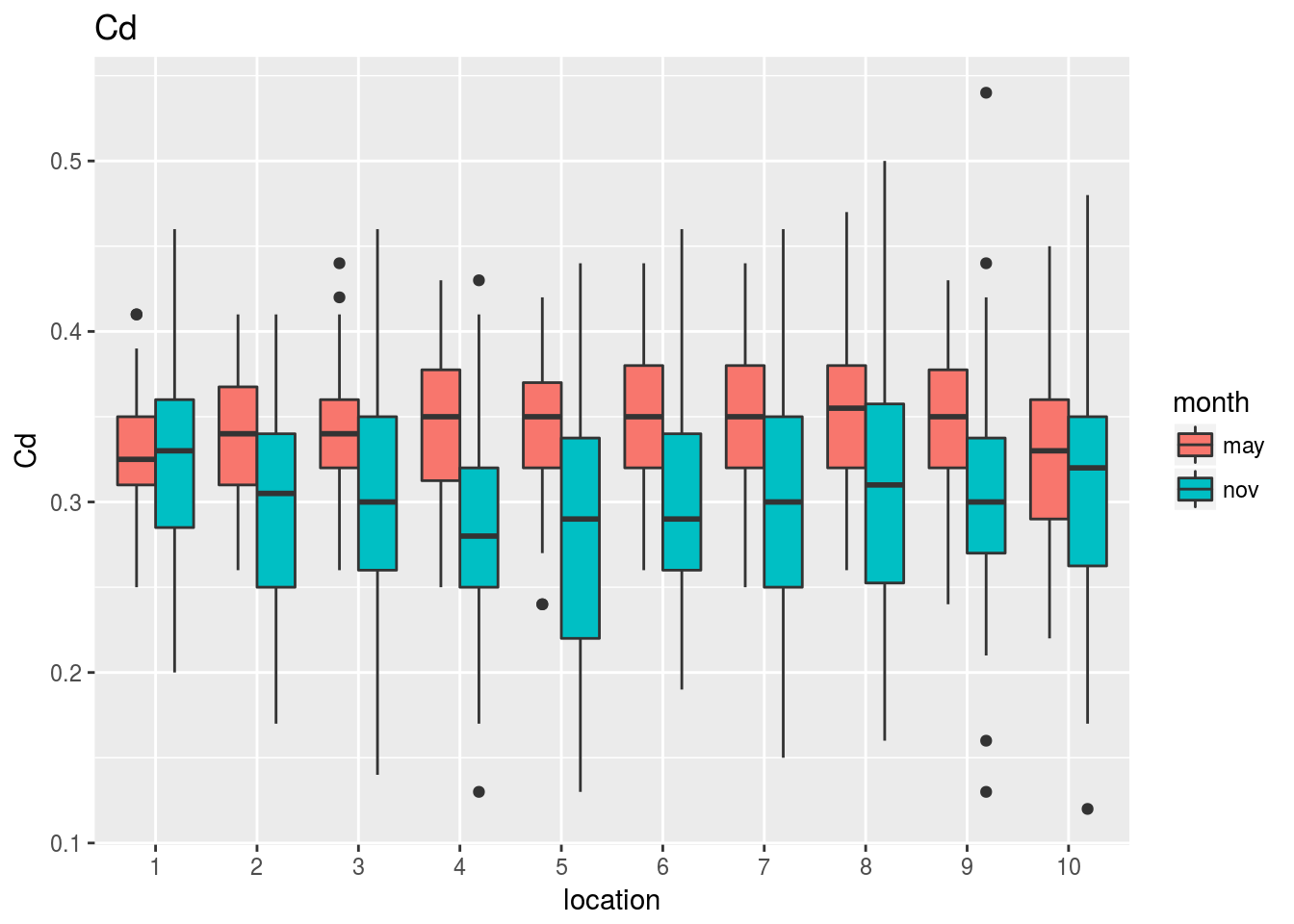
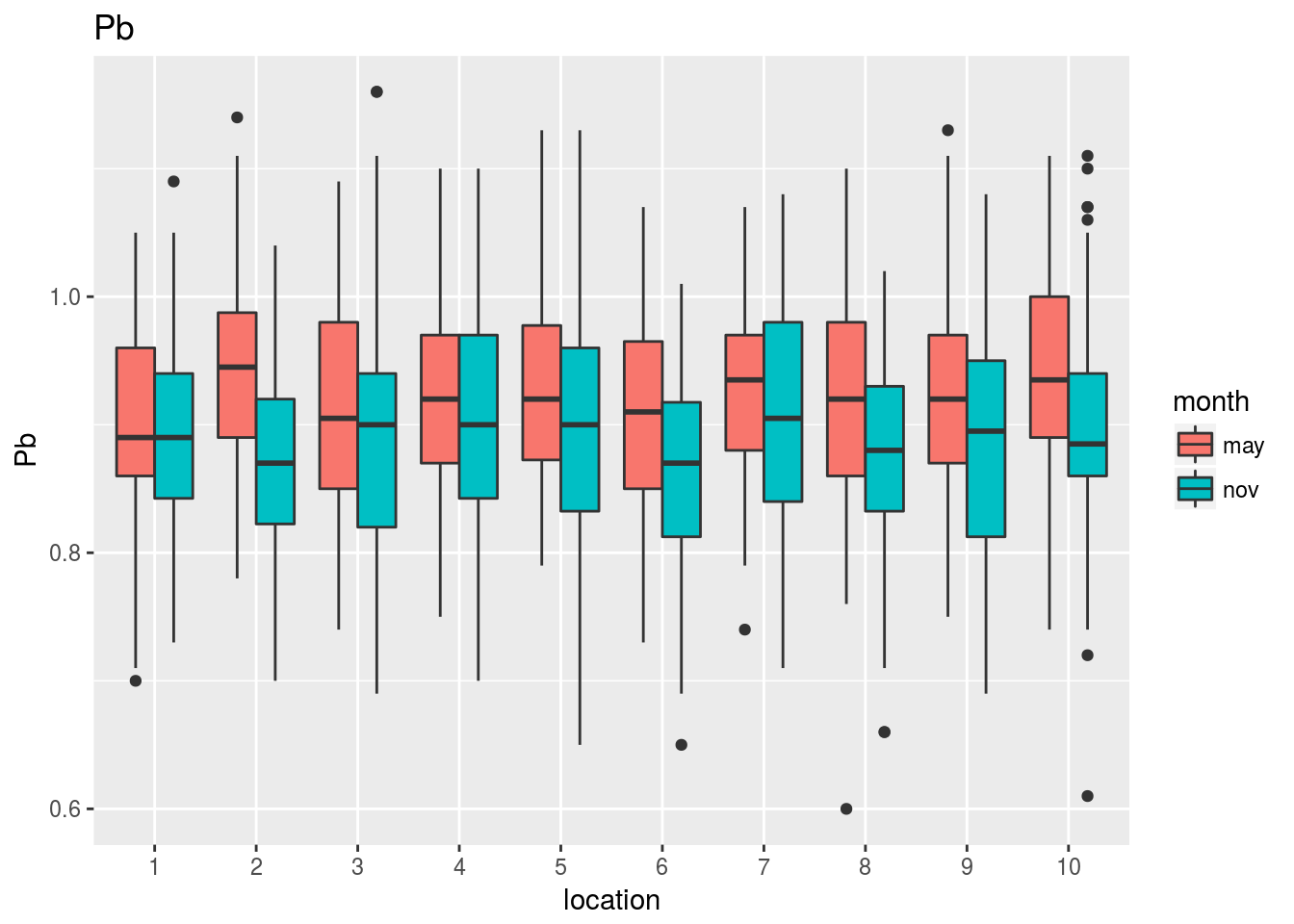
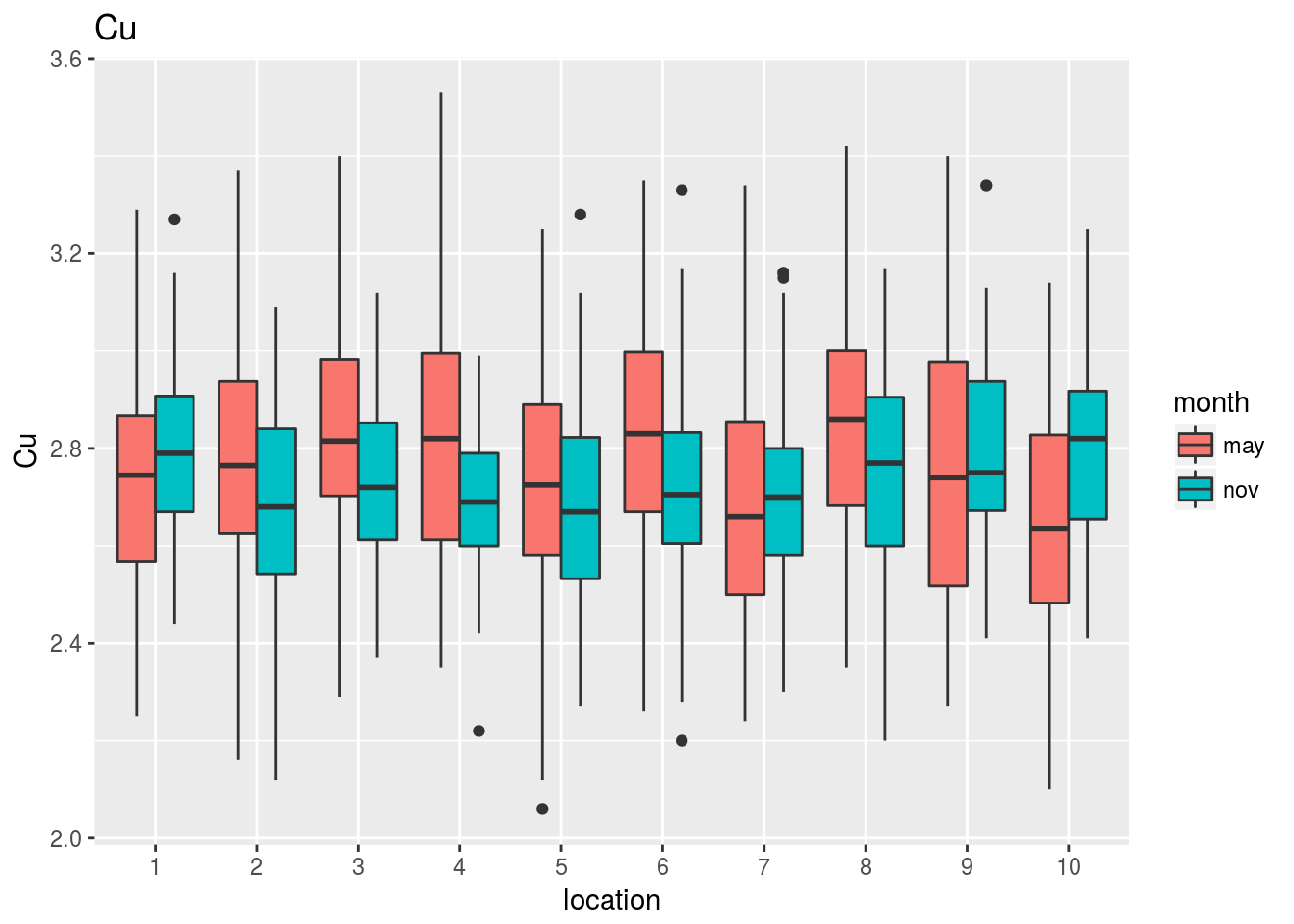
Observando estos gráficos vemos, por ejemplo, que:
La clorofila mantiene aproximadamente la misma diferencia entre mayo y noviembre cualquiera que sea la localización que se considere. En noviembre la concentración es siempre mayor que en mayo, y no parece haber diferencias en concentración entre localizaciones.
El PO4 no muestra diferencias notables entre noviembre y mayo ni, aparentemente tampoco entre estaciones.
La temperatura o el As muestran cambios tanto en la diferencia entre mayo y noviembre como en las diferencias entre estaciones.
En el Pb no parece haber grandes diferencias entre mayo y noviembre, aunque sí parece que hay algunas localizaciones con mayores niveles de Pb que otras.
Predicción de la clorofila.
La siguiente tabla nos muestra las correlaciones entre la clorofila y el resto de variables durante el mes de mayo (la clorofila está en la fila 11):
pander(cor(may[,1:17])[11,])| Temperature | pH | DO | NO2 | NO3 | NH4 | DIN | PO4 | COD | TSM |
|---|---|---|---|---|---|---|---|---|---|
| -0.1712 | -0.1988 | -0.3646 | 0.6013 | 0.589 | 0.09777 | 0.3751 | 0.2481 | 0.7621 | 0.2822 |
| Chla | As | Hg | Zn | Cd | Pb | Cu |
|---|---|---|---|---|---|---|
| 1 | 0.2892 | -0.07581 | 0.2253 | -0.05337 | 0.1098 | -0.1231 |
Asimismo las correlaciones durante el mes de noviembre son
pander(cor(nov[,1:17])[11,])| Temperature | pH | DO | NO2 | NO3 | NH4 | DIN | PO4 | COD | TSM |
|---|---|---|---|---|---|---|---|---|---|
| -0.1618 | -0.2013 | -0.2842 | 0.5037 | 0.4825 | 0.0438 | 0.2785 | 0.1685 | 0.7318 | 0.2924 |
| Chla | As | Hg | Zn | Cd | Pb | Cu |
|---|---|---|---|---|---|---|
| 1 | 0.3211 | -0.057 | 0.2372 | -0.1032 | 0.0547 | -0.1327 |
Vemos que en ambos meses las correlaciones más altas de la clorofila (mayores que 0.3) se producen con las variables DO, NO2, NO3, DIN y COD. La siguiente función calcula el modelo de regresión para predecir el nivel de clorofila a partir de estas variables durante el mes de mayo:
lm(Chla~DO+NO2+NO3+DIN+COD, data=may)##
## Call:
## lm(formula = Chla ~ DO + NO2 + NO3 + DIN + COD, data = may)
##
## Coefficients:
## (Intercept) DO NO2 NO3 DIN
## 2.15892 -0.15585 0.26820 0.24840 -0.06251
## COD
## 2.41981Lo mismo durante el mes de noviembre:
lm(Chla~DO+NO2+NO3+DIN+COD, data=nov)##
## Call:
## lm(formula = Chla ~ DO + NO2 + NO3 + DIN + COD, data = nov)
##
## Coefficients:
## (Intercept) DO NO2 NO3 DIN
## 3.2815 -0.2515 0.6206 0.1928 -0.1069
## COD
## 3.6175Obviamente los valores son distintos: ¿efecto del azar en el muestreo o diferencias significativas entre meses?
La siguiente función nos permite hacer un gráfico tridimensional de la relación de la clorofila con otras dos variables:
library(rgl)
with(may,plot3d(COD,NO2,Chla))
Si hubiésemos decidido incluir en el modelo aquellas variables con dan con la clorofila una correlación mayor que 0.25, deberíamos incluir también PO4, TSM y As. En tal caso, los modelos ajustados habrían sido:
lm(Chla~DO+NO2+NO3+DIN+COD+PO4+TSM+As, data=may)##
## Call:
## lm(formula = Chla ~ DO + NO2 + NO3 + DIN + COD + PO4 + TSM +
## As, data = may)
##
## Coefficients:
## (Intercept) DO NO2 NO3 DIN
## 0.21484 0.04846 0.25775 0.36422 -0.05421
## COD PO4 TSM As
## 2.46472 -2.25512 0.03759 -0.42333lm(Chla~DO+NO2+NO3+DIN+COD+PO4+TSM+As, data=nov)##
## Call:
## lm(formula = Chla ~ DO + NO2 + NO3 + DIN + COD + PO4 + TSM +
## As, data = nov)
##
## Coefficients:
## (Intercept) DO NO2 NO3 DIN
## 0.84065 -0.02439 0.50971 0.31505 -0.08178
## COD PO4 TSM As
## 3.62315 -9.00244 0.09521 -0.01337Nuevamente tenemos valores estimados distintos en ambos modelos, y además los valores estimados en cada variable son distintos de los que se estimaron cuando usamos menos variables. Podemos plantearnos la cuestión ¿Cuál es el mejor modelo para predecir la clorofila? ¿Sobra alguna de las variables que hemos considerado? ¿Habría que añadir alguna de las variables que hemos dejado fuera?
Una visión global de todos los problemas anteriores.
De modo resumido, los problemas anteriores parecen presentar problemas bastante diferentes:
Dada una variable continua (e.g. la temperatura) y una o varias variables categóricas (el mes, la localización) ¿existen diferencias en el valor medio de la variable continua según los distintos niveles de las variables categóricas? Este problema se conoce habitualmente como de análisis de la varianza
Dadas dos variables continuas (e.g. DIN frente a NO2) y una variable categórica (el mes), ¿la relación entre DIN y NO2 es fija o cambia con el mes? . Podríamos incluso extender la pregunta: ¿cambia con la localización? ¿cambia según la combinación de mes y localización? Estos problemas se conocen como de análisis de la covarianza.
Dadas una variable continua (respuesta), y otra (o varias) variables continuas (explicativas) ¿Cuál es el modelo lineal que mejor permite predecir la respuesta en función de las variables explicativas? ¿Como seleccionar las mejores variables explicativas? Estos problemas entran dentro del marco de la regresión lineal
Aunque todos estos problemas parecen tener una naturaleza muy diferente, desde el punto de vista teórico se encuadran dentro del mismo marco conceptual: el modelo lineal
El modelo lineal
Definición
Un vector de variables aleatorias \(\mathbf{Y}= \left(y_1,y_2,\dots, y_n\right)'\) sigue un modelo lineal si:
\[\mathbf{Y} \cong \mathbf{N_n} \left(\mathbf{X} \beta, \mathbf{I_n}\sigma \right)\]
donde:
- \(\mathbf{X}=\left(\begin{array}{ccccc} 1 & x_{11} & x_{21} & \ldots & x_{p1}\\ 1 & x_{12} & x_{22} & \vdots & x_{p2}\\ 1 & \vdots & \vdots & \ddots\\ 1 & x_{1n} & x_{2n} & \ldots & x_{pn} \end{array}\right)\) es una matriz de dimensión \(n\times p\), cuya columna \(j\)-ésima (j>2) contiene los valores observados en la variable \(X_j\).
- \(\beta=\left(\begin{array}{c} \beta_{0}\\ \beta_{1}\\ \vdots\\ \beta_{p} \end{array}\right)\) es un vector de parámetros \(p\)-dimensional.
- \(\mathbf{I_{n}}\) es la matriz identidad de orden \(n\).
De manera alternativa y equivalente, el modelo lineal puede expresarse como: \[\mathbf{Y=}\beta_{0}+\beta_{1}X_{1}+\beta_{2}X_{2}+\dots+\beta_{p}X_{p}+\varepsilon,\,\,\,\,\,\,\varepsilon\approx N \left(0,\sigma\right)\]
siendo las \(X_j\) las columnas de la matrix \(\mathbf{X}\) anterior.
la variable \(\mathbf{Y}\) se conoce habitualmente como variable respuesta y las variables \(\mathbf{X}\) como variables explicativas o predictoras.
Bajo esta definición general se engloban los modelos clásicos de:
Regresión: La variable \(\mathbf{Y}\) y las \(\mathbf{X}\) son continuas
Análisis de la varianza: La variable \(\mathbf{Y}\) es continua y las \(\mathbf{X}\) son categóricas.
Análisis de la covarianza: La variable \(\mathbf{Y}\) es continua y entre las \(\mathbf{X}\) hay variables categóricas y variables continuas
Cuando en este modelo la distribución normal se cambia por otra distribución y la relación entre la \(\mathbf{Y}\) y las \(\mathbf{X}\) es lineal a través de una función de enlace \(f\), nos encontramos ante los modelos lineales generalizados:
Regresión logística Binaria: La variable \(\mathbf{Y}\) es dicotómica y las \(X_i\) pueden ser categóricas y/o continuas. La distribución de \(\mathbf{Y}\) es binomial, \[\mathbf{Y} \cong \mathbf{B} \left(N,f(\mathbf{X} \beta)\right)\]
Regresión de Poisson: La variable \(\mathbf{Y}\) es una variable de recuento y las \(X_i\) pueden ser categóricas y/o continuas. La distribución de \(\mathbf{Y}\) es de Poisson, \[\mathbf{Y} \cong \mathbf{P} \left(f(\mathbf{X} \beta)\right)\]
Cuando la relación entre la \(\mathbf{Y}\) y las \(\mathbf{X}\) es de la forma: \[Y=f_{1} \left(X_{1}\right)+f_{2}\left(X_{2}\right)+\dots f_{p}\left(X_{p}\right)+\varepsilon\] nos encontramos ante los modelos aditivos generalizados (GAM).
Estimación del modelo lineal
El modelo lineal para predecir (o explicar) la variable respuesta \(\mathbf{Y}\) en función de la matriz de variables explicativas \(\mathbf{X}\) puede estimarse por dos métodos:
Mínimos cuadrados: Este método puede aplicarse siempre y consiste en encontrar los valores de los parámetros \(\beta_i\) que minimizan la distancia entre los valores observados y los predichos.
Máxima verosimilitud: Este método sólo es de aplicación cuando las observaciones son independientes y las distancias de los puntos al modelo (residuos) siguen una distribución normal. Cuando estas condiciones se cumplen, el método de máxima verosimilitud y el de mínimos cuadrados producen los mismos estimadores.
Sólo en el caso de que los residuos del modelo sean normales e independientes puede llevarse a cabo adecuadamente la inferencia clásica sobre los parámetros del modelo.
Los estimadores de los parámetros, utilizando cualquiera de los dos métodos anteriores, se obtienen mediante:
\[ \hat{\beta} = \left(X^{t}X\right)^{-1}X^{t}Y\]
\[\hat{\sigma}^{2}=\frac{1}{n-p}\sum_{i=1}^{n}\left(y_{i}-\hat{y_{i}}\right)^{2}=\frac{1}{n-p}\sum_{i=1}^{n}\left(y_{i}-X_{i}\hat{\beta}\right)^{2}=\frac{1}{n-p}\left(\mathbf{Y-X}\hat{\beta}\right)^{t}\left(\mathbf{Y-X}\hat{\beta}\right)\]
La matriz de covarianzas de los estimadores de \(\beta_i\) es:
\[\textrm{Var}\left(\hat{\beta}\right)=\hat{\sigma}^{2}\left(X^{t}X\right)^{-1}\]
La raiz cuadrada de la diagonal de esta matriz nos proporciona los errores estándar de los \(\beta_i\), que permiten construir contrastes e intervalos de confianza para estos parámetros. Concretamente, si llamamos \(\hat{\sigma}_{\beta_{i}}\) al error estándar del parámetro \(\beta_i\), puede obtenerse un intervalo de confianza a nivel \(1-\alpha\) para dicho parámetro mediante: \[\left[\hat{\beta}_{i}\pm t_{n-p,1-\alpha/2}\hat{\sigma}_{\beta_{i}}\right]\]
Descomposición de la variabilidad.
La variabilidad total presente en la variable respuesta puede descomponerse en la parte explicada por las variables explicativas, más la variabilidad residual:
VARIABILIDAD TOTAL = VARIABILIDAD EXPLICADA + VARIABILIDAD RESIDUAL
donde:
VARIABILIDAD TOTAL: VT = \(\sum_{i=1}^{n}\left(y_{i}-\overline{y}\right)^{2}\)
VARIABILIDAD EXPLICADA: VE = \(\sum_{i=1}^{n}\left(\hat{y}_{i}-\overline{y}\right)^{2}\)
VARIABILIDAD RESIDUAL: VR = \(\sum_{i=1}^{n}\left(y_{i}-\hat{y}_{i}\right)^{2}\)
Es decir:
\[\sum_{i=1}^{n}\left(y_{i}-\overline{y}\right)^{2}=\sum_{i=1}^{n}\left(\hat{y}_{i}-\overline{y}\right)^{2}+\sum_{i=1}^{n}\left(y_{i}-\hat{y}_{i}\right)^{2}\]
A partir de esta descomposición es posible definir coeficientes para medir la calidad de ajuste del modelo (coeficientes de determinación y de determinación corregido), y construir un contraste (conocido como análisis de la varianza del modelo) que permite decidir si las variables explicativas realmente son explicativas; dicho de otra forma, permite decidir si la variabilidad explicada por dichas variables representa una proporción significativa de la variabilidad total presente en el modelo.
Coeficiente de determinación \(R^2\)
Este coeficiente mide simplemente la proporción de la variabilidad en la variable respuesta que es explicada (o está determinada) por las variables explicativas. Cuanto más se aproxime su valor a 1, mejor es el modelo; cuanto más se aproxime a cero tanto peor:
\[R^2=\frac{VE}{VT}\]
Coeficiente de determinación corregido (o ajustado) \(\tilde{R}^2\)
El coeficiente de determinación que se acaba de definir presenta el problema práctico de que su valor se va aproximando a 1 a medida que se añaden variables explicativas \(X_i\) al modelo, aunque la aportación de cada una sea irrelevante. Interesa por ello redefinir el coeficiente de determinación de forma que se vea “penalizado” si su valor aumenta por introducir muchas variables que no expliquen nada en lugar de pocas variables que expliquen mucho. Para ello observemos en primer lugar que el coeficiente de determinación \(R^2\) puede expresarse como: \[R^{2}=\frac{VE}{VT}=1-\frac{VR}{VT}\]
Para penalizar el exceso de variables explicativas, el coeficiente de determinación corregido se define de manera similar mediante:
\[\tilde{R}^{2}=1-\frac{VR/\left(n-p\right)}{VT/\left(n-1\right)}=1-\frac{n-1}{n-p}\frac{VR}{VT}\]
De esta forma, el término \(\frac{n-1}{n-p}\) es mayor que 1 (tanto mayor cuanto mayor sea el número de variables explicativas \(p\)). Dicho término multiplica a la proporción de varianza no explicada \(\frac{VR}{VT}\), por lo cual su efecto es “inflar” la variabilidad no explicada y por tanto reducir la variabilidad explicada artificialmente por un exceso de variables. Si después de estos ajustes el coeficiente de determinación así obtenido es próximo a 1, ello significaría que aún penalizando la introducción de variables, la varibilidad explicada es alta, y por tanto las variables consideradas puede considerarse que tienen buena capacidad explicativa o predictiva de la variable respuesta.
Análisis de la varianza del modelo lineal
Se denomina así al contraste:
\[H_0:\beta_{1}=\beta_{2}=\dots=\beta_{p}=0\]
que de forma matricial puede expresarse como:
\[H_0:\left(\begin{array}{ccccc} 0 & 0 & 0 & \ldots & 0\\ 0 & 1 & 0 & \ldots & 0\\ 0 & 0 & 1 & \ldots & 0\\ \vdots & \vdots & \vdots & \ddots & \vdots\\ 0 & 0 & 0 & \ldots & 1 \end{array}\right)\left(\begin{array}{c} \beta_{0}\\ \beta_{1}\\ \beta_{2}\\ \vdots\\ \beta_{p} \end{array}\right)=\left(\begin{array}{c} 0\\ 0\\ 0\\ \vdots\\ 0 \end{array}\right)\]
Este contraste tiene como objetivo decidir si, consideradas conjuntamente, las variables \(X_i\) tienen capacidad explicativa/predictiva sobre la variable respuesta \(\mathbf{Y}\). En caso de acepar la hipótesis nula, ello significaría que el posible efecto de las \(X_i\) sobre la \(\mathbf{Y}\) puede atribuirse simplemente al azar; en caso de rechazarla, ello implicaría que las \(X_i\) (todas o algunas de estas variables) tienen realmente efecto sobre la \(\mathbf{Y}\), mas alla de lo que cabría esperar por azar.
Este es un contraste lineal de la forma:
\[H_0:\mathbf{A\beta}=0\]
Cuando \(H_0\) es cierta el test estadístico:
\[F=\frac{1}{q\hat{\sigma}^{2}}\hat{\beta}^{t}\mathbf{A}^{t}\left(\mathbf{A}\left(\mathbf{X}^{t}\mathbf{X}\right)^{-1}\mathbf{A}^{t}\right)^{-1}\mathbf{A\hat{\beta}}\]
(siendo \(q\) el número de filas de la matriz \(\mathbf{A}\)) sigue una distribución \(F\) de Fisher con \(q\) y \(n-p\) grados de libertad. El p-valor del contraste es
\[p-valor=P\left(F_{q,n-p}>F_{obs}\right)=1-P\left(F_{q,n-p} \le F_{obs}\right)\]
siendo \(F_{obs}\) el valor observado del test estadístico \(F\).
En el caso particular del contraste que hemos planteado inicialmente, la matriz \(\mathbf{A}\) es: \[\mathbf{A=}\left(\begin{array}{ccccc} 1 & 0 & 0 & \ldots & 0\\ 0 & 1 & 0 & \ldots & 0\\ 0 & 0 & 1 & \ldots & 0\\ \vdots & \vdots & \vdots & \ddots & \vdots\\ 0 & 0 & 0 & \ldots & 1 \end{array}\right)\] en cuyo caso puede probarse que:
\[F_{obs}=\frac{VE/(p-1)}{VR/(n-p)}\]
lo que nos indica que este contraste está, en definitiva, comparando la variabilidad explicada por el modelo con la variabilidad residual (tras ajustar por el número de casos y por el número de variables); sólo si la variabilidad explicada es significativamente mayor que la residual es cuando el valor de \(F_obs\) resultaría lo suficientemente grande para rechazar \(H_0\).
Nótese que este test simplemente decide si hay o no efecto de las \(X_i\) sobre la \(\mathbf{Y}\). No nos dice nada sobre la intensidad de dicho efecto, que deberá cuantificarse en términos del coeficiente de determinación.
Predicción
Una vez ajustado un modelo lineal, puede utilizarse para realizar predicciones sobre los valores de la variable respuesta \(Y\). Hay dos tipos de predicciones que se pueden realizar:
Predicción del valor medio de \(Y\) dado el valor de \(X\).
Predicción del valor individual de \(Y\) dado el valor de \(X\).
En ambos casos la predicción es análoga; si \(X=x_0\) el valor predicho es:
\[\hat{Y} = X\hat{\beta} = x_0\hat{\beta}\]
Aunque el valor predicho sea el mismo, el error estándar difiere entre ambas predicciones. De manera intuitiva, es obvio que el valor medio tiene menor dispersión que los valores individuales y, en efecto, así ocurre. La varianza de la predicción del valor medio dado que \(X=x_0\) es:
\[Var\left(\left.\hat{y}\right|X=x_{0}\right)=\hat{\sigma}^{2}x_{0}\left(X^{t}X\right)^{-1}x_{0}^{t} \]
y la varianza de la predicción del valor individual dado que \(X=x_0\) es:
\[Var\left(\left.\widetilde{y}\right|X=x_{0}\right)=\hat{\sigma}^{2}\left(1+x_{0}\left(X^{t}X\right)^{-1}x_{0}^{t}\right)\]
Los errores estándar son, respectivamente, las raíces cuadradas de estas varianzas. Llamando:
\[ \hat{y}_0 = x_0 \hat{\beta}\]
podemos construir intervalos de confianza para las predicciones mediante:
- Intervalo de confianza a nivel \(1-\alpha\) para la predicción del valor medio de \(Y\) cuando \(X=x_0\):
\[\left[\hat{y}_{0}\pm t_{n-p,1-\alpha/2}\hat{\sigma}\sqrt{x_{0}\left(X^{t}X\right)^{-1}x_{0}^{t}}\right]\]
- Intervalo de confianza a nivel \(1-\alpha\) para la predicción del valor individual de \(Y\) cuando \(X=x_0\):
\[\left[\hat{y}_{0}\pm t_{n-p,1-\alpha/2}\hat{\sigma}\sqrt{1+x_{0}\left(X^{t}X\right)^{-1}x_{0}^{t}}\right]\]