class: center, middle, inverse, title-slide .title[ # <br><br>Correlación y Regresión Lineal Simple ] .date[ ### <br>Métodos Estadísticos Avanzados. <br>Grado en Ciencias del Mar ] --- ## Asociación Lineal Se dispone de datos de 100 salmones ([Descarga los datos aquí](http://estadistica-dma.ulpgc.es/estadFCM/datos/salmonArgentino.csv)) capturados en tres zonas costeras de Argentina y Chile. Para cada ejemplar se ha medido su talla (en cm) y su peso (en kg). A continuación se muestran los datos de algunos de los salmones de esta muestra, y la nube de puntos talla-peso: <img src="01-Correlacion_y_regresion_lineal_simple_files/figure-html/unnamed-chunk-1-1.png" style="display: block; margin: auto;" /> --- ## Asociación Lineal Parece razonable modelar la relación talla-peso mediante una recta: <img src="01-Correlacion_y_regresion_lineal_simple_files/figure-html/unnamed-chunk-2-1.png" style="display: block; margin: auto;" /> --- ## Asociación Lineal Esta recta se denomina .blue[ __recta de regresión__] y su ecuación es de la forma: `$$y = b_0 + b_1 x$$` -- Si tomásemos .resalta[otra muestra] de 100 salmones en las mismas localizaciones, podríamos esperar una nube de puntos .resalta[_parecida_], y por tanto unos valores .resalta[_parecidos_] de `\(b_0\)` y `\(b_1\)` -- Si dispusiéramos de datos de la _población_ de salmones podríamos calcular la recta de regresión ajustada a la población: `$$y = \beta_0 + \beta_1 x$$` -- Si nuestra muestra es representativa, `\(b_0\)` es un estimador de `\(\beta_0\)` (.blue[ordenada]) y `\(b_1\)` es un estimador de `\(\beta_1\)` (.blue[pendiente]) -- .resalta[ ¿Cómo estimar `\(\beta_0\)` y `\(\beta_1\)`?, es decir, ¿cómo calculamos `\(b_0\)` y `\(b_1\)` a partir de una muestra de puntos? ] --- ## El modelo de regresión lineal simple La recta de regresión es, en realidad, un modelo aproximado de la relación entre `\(x\)` e `\(y\)`. Para cada sujeto de la población la relación exacta es de la forma: .resalta[ `$$y= \beta_0 + \beta_1 x + \varepsilon$$` ] donde `\(\varepsilon\)` representa la distancia entre el punto observado `\((x,y)\)` y la recta. -- <br> Si podemos asumir que el valor de `\(\varepsilon\)` es un valor aleatorio consecuencia de .blue[ _múltiples_] pequeñas causas .blue[ _independientes_] que se suman y contribuyen a apartar el punto de la recta, por efecto del Teorema Central del Límite es razonable modelar `\(\varepsilon\)` como una variable aleatoria con distribución normal: .resalta[ `$$\varepsilon \approx N\left(0,\sigma_\varepsilon\right)$$` ] --- ## Regresión Lineal: .blue[Estimación por Máxima Verosimilitud] Supondremos que: -- * Se dispone de `\(n\)` observaciones de dos variables `\(\left\{\left(X_{i},Y_{i}\right),\,i=1,\dots,n\right\}\)` -- * Los valores de `\(Y_i\)` se ajustan al modelo: `$$Y_i= \beta_0 + \beta_1 X_i + \varepsilon_i$$` -- * Los valores `\(\varepsilon_i\)` son `\(N(0,\sigma_{\varepsilon})\)` e independientes. -- * Por tanto: + Para cada `\(i=1,\dots, n\)`: `$$Y_{i}\approx N\left(\beta_{0}+\beta_{1}X_{i},\sigma_\varepsilon\right)$$` + Para cada valor `\(X_i=x\)` fijo: `$$E\left[Y\left|X_i=x\right.\right]=\beta_{0}+\beta_{1}x$$` -- .resalta[ Es decir, los valores individuales de `\(Y\)` se distribuyen alrededor la recta `\(y=\beta_0+\beta_1 x\)`, centrados en ella, y con varianza constante `\(\sigma^2_{\varepsilon}\)`. ] --- ## Regresión Lineal: .blue[Estimación por Máxima Verosimilitud] * Como `\(Y_{i}\approx N\left(\beta_{0}+\beta_{1}X_{i},\sigma_\varepsilon\right)\)`, la función de densidad de `\(Y\)` cuando `\(X=x_i\)` es: `$$f_{\beta_{0},\beta_{1},\sigma_\varepsilon}\left(y\left|X=x_{i}\right.\right)=\frac{1}{\sigma_\varepsilon\sqrt{2\pi}}\exp\left(-\frac{1}{2}\left(\frac{y-\left(\beta_{0}+\beta_{1}x_{i}\right)}{\sigma_\varepsilon}\right)^{2}\right)$$` -- * La función de verosimilitud cuando se ha observado la muestra `\(\left\{\left(x_{i},y_{i}\right),\,i=1,\dots,n\right\}\)` es entonces: `$$L\left(\beta_{0},\beta_{1},\sigma_\varepsilon\right)=\prod_{i=1}^{n}f_{\beta_{0},\beta_{1},\sigma_\varepsilon}\left(y_{i}\right)={\small \left(\frac{1}{\sigma_\varepsilon\sqrt{2\pi}}\right)^{n}\exp\left(-\frac{1}{2}\sum_{i=i}^{n}\left(\frac{y_{i}-\left(\beta_{0}+\beta_{1}x_{i}\right)}{\sigma_\varepsilon}\right)^{2}\right)}$$` -- * Tomando logaritmos se obtiene la log-verosimilitud: `$$\ell\left(\beta_{0},\beta_{1},\sigma_\varepsilon\right)=-n\log\left(\sigma_\varepsilon\right)-n\log\left(\sqrt{2\pi}\right)-\frac{1}{2\sigma_\varepsilon^{2}}\sum_{i=i}^{n}\left(y_{i}-\left(\beta_{0}+\beta_{1}x_{i}\right)\right)^{2}$$` --- ## Regresión Lineal: .blue[Estimación por Máxima Verosimilitud] Para obtener los valores de `\(\beta_{0}\)`, `\(\beta_{1}\)` y `\(\sigma_\varepsilon\)` que maximizan la log-verosimilitud derivamos e igualamos a 0: `$${\small \frac{\partial}{\partial\beta_{0}}\ell\left(\beta_{0},\beta_{1},\sigma_\varepsilon\right) =\frac{1}{\sigma_\varepsilon^{2}}\sum_{i=i}^{n}\left(y_{i}-\left(\beta_{0}+\beta_{1}x_{i}\right)\right)=0\Rightarrow\sum_{i=i}^{n}\left(y_{i}-\left(\beta_{0}+\beta_{1}x_{i}\right)\right)=0}$$` -- `$${\small\frac{\partial}{\partial\beta_{1}}\ell\left(\beta_{0},\beta_{1},\sigma_\varepsilon\right) =\frac{1}{\sigma_\varepsilon^{2}}\sum_{i=i}^{n}\left(y_{i}-\left(\beta_{0}+\beta_{1}x_{i}\right)\right)x_{i}=0\Rightarrow\sum_{i=i}^{n}\left(y_{i}-\left(\beta_{0}+\beta_{1}x_{i}\right)\right)x_{i}=0}$$` -- `$${\small\frac{\partial}{\partial\sigma_\varepsilon}\ell\left(\beta_{0},\beta_{1},\sigma_\varepsilon\right) =-\frac{n}{\sigma_\varepsilon}+\frac{1}{\sigma_\varepsilon^{3}}\sum_{i=i}^{n}\left(y_{i}-\left(\beta_{0}+\beta_{1}x_{i}\right)\right)^{2}=0\Rightarrow\sum_{i=i}^{n}\left(y_{i}-\left(\beta_{0}+\beta_{1}x_{i}\right)\right)^{2}=n\sigma_\varepsilon^{2}}$$` <br> <center> .red[ __(Ecuaciones normales de la regresión)__]</center> --- ## Regresión Lineal: .blue[Estimación por Máxima Verosimilitud] De la primera ecuación se obtiene: `$$\sum\limits _{i=1}^{n}{(y_{i}-\beta_{0}-\beta_{1}x_{i})}=0\Rightarrow\sum\limits _{i=1}^{n}y_{i}-\sum\limits _{i=1}^{n}\beta_{0}-\sum\limits _{i=1}^{n}{\beta_{1}x_{i}}=0\Rightarrow$$` -- `$$\Rightarrow\sum\limits _{i=1}^{n}{y_{i}}-n\beta_{0}-\beta_{1}\sum\limits _{i=1}^{n}{x_{i}}=0\Rightarrow\beta_{0}=\frac{{\sum\limits _{i=1}^{n}{y_{i}}}}{n}-\beta_{1}\frac{{\sum\limits _{i=1}^{n}{x_{i}}}}{n}\Rightarrow$$` -- <br> `$$\Rightarrow\beta_{0}=\bar{y}-\beta_{1}\bar{x}$$` --- ## Regresión Lineal: .blue[Estimación por Máxima Verosimilitud] Sustituyendo en la segunda ecuación: `$$\sum{(y_{i}-\beta_{0}-\beta_{1}x_{i})}x_{i}=0 \Rightarrow \sum\limits _{i=1}^{n}{(y_{i}-\left(\bar{y}-\beta_{1}\bar{x}\right)-\beta_{1}x_{i})}x_{i}=0\Rightarrow$$` -- `$$\sum\limits _{i=1}^{n}\left(y_{i}-\overline{y}\right)x_{i}-\beta_{1}\sum\limits _{i=1}^{n}\left(x_{i}-\overline{x}\right)x_{i}=0 \Rightarrow$$` -- `$$\beta_{1}=\frac{\sum\limits_{i=1}^{n}\left(y_{i}-\overline{y}\right)x_{i}}{\sum\limits _{i=1}^{n}\left(x_{i}-\overline{x}\right)x_{i}}=\frac{\sum\limits _{i=1}^{n}\left(y_{i}-\overline{y}\right)\left(x_{i}-\overline{x}\right)}{\sum\limits _{i=1}^{n}\left(x_{i}-\overline{x}\right)\left(x_{i}-\overline{x}\right)}=\frac{S_{xy}}{S_{x}^{2}}$$` -- <br> __NOTA:__ Se ha usado que `\(\sum\limits _{i=1}^{n}\left(y_{i}-\overline{y}\right)\overline{x}=\sum\limits _{i=1}^{n}\left(x_{i}-\overline{x}\right)\overline{x}=0\)` --- ## Regresión Lineal: .blue[Estimación por Máxima Verosimilitud] Como `\(\beta_{0}=\bar{y}-\beta_{1}\bar{x}\)`: `$$\hat\beta_{0}=\bar{y}-\hat\beta_{1}\bar{x}=\bar{y}-\frac{S_{xy}}{S^2_x}\bar{x}$$` -- Por último, de la tercera ecuación se obtiene: `$$\sigma_\varepsilon^{2}=\frac{1}{n}\sum_{i=i}^{n}\left(y_{i}-\left(\beta_{0}+\beta_{1}x_{i}\right)\right)^{2}$$` -- Sustituyendo `\(\beta_{0}\)` por `\(\overline{y}-\beta_{1}\overline{x}\)`, tras operar y simplificar, queda: `$$\sigma^2_\varepsilon=\frac{1}{n}\left(\sum\limits _{i=1}^{n}\left(y_{i}-\overline{y}\right)^{2}-\beta_{1}^{2}\sum\limits _{i=1}^{n}\left(x_{i}-\overline{x}\right)^{2}\right)=\frac{1}{n}\left(\left(n-1\right)S_{y}^{2}-\beta_{1}^{2}\left(n-1\right)S_{x}^{2}\right)$$` -- y por tanto: `$$\hat\sigma^{2}_\varepsilon=\frac{n-1}{n}\left(S_{y}^{2}-\hat\beta_{1}^{2}S_{x}^{2}\right)=\frac{n-1}{n}\left(S^2_y-\frac{S^2_{xy}}{S^2_x}\right)$$` --- ## Regresión Lineal: .blue[Estimación por Máxima Verosimilitud] En resumen: .resalta[ `$$\hat{\beta}_{1}=\frac{S_{xy}}{S^2_x}$$` `$$\hat\beta_{0}=\bar{y}-\hat\beta_{1}\bar{x}$$` ] -- Además teniendo en cuenta que el _coeficiente de correlación lineal_ es: `$$r=\frac{S_{xy}}{S_x S_y}$$` -- se tiene finalmente: .resalta[ `$$\hat\sigma^{2}_\varepsilon=\frac{n-1}{n}\left(S^2_y-\frac{S^2_{xy}}{S^2_x}\right)=\frac{n-1}{n}S^2_y\left(1-r^2\right)$$` ] --- ## Regresión Lineal: .blue[Estimación por Máxima Verosimilitud] Nótese que maximizar la verosimilitud: `$$\ell\left(\beta_{0},\beta_{1},\sigma_\varepsilon\right)=-n\log\left(\sigma_\varepsilon\right)-n\log\left(\sqrt{2\pi}\right)-\frac{1}{2\sigma_\varepsilon^{2}}\sum_{i=i}^{n}\left(y_{i}-\left(\beta_{0}+\beta_{1}x_{i}\right)\right)^{2}$$` -- es equivalente a minimizar: `$$D\left(\beta_{0},\beta_{1},\sigma_\varepsilon\right)=\sum_{i=i}^{n}\left(y_{i}-\left(\beta_{0}+\beta_{1}x_{i}\right)\right)^{2}$$` -- Por tanto: .resalta[ Si `\(\varepsilon \approx N(0,\sigma_\varepsilon)\)` los estimadores MV de `\(\beta_0\)` y `\(\beta_1\)` son los que minimizan la suma de cuadrados de las distancias de los puntos a la recta; en definitiva, producen una recta que pasa por el _centro_ de la nube de puntos. ] --- ## .blue[Ejemplo] Volvemos a los datos de los salmones que vimos al principio. En este caso `\(x\)`= _Talla_ e `\(y\)`= _Peso_. Queremos, por tanto, ajustar la recta: `$$Peso = \beta_0 + \beta_1 \cdot Talla$$` -- Utilizando R obtenemos: ``` ## mean(talla) mean(peso) var(talla) var(peso) ## 83.520 12.258 285.548 17.107 ``` ``` ## cov(talla,peso) ## 62.792 ``` -- `$$\hat\beta_1=\frac{S_{xy}}{S^2_x}=\frac{62.792}{285.548}=0.22$$` -- `$$\hat\beta_0=\bar{y}-\hat{\beta_1}\bar{x}=12.258 - 0.22 \cdot 83.52=-6.108$$` -- Por tanto la recta es: `$$Peso = -6.108 + 0.22 \cdot Talla$$` --- ## .blue[Ejemplo:] Regresión lineal con R R dispone de la función `lm` para ajustar la recta de regresión: ```r recta <- lm(peso~talla,data=salmones) recta ``` ``` ## ## Call: ## lm(formula = peso ~ talla, data = salmones) ## ## Coefficients: ## (Intercept) talla ## -6.1082 0.2199 ``` --- ## .blue[Ejemplo:] Regresión lineal con R La varianza del error es: `$$\hat\sigma^{2}_\varepsilon=\frac{n-1}{n}\left(S^2_y-\frac{S^2_{xy}}{S^2_x}\right)=\frac{99}{100}\left(17.107-\frac{62.792^2}{285.548}\right)= 3.333$$` -- <br> Con R puede calcularse mediante: ```r summary(recta)$sigma^2 ``` ``` ## [1] 3.332849 ``` --- ## Interpretación de los coeficientes de la regresión: .resalta[ `$$y = \beta_0 + \beta_1 x + \varepsilon, \quad \varepsilon\approx N(0,\sigma_\varepsilon)$$` ] -- * .blue[ __Pendiente__] ( `\(\beta_1\)`): Representa el cambio que se produce en `\(y\)` por cada unidad de incremento en el valor la variable `\(x\)`. -- * .blue[ __Ordenada__] ( `\(\beta_0\)`): Representa el valor esperado de `\(Y\)` cuando la `\(x=0\)`. Sólo tiene sentido interpretar así este coeficiente cuando los puntos observados realmente pasan por `\(x=0\)`. En caso contrario `\(\beta_0\)` debe entenderse como un simple coeficiente de ajuste sin mayor interpretación. -- * .blue[ __Predicción__] ( `\(\beta_0 +\beta_1 x_i\)`): representa el valor esperado de `\(Y\)` cuando la `\(X\)` vale `\(x_i\)`. -- * .blue[ __Desviación típica residual__] ( `\(\sigma_\varepsilon\)`): representa la variabilidad de `\(Y\)` en torno a su valor esperado `\(\beta_0 +\beta_1 x_i\)` cuando `\(X=x_i\)`. Se asume que es constante a lo largo de todo el recorrido de la recta. --- ## .blue[Ejemplo:] Interpretación de los coeficientes. En el caso de los salmones: `$$Peso = -6.108 + 0.22 \cdot Talla$$` .pull-left[ 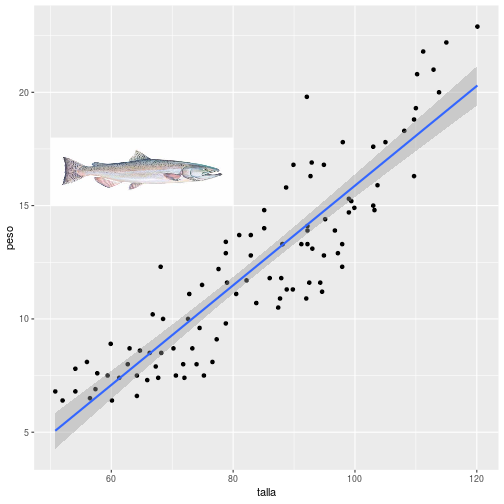<!-- --> ] -- .pull-right[ * `\(\hat\beta_1=0.22\)`: <font size = "3"> significa que por cada centímetro que se incrementa la longitud de un salmón, su peso esperado se incrementa en 0.22 kg. </font> * `\(\hat\beta_0=-6.108\)`: <font size = "3"> si quisiéramos interpretarlo como el valor de `\(Y\)` cuando `\(x=0\)`, significaría que un salmón de 0 cm de longitud pesaría `\(-6.108\)` kg; dado que no se han observado salmones de 0 cm de longitud (ni siquiera existen), el valor `\(\hat\beta_0\)` solo puede interpetarse como un coeficiente de ajuste, necesario para que la recta pase por la nube de puntos. </font> ] --- ## .blue[Ejemplo:] Interpretación de los coeficientes. .resalta[ .red[ __Nunca__] debe utilizarse una recta de regresión para extrapolar ya que, en general, no podemos estar seguros de que la relación entre `\(X\)` e `\(Y\)` sea la misma fuera del rango observado. ] -- .pull-left[ 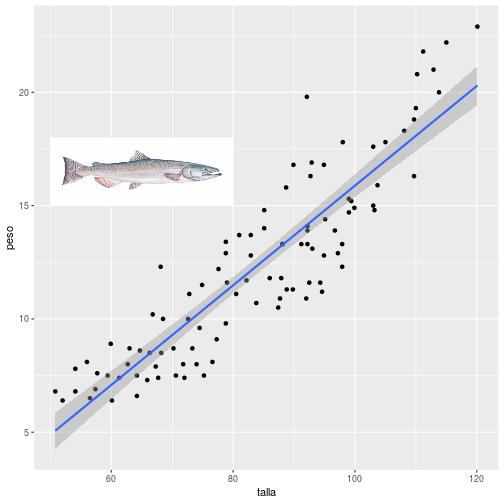<!-- --> ] -- .pull-right[ * Para `\(talla = 80\; cm\)`, la recta predice un peso esperado de: `$$peso= -6.108 + 0.22 \cdot 80= 6.484\; kg$$` * Para `\(talla = 100\; cm\)`, la recta predice un peso esperado de: `$$peso= -6.108 + 0.22 \cdot 100= 10.882\; kg$$` * Para `\(talla = 200\; cm\)`: <font size = "3" color="red" >no deben hacerse predicciones, pues no se han observado salmones en ese rango de talla</font> ] --- ## Interpretación de los coeficientes: no extrapolar Los riesgos de extrapolar: 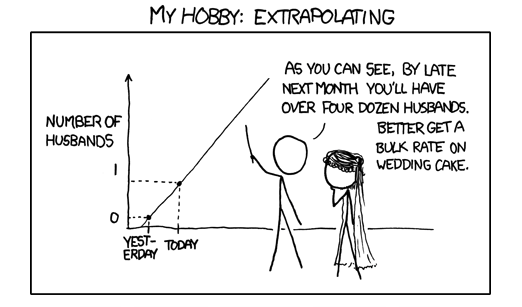 [Enlace a la fuente original de la viñeta](https://xkcd.com/605/) --- ## Predicción usando la recta de regresión con R Para realizar predicciones de la recta de regresión utilizando R se utiliza la función `predict`. -- ### .blue[Ejemplo:] Para prededir, con la recta anterior, el peso esperado de salmones para tallas de 80 y 100 cm: ```r predict(recta,newdata=data.frame(talla=c(80,100))) ``` ``` ## 1 2 ## 11.48395 15.88197 ``` --- ## Inferencia en regresión lineal * Cuando ajustamos una recta a una nube de puntos observados obtenemos unos valores estimados `\(\hat\beta_0\)`, `\(\hat\beta_1\)` y `\(\hat\sigma^2_\varepsilon\)` -- * Si de la misma población observamos una nueva muestra de puntos, obtendremos otros valores de `\(\hat{\beta_0}\)`, `\(\hat{\beta_1}\)` y `\(\hat\sigma^2_\varepsilon\)` distintos (aunque seguramente parecidos) a los anteriores. -- .resalta[ * ¿Cuánto se aproximan estos valores estimados a los verdaderos valores de `\(\beta_0\)`, `\(\beta_1\)` y `\(\sigma^2_\varepsilon\)` en la población? ] -- .resalta[ * Cuando se realiza una predicción utilizando los valores estimados `\(\hat\beta_0\)` y `\(\hat\beta_1\)`, ¿qué margen de error cabe esperar en esta predicción? ] --- ## Inferencia en regresión lineal simple Si las observaciones `\(\left\{(x_i,y_i)\; i=1,\dots, n\right\}\)` se ajustan al modelo `\(y_i = \beta_0 + \beta_1 x_i + \varepsilon_i\)`, siendo los `\(\varepsilon_i\approx N(0,\sigma_\varepsilon)\)` e .red[ __independientes__], se puede demostrar que: `$$\frac{\beta_{1}-\hat{\beta}_{1}}{\hat{\sigma}_{\varepsilon}\sqrt{\frac{1}{S_{x}^{2}\left(n-1\right)}}}\approx t_{n-2}$$` -- `$$\frac{\beta_{0}-\hat{\beta}_{0}}{\hat{\sigma}_{\varepsilon}\sqrt{\frac{1}{n}+\frac{\overline{x}^{2}}{S_{x}^{2}\left(n-1\right)}}}\approx t_{n-2}$$` -- `$$\frac{\left(n-2\right)\hat{\sigma}_{\varepsilon}^{2}}{\sigma_{\varepsilon}^{2}}\approx\chi_{n-2}^{2}$$` --- ## Inferencia en regresión lineal simple: Intervalos de confianza De las distribuciones anteriores es fácil deducir los siguientes intervalos de confianza: * Pendiente: .resalta[ `$$\beta_{1}\in\left[{\hat{\beta}_{1}\pm t_{n-2,\alpha/2}\frac{{\hat{\sigma}_{\varepsilon}}}{{\sqrt{n-1}\,S_{x}}}}\right]$$` ] -- * Ordenada: .resalta[ `$$\beta_{0}\in\left[{\hat{\beta}_{0}\pm t_{n-2,\alpha/2}\hat{\sigma}_{\varepsilon}\sqrt{\frac{1}{n}+\frac{{\bar{x}^{2}}}{{\left({n-1}\right)S_{x}^{2}}}}}\right]$$` ] --- ## Inferencia en regresión lineal simple: Intervalos de confianza * Varianza residual `\(\sigma^2_\varepsilon\)` .resalta[ `$$\sigma_{\varepsilon}^{2}\in\left(\frac{\left(n-2\right)\sigma_{\varepsilon}^{2}}{\chi_{n-2,\alpha/2}^{2}},\frac{\left(n-2\right)\sigma_{\varepsilon}^{2}}{\chi_{n-2,1-\alpha/2}^{2}}\right)$$` ] --- ## Inferencia en regresión lineal simple: Intervalos de confianza * También puede probarse que un intervalo de confianza para la __predicción__ de los posibles valores de `\(y\)` cuando `\(X=x\)` es: .resalta[ `$$y\left(x\right)\in\left[{\hat{y}\left(x\right)\pm t_{n-2,\alpha/2}\hat{\sigma}_{\varepsilon}\sqrt{\,1+\frac{1}{n}+\frac{{(x-\bar{x})^{2}}}{{\left({n-1}\right)S_{X}^{2}}}}}\right]\qquad \hat{y}(x)=\hat\beta_0+\hat\beta_1 x$$` ] -- * Si se desea un intervalo de confianza para el __valor medio de todas las y que se pueden observar para un x fijo__, éste es de la forma: .resalta[ `$$\bar{y}\left(x\right)\in\left[{\hat{y}\left(x\right)\pm t_{n-2,\alpha/2}\hat{\sigma}_{\varepsilon}\sqrt{\,\frac{1}{n}+\frac{{(x-\bar{x})^{2}}}{{\left({n-1}\right)S_{X}^{2}}}}}\right]\qquad \hat{y}(x)=\hat\beta_0+\hat\beta_1 x$$` ] --- ## .blue[Ejemplo:] Intervalos de confianza para la regresión en R. En R es muy fácil obtener los intervalos de confianza. Una vez ajustada la recta mediante `lm`, los intervalos para los coeficientes (ordenada y pendiente) se obtienen mediante `confint`: -- ```r recta <- lm(peso~talla,data=salmones) recta ``` ``` ## ## Call: ## lm(formula = peso ~ talla, data = salmones) ## ## Coefficients: ## (Intercept) talla ## -6.1082 0.2199 ``` ```r confint(recta) ``` ``` ## 2.5 % 97.5 % ## (Intercept) -7.943903 -4.2724186 ## talla 0.198354 0.2414487 ``` --- ## .blue[Ejemplo:] Intervalos de confianza para la regresión en R. * Los intervalos para las predicciones individuales se obtienen mediante: ```r predict(recta, newdata=data.frame(talla=c(80,100)), interval="prediction") ``` ``` ## fit lwr upr ## 1 11.48395 7.842226 15.12567 ## 2 15.88197 12.223767 19.54018 ``` -- * Los intervalos para la predicción de valores medios se obtienen mediante: ```r predict(recta, newdata=data.frame(talla=c(80,100)), interval="confidence") ``` ``` ## fit lwr upr ## 1 11.48395 11.11381 11.85409 ## 2 15.88197 15.37468 16.38927 ``` -- .resalta[ Nótese que la predicción del valor medio tiene un intervalo más estrecho que la predicción de valores individuales; es lógico que sea así, pues el valor medio es siempre menos variable que los valores individuales. ] --- ## Contrastes sobre la pendiente de la regresión 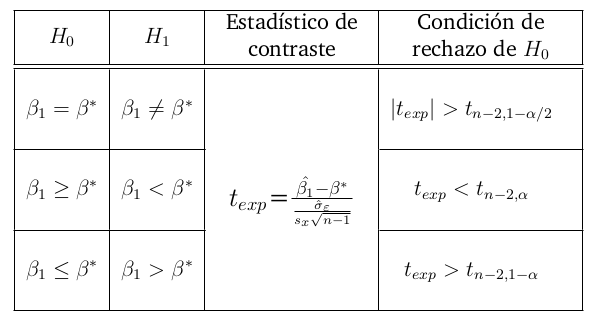 El caso particular de `\(\beta^*=0\)`, es decir, el contraste donde `\(H_0:\beta_1=0\)` se denomina __contraste de la regresión__. Si se acepta la hipótesis nula, ello significa que la recta es horizontal y que la variable `\(x\)` no predice (al menos de forma lineal) los valores de `\(y\)` --- ## Contrastes sobre la pendiente de la regresión El p-valor del contraste de la regresión es el correspondiente a la talla: ``` ## ## Call: ## lm(formula = peso ~ talla, data = salmones) ## ## Residuals: ## Min 1Q Median 3Q Max ## -3.4945 -1.4121 -0.0193 1.2252 5.6552 ## ## Coefficients: ## Estimate Std. Error t value Pr(>|t|) ## (Intercept) -6.10816 0.92506 -6.603 2.09e-09 *** ## talla 0.21990 0.01086 20.252 < 2e-16 *** ## --- ## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 ## ## Residual standard error: 1.826 on 98 degrees of freedom ## Multiple R-squared: 0.8071, Adjusted R-squared: 0.8052 ## F-statistic: 410.2 on 1 and 98 DF, p-value: < 2.2e-16 ``` Como p-valor< `\(\alpha\)` (en este caso da igual qué `\(\alpha\)` pongamos ya que el p-valor vale 0), se puede rechazar la hipótesis nula, y por tanto tenemos evidencia suficiente de que `\(\beta_1>0\)` --- ## Validación del modelo de regresión Toda la inferencia del modelo de regresión se apoya en la hipótesis de que los residuos de la regresión son normales, independientes y con varianza constante (homoscedasticidad) a lo largo de la recta. -- * __Validación gráfica de la independencia y la homoscedasticidad:__ el gráfico de los residuos frente a la variable `\(x\)` no debe presentar tendencias, irregularidades en la varianza a lo largo de su recorrido, ni valores anómalos; los residuos además deben estar centrados en el cero. -- * __Validación de la normalidad:__ Test de Shapiro-Wilk sobre los residuos y gráfico qqPlot (de la librería `car`) para identificar posibles outliers --- ## Validación del modelo de regresión * Residuos frente a la variable `\(x\)` (en este caso la talla) <img src="01-Correlacion_y_regresion_lineal_simple_files/figure-html/unnamed-chunk-14-1.png" style="display: block; margin: auto;" /> --- ## Validación del modelo de regresión * qqPlot de los residuos y test de Shapiro-Wilk: <img src="01-Correlacion_y_regresion_lineal_simple_files/figure-html/unnamed-chunk-15-1.png" style="display: block; margin: auto;" /> ``` ## ## Shapiro-Wilk normality test ## ## data: residuals(recta) ## W = 0.98308, p-value = 0.229 ``` --- ## Coeficiente de correlación El coeficiente de correlación lineal de Pearson es: `$$\rho=\frac{Cov(X,Y)}{\sigma_X\cdot\sigma_Y}$$` -- Se estima a partir de la muestra mediante: `$$r=\frac{S_{xy}}{S_x\cdot S_y}$$` -- * Valores próximos a 1 (o a -1) indican _habitualmente_ un buen ajuste a una recta de pendiente positiva (o negativa, respectivamente) -- * Valores próximos a cero indican que la nube de puntos no se parece a una recta (aunque podría adoptar alguna otra forma geométrica, lo que indicaría algún otro tipo de asociación entre `\(x\)` e `\(y\)`). --- ## Coeficiente de correlación .resalta[ ¡¡ Siempre conviene representar gráficamente la nube de puntos. !! ] -- .pull-left[ <!-- --> ] -- .pull-right[ <br> .red[ Cuarteto de Anscombe:] En los cuatro casos `\(r=0.82\)`; sin embargo las nubes de puntos son completamente diferentes y en los casos II, III y IV se apartan notablemente de la linealidad. .red[ Datasaurus dozen:] Otro ejemplo de nubes de puntos completamente diferentes y con la misma correlación [aquí](https://cran.r-project.org/web/packages/datasauRus/vignettes/Datasaurus.html) ] --- ## Intervalo de confianza para el coeficiente de correlación Si definimos: `$$z_{r}=\frac{1}{2}\ln\left(\frac{1+r}{1-r}\right)$$` -- se puede probar (Fisher) que: `$$z_{r}\approx N\left(\frac{1}{2}\ln\left(\frac{1+\rho}{1-\rho}\right),\frac{1}{\sqrt{n-3}}\right)$$` -- Por tanto: `$$P\left(\left|z_{r}-\frac{1}{2}\ln\left(\frac{1+\rho}{1-\rho}\right)\right|\le z_{\alpha/2}\frac{1}{\sqrt{n-3}}\right)=1-\alpha$$` -- de donde: `$$\frac{1}{2}\ln\left(\frac{1+\rho}{1-\rho}\right)\in\left[z_{r}\pm z_{\alpha/2}\frac{1}{\sqrt{n-3}}\right]$$` --- ## Intervalo de confianza para el coeficiente de correlación A partir de la expresión anterior, llamando: `$$z_{inf}=z_{r}-z_{\alpha/2}\frac{1}{\sqrt{n-3}},\,\,\,z_{sup}=z_{r}+z_{\alpha/2}\frac{1}{\sqrt{n-3}}$$` -- se obtiene el siguiente intervalo de confianza a nivel `\(1-\alpha\)` para `\(\rho\)`: .resalta[ `$$\rho\in\left[\frac{e^{2z_{inf}}-1}{e^{2z_{inf}}+1},\frac{e^{2z_{sup}}-1}{e^{2z_{sup}}+1}\right]$$` ] --- ## .blue[Ejemplo:] Coeficiente de correlación en R * Cálculo del coeficiente de correlación entre la talla y el peso de los salmones: ```r with(salmones,cor(talla,peso)) ``` ``` ## [1] 0.898414 ``` -- <br> * Intervalo de confianza: ```r with(salmones,cor.test(talla,peso)$conf.int) ``` ``` ## [1] 0.8524176 0.9306119 ## attr(,"conf.level") ## [1] 0.95 ``` --- ## Correlación y causalidad .resalta[ Que dos variables `\(X\)` e `\(Y\)` tengan una fuerte asociación lineal (valor alto de correlación, próximo a 1 ó a -1) .blue[ __no implica__] que `\(X\)` sea la .blue[ __causa__] de `\(Y\)`, ni que `\(Y\)` sea la .blue[ __causa__] de `\(X\)`. ] -- ## .blue[Ejemplo:] Se ha observado que cuando aumenta la venta de helados, aumenta de forma prácticamente lineal el número de personas que son atacadas por tiburones: ¿significa eso que podemos disminuir el número de ataques de tiburón simplemente vendiendo menos helados? --- ## Correlación y causalidad  --- ## Correlación y causalidad Obviamente la respuesta es no; la venta de helados no es la causa de los ataques de los tiburones (ni al revés), por muy fuerte que sea la asociación entre estas dos variables. -- <br> En este caso es una tercera variable (.red[factor de confusión]), la temperatura en este caso, la que dispara simultáneamente la compra de helados y el número de bañistas en la playa (que tiene como efecto colateral que hay más posibilidades de que alguien sea atacado por un tiburón) --- ## Correlación y causalidad * Se pueden encontrar muchos más ejemplos de correlaciones espurias (relación entre variables sin conexión lógica entre ellas) en la web [tylervigen](http://www.tylervigen.com/spurious-correlations) -- <br> .resalta[ Para establecer que la asociación entre dos variables es real y no espuria (inducida por un tercer factor oculto, o factor de confusión) es preciso .blue[ __encontrar un mecanismo plausible__] que explique la causalidad, y .blue[ __ponerlo a prueba__] mediante la realización de experimentos adecuadamente diseñados para descartar la intervención de factores ocultos. ] --- ## Coeficiente de determinación `\(R^2\)` Sean `\(\hat{y}_{i}=\hat{\beta}_{0}+\hat{\beta}_{1}x_{i}\)` los valores predichos por la recta de regresión ajustada a una nube de puntos `\(\left\{ \left(x_{i},y_{i}\right),i=1,\dots.n\right\}\)`. Es fácil comprobar que se cumple la siguiente igualdad: .resalta[ `$$\sum_{i=1}^{n}\left(y_{i}-\overline{y}\right)^{2}=\sum_{i=1}^{n}\left(\hat{y}_{i}-\overline{y}\right)^{2}+\sum_{i=1}^{n}\left(y_{i}-\hat{y}_{i}\right)^{2}$$` ] -- Los términos de esta igualdad reciben los nombres siguientes: __VARIABILIDAD TOTAL:__ `\(V_T=\sum_{i=1}^{n}\left(y_{i}-\overline{y}\right)^{2}\)` __VARIABILIDAD EXPLICADA:__ `\(V_E = \sum_{i=1}^{n}\left(\hat{y}_{i}-\overline{y}\right)^{2}\)` __VARIABILIDAD RESIDUAL:__ `\(V_R = \sum_{i=1}^{n}\left(y_{i}-\hat{y}_{i}\right)^{2}\)` -- `$$V_T=V_E+V_R$$` --- ## Coeficiente de determinación `\(R^2\)` Entonces: * Si las predicciones `\(\hat{y}_i\)` coinciden con los valores observados `\(y_i\)`, la variabilidad residual es cero: `\(V_R = \sum_{i=1}^{n}\left(y_{i}-\hat{y}_{i}\right)^{2}=0\)` (la recta .red[ _"explica"_] completamente la variabilidad en la variable respuesta `\(y\)`). * Si los valores de `\(x\)` no están asociados con `\(y\)`, la recta es horizontal (predice el mismo valor de `\(y\)` cualquiera que sea el valor de `\(x\)`); la pendiente es cero y en tal caso las predicciones son siempre `\(\hat{y}_i=\bar{y}\)`; la variabilidad explicada en tal caso es cero: `\(V_E = \sum_{i=1}^{n}\left(\hat{y}_{i}-\overline{y}\right)^{2}=0\)` * Se denomina .blue[ __Coeficiente de determinación__] al cociente: .resalta[ `$$R^2=\frac{V_E}{V_T}=\frac{\sum_{i=1}^{n}\left(\hat{y}_{i}-\overline{y}\right)^{2}}{\sum_{i=1}^{n}\left(y_{i}-\overline{y}\right)^{2}}$$` ] --- ## Coeficiente de determinación `\(R^2\)` `$$R^2=\frac{V_E}{V_T}=\frac{\sum_{i=1}^{n}\left(\hat{y}_{i}-\overline{y}\right)^{2}}{\sum_{i=1}^{n}\left(y_{i}-\overline{y}\right)^{2}}$$` .resalta[ Este coeficiente mide la proporción de la variabilidad en la variable respuesta `\(Y\)` que es explicada (o está determinada) por la variable explicativa `\(X\)`. ] -- <br> .resalta[ Cuanto más se aproxime su valor a 1, mejor es el modelo; cuanto más se aproxime a cero tanto peor. ] -- * Se puede probar que `\(R^2=r^2\)` -- * `\(R^2\)` se suele expresar en porcentaje.